서 론
광대버섯속(Genus Amanita)은 계통학적으로 담자균문(Basidiomycota) 주름버섯목(Agricales) 광대버섯과(Amanitaceae)에 속하며, 세계적으로 500여 종이 보고되어 있다[1-3]. 또한, 전통적인 분류방식인 포자의 요오드 반응 유무에 의해 두 아속(Subgenus) Amanita와 Lepidella로 나뉜다[4,5]. 광대버섯은 여름에서 가을 사이에 침염수림, 활엽수림 또는 혼합림 내 지상에서 산생, 단생 또는 소수 군생하는 외생균근성 균류(ectomycorrhizal fungi)로 산림 생태계에 매우 중요한 역할을 하고 있으며, 식용버섯과 독버섯이 공존한다[6]. 독광대버섯군 (lethal amanitas)은 Lepidella 아속의 Phalloideae에 속하는 분류군으로 간독성 독소인 amatoxin을 생산할 수 있어 유럽에서는 지난 한세기 동안 때때로 죽음을 불러오는 치명적인 독버섯 중독사고의 원인으로 알려져 있다[7,8]. Amanita virosa는 1866년 Bertillon에 의해 처음 보고된 독광대버섯군에 속하는 대표적인 독버섯이다. 이 종은 1943년 Takagi에 의해 국내 서식종으로 처음 보고되었고, 이후 1975년 한국산 고등균류의 우리나라 이름에서 한국명 ‘독우산버섯’으로 기록하였다가, 1978년 한국말 버섯이름 통일안을 통해 ‘독우산광대버섯’이란 한국명을 사용하게 되었다[9]. A. virosa는 동아시아 지역에서도 독버섯 중독 사망사고를 자주 일으킨다. 하지만, 기존 연구[8]에 의하면 A. virosa는 A. subjuquilla var. alba, Amanita oberwinklerana와 형태적으로 매우 유사하여 오동정의 가능성이 있어 A. virosa에 의한 중독사고인지 의심해 봐야 한다고 보고하였다. 같은 연구에서 신종으로 보고된 Amanita pallidorosea와 Amanita rimosa 역시 형태적으로 매우 유사하여 오동정의 가능성이 높아 국내에서 수집되어 A. virosa로 동정된 표본들의 정확한 재동정이 요구되었다.
본 연구에서는 국내에 수집되었으나, 명확한 실험적 근거 없이 A. virosa로 동정된 표본을 대상으로, 형태학적 특징과 internal transcribed spacer (ITS) rDNA 유전자의 염기서열을 기초로 한 분자계통학적 분석을 통해 A. virosa와 그 근연종을 재동정하였다. 더불어 각각의 종의 특징을 기술하고 계통학적 위치를 확인하였다.
재료 및 방법
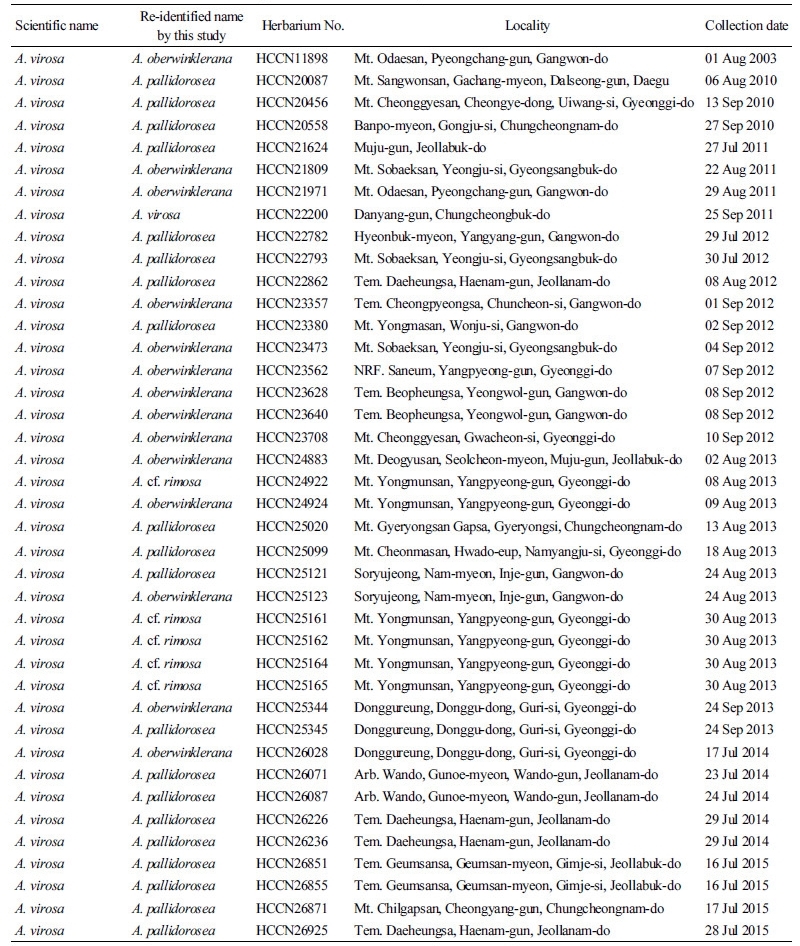
Amanita virosa의 정확한 종 동정을 위해 2000년부터 2015년까지 수집되어 국립농업과학원 표본실(Herbarium Conservation Center of NAS, HCCN)에 보관되어 있는 광대버섯 표본 중 형태학적으로 A. virosa로 동정된 40개 표본을 사용하였다(Table 1). 채집한 버섯은 현장에서 사진을 촬영하였고, 자실체의 발생 양상 및 서식지 환경에 대해 기록하였으며, 형태적인 특성은 갓, 대, 대기부 및 주름살의 색깔, 크기, 표면 상태 등의 육안적 특징 및 5% KOH용액을 이용하여 갓 상단의 화학적 색 변화를 조사, 기록하였다[10]. 미세구조관찰은 광학현미경 (Axioplan 2; Carl Zeiss, Oberkochen, Germany)을 이용하여 1% Congo red 용액으로 세포를 염색한 후 포자, 담자기, 시스티디아 등 미세구조를 관찰하고 상세하게 기록하였다[8]. 또한, 포자는 Melzer 용액을 이용하여 요오드 반응을 확인하였다. 관찰된 미세구조의 특성은 사진을 찍어 종 특성 자료로 첨부하고, 삽화를 본 논문에 기재하였다. 모든 표본은 학술적 자료로 영구 보존하기 위해 약 40°C, 6~12 시간 열풍 건조하여 상대습도 40±2%, 18~20°C의 HCCN에 보존하였다.
|
Table 1. Information on the 40 Amanita specimens used in this study |
|
|
|
HCCN, Herbarium Conservation Center of NAS; NRF, Natural Recreation Forest; Arb., Arboretum; Temple. |
분자유전학적 종동정과 계통학적 위치를 확인하기 위해 건조 표본과 생버섯에서 cetyl trimethylammonium bromide (CTAB) 추출법을 이용하여 DNA를 추출하였고[11], ITS1와 ITS4 primer를 사용하여 ITS 영역을 증폭하였다[12]. PCR 반응조건은 pre-denaturation 94°C, 5 min후, denaturation 94°C, 1 min, annealing 56°C, 1 min, extension 72°C, 1 min로 하여 총 35 cycles, final extension 72°C, 10 min 순으로 진행하였다. PCR 산물은 purification kit (AccuPrep PCR & Gel Extraction Kit; Bioneer, Daejeon, Korea)로 정제한 후 Macrogen (Seoul, Korea)사에 의뢰하여 분석하였다. 분석된 염기서열은 National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov, USA)의 BLAST program을 사용하여 GenBank에 등록되어 있는 염기서열과 비교하였다.
염기서열은 ClustalX 1.83 program을 사용하여 다중염기서열정렬 하였고[13], BioEdit v7.0.5 program[14]을 사용하여 수정하였다. 계통수 분석을 위해 maximum likelihood (ML) analysis은 MEGA 6.0 program[15]을 사용하였고, Bayesian inference (BI) 분석은 MrBayes v3.1.2 program[16]을 이용하여 수행하였다. ML tree는 1,000번의 bootstrapping 분석을 수행하였고, 60% 이상의 bootstrap 값만 계통수에 표시하였다. BI는 metropolis-coupled Markov- chain Monte Carlo (MCMC) 방법을 사용하여 1.5×106 generation 진행하는 동안 split frequency의 표준편차가 0.01 이하가 되었을 때 얻어진 계통수를 사용하였다. BI 값은 0.9 이상의 값만을 선택하였다.
결과 및 고찰
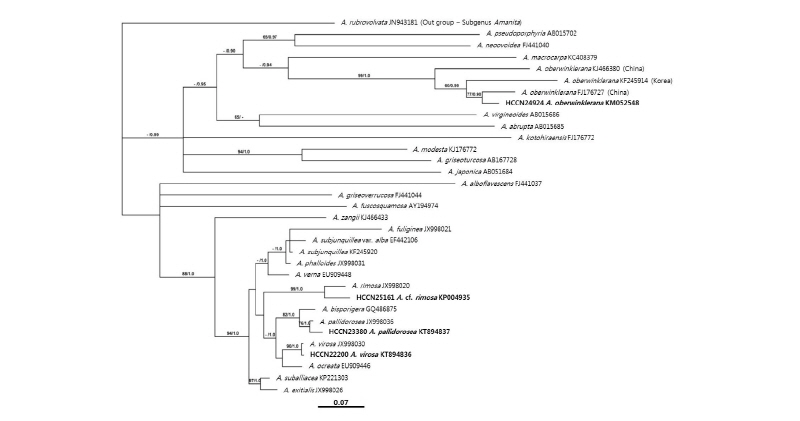
A. virosa로 동정된 40개의 표본은 모두 Phalloideae 절(Section)의 4종의 분류군(A. virosa 1개 표본, A. pallidorosea 20개 표본, A.cf. rimosa 5개 표본, A. oberwinklerana 14개 표본)으로 재동정되었다(Table 2). 계통학적 위치를 확인하기 위한 계통수는 각 그룹을 대표하는 하나의 염기서열만을 사용하여 분석하였으며, maximum likelihood bootstraps (LB), Bayesian posterior probabilities (PP) 값을 함께 나타내었다(Fig. 1). HCCN22200 (accession no. KT894836)은 Yang et al.의 A. virosa HKAS56694의 ITS 염기서열과 99% 상동성을 나타내었고, HCCN23380 (accession no. KT894837) 등 20개 표본은 A. pallidorosea HKAS75784와 99% 상동성을 나타내었으며, 두 종 모두 높은 bootstrap 수치 (LB > 70 and PP > 0.9)를 보이며 각각의 그룹을 형성하였다. HCCN25161 (accession no. KP004935) 등 5개 표본은 높은 bootstrap 값(LB = 99, PP = 1.0)을 가지고 A. rimosa와 하나의 그룹을 형성하였지만, HKAS75779 표본의 ITS 염기서열과 94% 상동성을 나타내어 A. rimosa와 차이를 보였다. HCCN24924 (accession no. KM052548) 등 14개 표본은 Zhang 등[8]의 A. oberwinklerana MHHNU6826 표본의 ITS 염기서열과 97%, Cai et al.의 HKAS 77330 표본의 ITS sequence와 89%, Kim et al.의 KA12-0898과 93%의 상동성을 나타내었다. 본 연구에서 분석된 A. oberwinklerana의 ITS 염기서열은 중국에서 연구된 두 표본 그리고 한국의 이전 연구에서 보고된 하나의 표본과 다소의 염기서열상의 차이를 보였지만, 하나의 그룹을 형성하였다.
A. virosa의 형태적 특성을 확인한 결과 갓의 직경은 약 29~123 mm로 백색이며, 편평반반구형이다(Fig. 2). 표면은 매끄럽고 갓 주변에 선이나 부착물은 없다. 대의 길이는 약 50~165 mm이며, 원통형이고 백색이다. 턱받이 아래쪽으로 미소섬유가 비늘처럼 되어있다. 턱받이는 대의 상부에 존재하고, 백색이며, 내린 치마모양의 막질로 되어있으나 부서지기 싶다. 대주머니는 백색의 막질로 되어있으며, 이차피실성(limbate) 형태로 대기부 상층은 떨어져 있다. 주름살은 촘촘하며, 백색이며 주름살 날은 분질 상이다. 포자는 8.2~11.3 × 6.7~9.7 µm로 대부분 구형에서 넓은 타원형이고 아밀로이드 성이다. 5% KOH용액에 노란색으로 반응한다.
A. pallidorosea의 경우 갓의 직경은 약 50~80 mm이고, 성숙하였을 때 중앙에 돌기가 존재한다(Fig. 3). 갓 중앙이 평활하고, 옅은 분홍색이며 갓 가장자리로 갈수록 백색으로 옅어진다. 하지만 옅은 분홍색이 항상 존재하는 것은 아니다. 버섯에 따라 순수한 백색도 존재한다. 대는 약 80~150 mm로 원통형에 가깝고, 위로 갈수로 약간 가늘어진다. 표면은 백색이며 미소섬유가 작은 비늘처럼 되어있다. 대주머니는 A. virosa와 형태적으로 같다. 턱받이는 대의 상부 근처에 막질로 위치하며, 내린 치마모양이고 질기다. 주름살은 떨어진 주름살이다. 포자는 (6.0) 6.5~8.0 (10.0) × 6.0~7.5 µm로 대부분 원형이며 아밀로이드 성이다. 5% KOH용액에 노란색으로 반응한다.
A. oberwinklerana의 형태적 특성을 확인한 결과 갓은 직경 30~60 mm 백색이고 때때로 갓 상부 중앙부는 크림색이다(Fig. 4). 갓의 가장자리는 매끄럽고 부착물이 없다. 대는 50~70 (~100) × 5~10 (~15) mm로 위로 갈수록 가늘어지며, 표면은 백색이고 손거스러미 모양의 비늘로 덮여있다. 대주머니는 이차피실성 대기부 상부 10 mm 위로 떨어진 이차피실성막을 가지고 있다. 턱받이는 흰색의 막질로 조직은 약하다. 포자는 8.4~9.2 (9.9) × 6.3~6.6 µm로 타원형 또는 넓은 타원형이며, 아밀로이드 성이다. 이 버섯은 5% KOH용액에 반응하지 않는 것이 특징으로 국내 보고종인 A. verna와 매우 유사하다.
A. cf. rimosa의 형태적 특성을 확인한 결과 갓의 직경은 45~60 mm의 편평반반구형이며, 백색이고, 갓 가장자리에 옅은 홈선이 존재한다(Fig. 5). 이 특성은 Zhang 등[8]의 기존 연구에서 발표된 A. rimosa와는 차이가 있다. 또한 본 연구에 사용된 표본에서는 A. rimosa와 같은 갓 전체의 균열이 발견되지 않았다. 대는 70~80 mm로 A. pallidorosea와 유사하게 원통형에 가깝고 위로 갈수록 가늘어진다. 또한 표면은 백색으로 미소섬유가 작은 비늘처럼 되어있다. 턱받이는 대의 상부에 백색의 막질 형태로 존재하며, 질기고, 방사상의 줄무늬가 있다. 포자는 7.2~10.1 (~10.2) × (6.3~) 6.6~8.3 (~8.5) µm로 대부분 원형에서 넓은 타원형이고 아밀로이드 성이다. 5% KOH 용액에 노란색으로 반응한다. 이와 같이 4종의 버섯들은 그 형태와 특성들이 매우 유사하지만, 각각의 종 특성들이 분명히 존재한다.
본 연구에서 사용된 40개의 표본은 4종의 그룹으로 나뉜다는 것을 확인하였다. A. virosa의 근연종 중 가장 많이 채집된 종은 20개 표본의 A. pallidorosea로 전국적으로 분포되어 있었으며, A. oberwinklerana는 14개 표본으로 강원, 경기 및 전북에서 대부분 발견되었다. A. cf. rimosa는 5개 표본으로 오직 경기도 양평 용문산에서만 발견되었으며, A. virosa는 하나의 표본만이 충북 단양에서 채집되었다. 따라서, 국내에 보고된 A. virosa에 의한 독버섯중독사고는 다른 3종이 원인이 된 중독사고일 가능성이 분명 존재한다. 물론 위 4종 모두 amatoxin을 가진 무서운 맹독버섯임은 분명하지만, 정확한 종동정에 의한 독버섯 중독의 원인을 규명하는 것 또한 중요한 일이라 하겠다. 또한 기존에 보고된 종들과 형태적, 계통학적으로 차이를 보인 A. cf. rimosa에 대한 지속적이고 추가적인 실험이 필요할 것으로 보인다. 또한, A. virosa와 가장 형태학적 특성이 유사한 것으로 국내에 보고된 A. verna (흰알광대버섯)는 형태학적 특성을 비교하였을 때 본 연구에서 확인된 A. oberwinklerana와 동일 종일 가능성이 존재하였다. 그러므로 국내에 기록된 A. verna의 기준 종과 표본에 대해 재동정이 필요할 것으로 여겨진다.
적 요
본 연구는 2000년부터 2015년까지 한국에서 수집되어 Amanita virosa로 동정된 40개 표본의 분자계통학적 종동정을 위해 internal transcribed spacer rDNA 영역의 염기서열을 사용하여 수행하였다. 이번 연구에서 Lepidella 아속의 Phalloideae절에 속하는 Amanita oberwinklerana의 14개 표본, A.cf. rimosa 5개 표본, A. pallidorosea 20개 표본 및 A. virosa 1개 표본을 확인하였고, 그들의 형태학적 특성을 재조사하였다. 백색 계열의 맹독성 버섯에 속하는 위 3종(A. oberwinklerana, A.cf. rimosa, A. pallidorosea)은 아직 국내에는 잘 알려지지 않았지만, 형태학적 특징들이 A. virosa와 매우 유사하여 존재하더라도 오동정되었을 가능성이 높다. 본 연구에 사용된 모든 표본은 국립농업과학원 표본실에 보관되어 있다.