서론
Aureobaisdium속은 세계적으로 다양한 환경에서 널리 분포하고 자낭균 Dothideomycetes강(class)에 속하는 흑효모이다[1]. 특히 Aureobasidium pullulans는 식품 첨가물로 사용되는 pullulan 생산뿐만 아니라 amylases, cellulases, lipases, proteases, xylanases, b-fructofuranosidases, maltosyltransferases, mannanases, laccases [2] 등 다양한 세포 외 가수분해 효소 활성 작용으로 제약 및 식품산업 등에 활용되고 있어 생물공학적으로 중요한 효모로 알려져 있다[3-4].
Aureobasidium속은 형태 및 영양생리학적 특성을 기준으로 분류되었을 경우 종 단위로는 크게 Aureobasidium pullulans, A. leucospermi, A. proteae로 나누어지고, A. pullulans는 다시 4가지 변종, var. pullulans, var. melanogenum, var. subglaciale, var. namibiae 으로 구분되었다[5]. 하지만 최근 Gostinčar 등[3]에서 A. pullulans의 주요 변종 4가지의 genome을 분석한 결과 4가지의 변종 사이의 염기서열이 종(species)으로 정의 될 만큼 유의한 차이가 확인됨으로써 4가지 변종은 A. pullulans, A. melanogenum, A. subglaciale, A. namibiae 으로 각각 승격 되었다[3]. 이처럼 Aureobasidium속은 변이로 인해 다양한 종 또는 변종이 존재하고[5], 아직 명확한 종간 동정 방법이 개발되지 않았기 때문에[6] 분자적 특성과 함께 형태적 특성도 함께 조사되어야 정확한 동정이 이루어질 수 있다.
국내에서 연구되는 효모는 고부가가치 의약 산업이나 식품 산업을 중심으로 연구되고 있기 때문에 주로 전통 발효 식품 위주로 분리되었으나[7-11], 발효된 원료에서는 제한된 효모 종(species)만 서식하여 다양한 효모가 분리되지 않았다. 최근에는 다양한 효모를 산업적으로 활용하기 위해 과일, 토양, 꽃 등 자연에 분포하는 야생 효모를 발굴하고 그 다양성을 확인하는 연구가 이루어지고 있다[12-15]. 본 연구에서는 호남지역 꽃에서 분리한 효모 중 우점종인 Aureobasidium의 분자 및 형태적 특성 분석을 통해 Aureobasidium속의 분포와 다양성에 대하여 알아보고 산업적으로 유용한 효모 자원을 구축 하고자 하였다.
재료 및 방법
호남지역 꽃 채집 및
호남지역 꽃 채집 및
호남지역 꽃 채집 및
2018년 전라북도 전주, 김제, 익산, 전라남도 구례, 광양, 순천, 여수, 해남 지역 일대의 꽃 54종을 채취한 후, chloramphenicol (17mg/L) (Sigma-Aldrich, St, Louis, MO, USA)과 propionic acid (1.25g/L) (Sigma-Aldrich)가 첨가된 5종 배지, malt extract agar (Oxoid, Hampshire, UK), dichloran-glycerol agar (Oxoid), dichloran rose bengal chloramphenicol agar (Oxoid), yeast extract-peptone-dextrose agar (MB cell, Seoul, Korea), glucose-peptone-yeast extract agar (MB cell, Seoul, Korea)에 희석평판 및 3차 계대배양을 통해 효모 균주를 순수분리 하였다.
Aureobasidium
Aureobasidium
다양하게 분리된 효모들을 동정하기 위하여 마크로젠(Macrogen, Seoul, Korea)에 염기서열 분석을 의뢰하였다. large-subunit (LSU) ribosomal DNA gene의 D1/D2 영역과 internal transcribed spacers (ITS) ribosomal DNA 영역을 NL1 (5’-GCATATCAATAAGCGGAGGAAAAG-3), NL4 (5’-GGTCCGTGTTTCAAGACGG-3′)와 ITS1 (5’-TCCGTAGGTGAACCTGCGG-3’), ITS4 (5’-TCCTCCGCTTATTGATATGC-3’) primer를 각각 사용하여 PCR 증폭 및 염기서열을 분석하였다[16]. 염기서열 분석 및 계통수 제작은 MEGA ver. 7.0 [17] software를 사용하였으며, maximum likelihood statistical method (ML)로 계통수를 작성하면서 Bootstrap method (n = 1,000)로 분석을 수행하였다. Aureobasidium속 계통수 분석을 위한 표준균주 염기서열 정보는 NCBI (https://www.ncbi.nlm.nih.gov) 데이터베이스로부터 확보하였고, 상동성 비교는 DNAMAN 7.0 (Informer Technologies, Los Angeles, CA, USA) software를 사용하였다.
Aureobasidium
Aureobasidium
염기서열 분석을 통해 Aureobasidium속으로 동정된 균주들의 형태적 특성을 비교하기 위해 콜로니 변화를 관찰하였다. 멸균수에 단일 콜로니를 각각 희석하여 potato dextrose agar (PDA; BD, Sparks, MD, USA)와 malt extract agar (MEA) 배지에 접종한 후, 암조건 하에 25℃에서 7~14일간 배양 후 변화를 관찰하였다.
결과 및 고찰
Aureobasidium
Aureobasidium
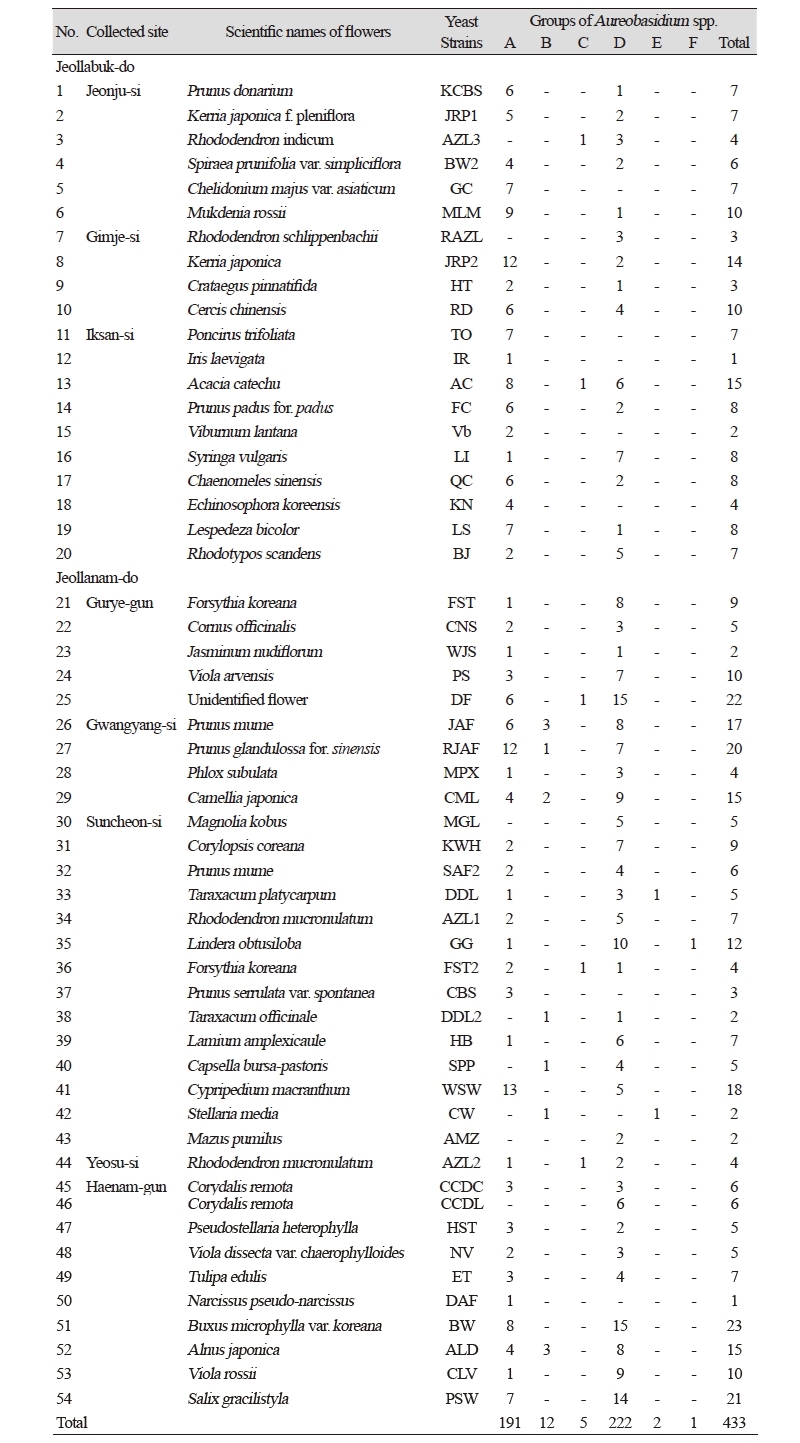
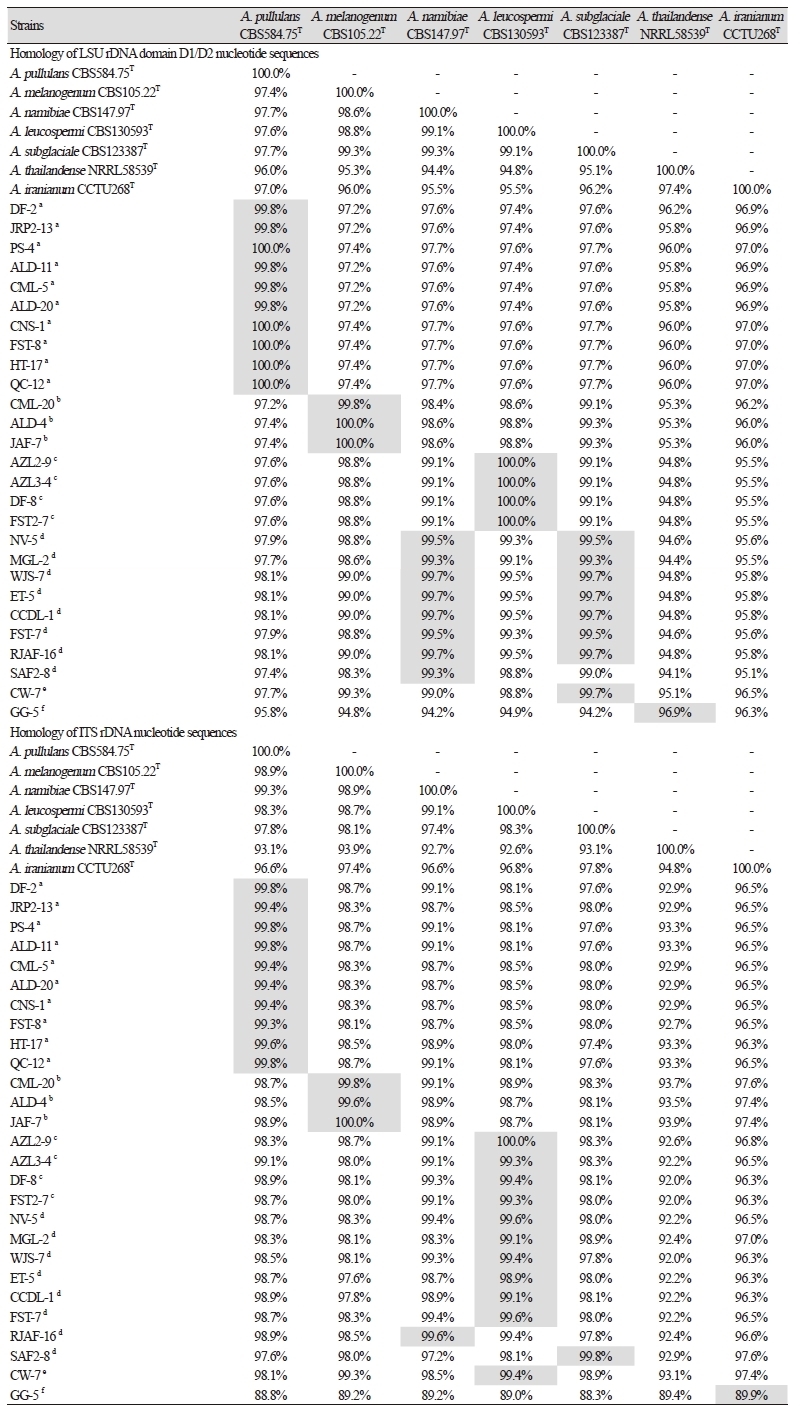
전라도 지역 일대의 54종 식물의 꽃으로부터 야생효모들을 분리하여 가장 우점하는 속(genus)인 Aureobasidium속 효모 433균주를 분리하였다(Table 1). Aureobasidium속의 종 수준 동정을 위하여 433균주 중 27개를 대표적으로 선발하였다. 대표 균주 선발은 LSU와 ITS 염기서열 상동성을 비교한 후 100% 일치하는 균주 중 하나씩 선발하여, 각기 다른 염기서열을 갖는 27균주가 최종 선택되었다. 대표 균주들의 LSU D1/D2 영역 염기서열을 이용한 계통수를 기준으로 전라도 지역의 Aureobasidium속 균주들은 6그룹(Groups A~F)으로 구성할 수 있었다(Table 1, Fig. 1).
전라도지역 꽃 전반에 걸쳐서 Aureobasidium속은 Group D (222균주)와 Group A (191균주)가 우점하는 경향을 보였고, Group C는 소수 종으로 존재하였으나 지역적으로 고르게 분포하였다. Group B (12균주), E (2균주), F (1균주)는 전라남도 지역에서만 분리된 것으로 보아 Aureobasidium은 전라북도 지역보다 전라남도 지역에서 더 다양하게 분포하는 것으로 추정된다(Table 1).
Aureobasidium
Aureobasidium
LSU와 ITS 계통수 및 상동성 비교에서 Group A는 LSU와 ITS 계통수에서 모두 A. pullulans와 그룹을 형성하고, LSU는 99.8%~100%와 ITS는 99.3~99.8%의 상동성으로 A. pullulans로 동정되었다. Group B도 LSU와 ITS 계통수에서 모두 A. melanogenum와 그룹을 형성하고, LSU는 99.8%~100%와 ITS는 99.6~100%의 상동성으로 A. melanogenum로 명확하게 동정되었다. Group C는 A. leucospermi와 비교하여 LSU는 100%, ITS는 99.3~100%의 상동성을 보이고, LSU 계통수에서도 A. leucospermi와 그룹을 형성하였으나 ITS 계통수에서는 A. leucospermi와 A. namibiae 사이에 위치하였다. Group D는 LSU 계통수 분석에서 A. namibiae로 그룹을 형성하고 ITS 계통수에서는 A. leucospermi, A. namibiae, A. subglaciale 사이에서 위치하였다. 또한 Group D의 LSU는 A. namibiae 또는 A. subglaciale과 99.3~99.7%의 상동성을 보이고, ITS는 A. leucospermi와 98.9~99.6%의 상동성을 보였다. Group E의 경우 LSU와 ITS 계통수 모두에서 A. subglaciale과 A. leucospermi의 공통 조상에서 분리된 종으로 보이며, LSU 분석에 의하면 A. subglaciale과 99.7%, ITS 분석의 경우 A. leucospermi와 99.4%로 A. subglaciale과 A. leucospermi 사이의 종으로 추정되었다. Group F는 Aureobasidium 신종으로 예상되며 추가 실험을 통해 신종 균주의 특성을 국내외에 보고하여야 할 것이다(Table 2, Fig. 1).
결론적으로 전라도 지역 꽃에서 분리한 Group A는 A. pullulans, Group B는 A. melanogenum, Group F는 Aureobasidium 신종 후보군으로 분류되었고, Group C, D, E는 A. leucospermi, A. namibiae, A. subglaciale 사이에 위치하지만 LSU와 ITS로는 정확한 종 수준 동정이 어렵기 때문에 추가적인 유전자 염기서열 부위 탐색을 위한 연구가 실시되어야 할 것이다. 또한 Lee 등[18]에 따르면 나무에서 분리된 Aureobasidium속은 A. melanogenum이 우점종이었으나, 본 논문에 의하면 호남지역 꽃에서는 A. pullulans와 A. namibiae 등이 우점종인 것으로 보아 Aureobasidium속은 지역적 분포 특성도 있지만, 서식지(origin)에 따라 종의 분포도 달라지는 것으로 판단된다.
Aureobasidium속은 배양 초기에 노란색, 크림색, 밝은 분홍색, 밝은 갈색을 띠는 콜로니에서 후기로 갈수록 멜라닌과 같은 어두운 색의 색소를 분비하면서 점차 거무스름한 콜로니로 변하는 특징이 있다[5]. Zalar 등[19]에 따르면 PDA나 MEA에서 A. pullulans는 배양 3주 이내, A. namibiae는 최소 1주 이내에는 분홍빛을 띤 콜로니가 유지되다가 점차 거무스름하게 변하고, A. melanogenum는 1주 이내에 검은색으로 변한다고 하였으나, 이 연구에서 사용된 균주들은 Group A (A. pullulans)의 경우 1주 이내에 이미 밝은 갈색에서 검은색을 띄기도 하였다. Group B (A. melanogenum)는 알려진 바와 같이 1주일 이내에 모두 밝은 갈색에서 검은색으로 변하여 형태적 특성이 분자적 특성과 일치하였고, LSU와 ITS 염기서열의 계통분석으로 구분이 어려웠던 Group C는 초기에 크림색에서 약간 밝은 갈색을 띄면서 Group D와 구분되는 특징을 보였다. Group D는 모두 1주일 이내에 밝은 갈색에서 검은색으로 변하였고, 일부 균주는 MEA에서 초기부터 검은색 색소를 분비하기도 하였다(Fig. 2). 이처럼 변종이 많은 Aureobasidium속은 단일 유전자 분석(LSU 또는 ITS)만으로는 종 동정이 어렵기 때문에 형태적 분석이나 생리적 특성 검정 등을 병행함으로써 다각적인 방법을 통한 동정이 요구된다.
이 연구에서는 호남지역 꽃에서 분리한 Aureobasidium속의 분포 및 다양성을 분자계통학적 및 형태적 특성을 통해 조사하였다. 이러한 Aureobasidium속에 대한 연구결과는 유용한 효모 선발과 미기록 효모 발굴 등을 위한 귀중한 자료로 활용 될 것으로 기대된다.
적요
다양한 Aureobaisidum속을 발굴하여 흑효모의 유용한 특성을 활용하기 위해 전라도 지역 꽃에서 효모 433균주를 분리하고 분자계통학 및 형태학적 분석을 통해 다양성을 확인하였다. large subunit (LSU) rDNA 염기서열 분석을 기준으로 전라도 지역의 Aureobasidium속을 6그룹으로 분류한 후, LSU와 ITS rDNA 염기서열 분석을 통해 전라도 지역 꽃에서 유래한 효모의 주요 우점종이 Group A와 Group D임을 확인할 수 있었다. Group B, E, F는 전라남도에만 분포하는 것으로 보아 전라북도에 비해 전라남도에서 다양한 Aureobasidium종이 분포하는 것으로 생각된다. Group A는 A. pullulans, Group B는 A. melanogenum, Group F는 Aureobasidium 신종 후보군으로 분류할 수 있었다. Group C, D, E는 LSU와 ITS rDNA 분석에서 A. leucospermi, A. namibiae, A. subglaciale 사이의 명확하게 분류되지 않았으나 콜로니 형태에서 구분 가능한 특성을 보인 것으로 보아, Aureobasidium은 분자계통학적 분석방법과 형태적 동정을 병행하여 종 수준 동정을 보완할 수 있을 것으로 생각된다.