땅콩(Arachis hypogaea L.)은 콩과식물로 땅콩속에 속하는 1년생 초본이며 국내 재배 규모가 확대되고 있는 경제적으로 중요한 작물 중 하나이다[1]. 국내 땅콩에 발생하는 병은 진균에 의한 병 13종, 세균에 의한 병 1종과 바이러스에 의한 5종 총 19종이 보고되어 있다[2]. 땅콩 잎에 발생하는 병으로는 Cercospora arachidicola에 의한 갈색무늬병, Cer. personata에 의한 검은무늬병, Phoma arachidicola에 의한 그물무늬병, Puccinia arachidis에 의한 녹병, Colletotrichum gloeosporioides에 의한 탄저병 등이 보고되어 있지만 C. sojae에 의한 탄저병은 보고된 바 없다[2,3]. 국외에서도 C. truncatum, C. chlorophyti, C. fructicola, C. gloeosporioides에 의한 땅콩 탄저병 발생이 보고되어 있지만 C. sojae에 의한 땅콩 탄저병은 보고된 바 없다[4-7].
Colletotrichum은 전 세계 식물병리학자들이 꼽은 가장 중요한 10대 식물병원진균 중 하나이고[8], 200종이 넘는 종복합체를 포함하는 아주 큰 속이다[10]. 그 중 C. sojae는 C. orchidearum species complex에 속한다[10].
2023년 충북 청주시 북이면 땅콩 포장에서 재배중인 땅콩 잎에 기존에 보고된 점무늬 병징과는 다른 반점을 발견하였고, 원인균을 구명하기 위해 이병시료로부터 균주를 분리 및 동정하였다. 병원성 검정을 통해 땅콩 병원균임을 확인하였으며 염기서열 및 유연관계 분석을 통해 C. sojae로 동정하였다.
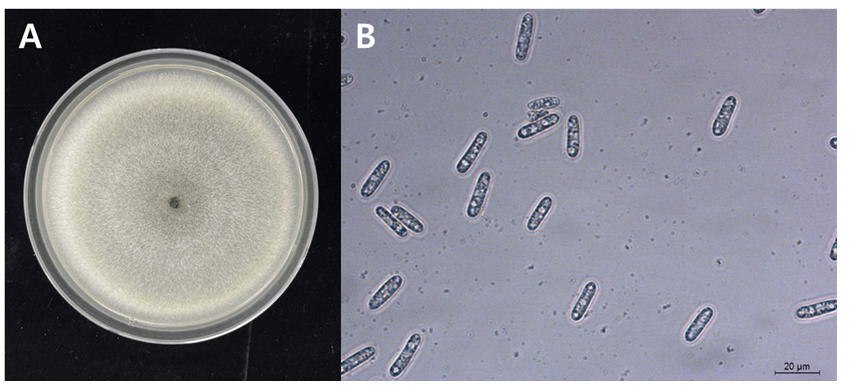
2023년 8월 청주시 청원구 북이면의 땅콩 재배 포장(36°45′56.9894” N, 127°34′36.3640” E)의 1% 미만 정도의 땅콩에서 잎 가장자리에 갈색 반점이 발생한 것을 발견하여 채집하였다(Fig. 1). 채집한 병든 잎에서 원인균을 분리하기 위해 반점이 형성된 부분과 건전한 부분의 경계 조직을 잘라 70% 에탄올에 30초간 담가 표면 살균한 후, 멸균수로 헹구어 내고 멸균된 여과지로 물기를 제거하였다. 표면 살균된 조직을 streptomycin 함유(100 μg/mL) potato dextrose agar (PDA) 배지에 치상하여 25℃에서 배양한 후 균사의 선단을 잘라 새로운 PDA 배지에 10일간 배양하였다. PDA 배지상에서 균총은 회백색을 띠었고(Fig. 2A), 주황색의 분생포자층을 형성하였다. 분생포자가 형성된 균총에서 포자를 취하여 단포자 분리를 하였다. 분생포자는 투명하고 격벽이 없으며 원통형이고 크기는 평균 18.28 μm (14.87-21.33 μm, n=50)였다(Fig. 2B). 순수 분리한 균주를 23PLD1으로 명명하고 streptomycin 함유 (100 μg/mL) 멸균수에 저장 및 보관하였고 동정 및 병원성 검정에 사용하였다. 23PLD1 균주는 농업유전자원정보센터에 기탁하였다(KACC 410619).
Fig. 2
Colletotrichum sojae. (A) fungal colony morphology of C. sojae on potato dextrose agar, (B) conidia of C. sojae.
분리한 C. sojae 23PLD1 균주의 병원성을 검정하기 위해 PDA 배지에 치상하여 25℃에서 14일간 배양 후, 형성된 분생포자를 수확하여 1×106 conidia/mL 밀도의 포자 현택액을 제조하였다. 지름 10 cm 포트에 땅콩 ʻ신팔광’ 종자를 파종하여 3주간 재배한 뒤 병원균 포자현탁액을 분무 접종하였고, 무처리구에는 멸균수를 같은 방법으로 접종하였다. 접종한 식물체는 상대습도 95% 이상, 온도 25℃ 조건에서 48시간동안 습실처리하여 병 발생을 유도하였다. 접종 5일 뒤 땅콩 잎 가장자리 부분에서 수침상 병징이 나타났으며(Fig. 3A), 접종 7일 뒤 반점 증상을 관찰하였다(Fig. 3B). 병징이 나타난 잎에서 병원균을 재분리하여 동정한 결과 접종 균주와 동일한 균임을 확인하였다.
Fig. 3
Symptoms of artificial inoculation test on peanut seedlings with an isolate. (A) water-soaking symptom, (B) leaf spot symptom.

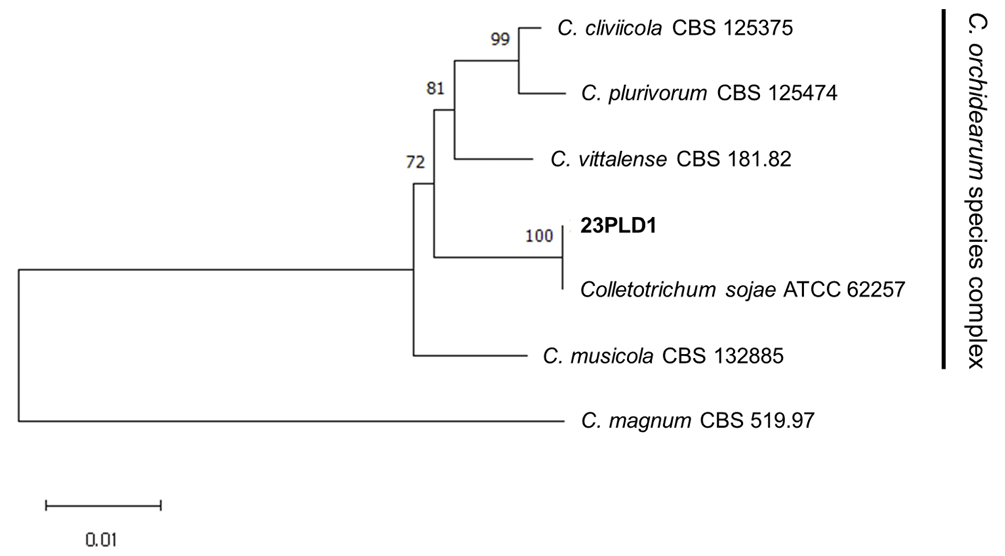
땅콩 잎에서 분리한 23PLD1 균주를 정밀 동정하기 위해 종 동정 관련 유전자들의 염기서열을 분석하였다. 셀로판지를 깐 PDA배지에 23PLD1 균주를 25℃에서 7일간 배양한 후, 균사만을 수집하여 마쇄한 후 DNeasy Plant Mini Kit (Qiagen, Hilden, Germany)를 이용하여 genomic DNA를 추출하였다. 추출한 DNA로 internal transcribed spacers (ITS), glyceraldehyde-3-phosphate dehydrogenase (GAPDH), chitin synthase-1 gene (CHS-1), actin gene (ACT), β-tubulin gene (TUB2) 영역을 증폭하였다. ITS 영역은 ITS5/4 [11], GAPDH 영역은 GDF/GDR [12], CHS-1 영역은 CHS-79F/CHS-354R [13], ACT 영역은 ACT-512F/ACT783R [14], TUB2 영역은 T1/Bt2b [14,15] 프라이머를 사용하였다. 각 영역의 증폭된 PCR 산물은 Macrogen (Seoul, Korea)에 염기서열 분석을 의뢰하였다. 각 유전자의 염기서열을 NCBI (National Center for Biotechnology Information, 미국국립생물정보센터)에서 BLAST 분석한 결과, GenBank에 등록되어 있는 C. sojae 균주의 유전자와 100% 상동성을 나타냄을 확인하였으며, 23PLD1 균주의 ITS 영역 염기서열을 NCBI의 GenBank database에 Acc no. OR946209로 등록하였다. 계통학적 분석을 위해 ACT (287 bp)-CHS-1 (300 bp)-GAPDH (331 bp)-ITS (557 bp)TUB2 (547 bp) 염기서열들을 연결한 후 MEGA11 프로그램으로 neighbor-joining method로 계통수를 작성하였다[16]. 계통수 분석 결과 23PLD1 균주는 C. orchidearum 종 복합체 clade에 속하였으며, C. sojae와 묶여 다른 종들과 뚜렷하게 구분되었다(Fig. 4).
Fig. 4
Phylogenetic tree constructed with the neighbor-joining method using combined multilocus gene (ITS, GAPDH, CHS-1, ACT and TUB2) from the peanut anthracnose pathogen 23PLD1 and those of other Colletotrichum spp. number on nodes are supporting percentages by 1,000 bootstrap replicates. C. magnum strain CBS 519.97 is used as outgroup.
적요
2023년 8월 청주 땅콩 재배포장에서 기보고되어 있는 점무늬 병징과는 다른 양상의 잎 반점을 발견하였다. 갈색 반점은 주로 잎 가장자리에 형성되었으며, 반점 부위에서 원인균을 분리하였다. 순수 분리한 병원균을 땅콩 유묘에 인공접종하여 병원성 검정을 수행하였다. 그 결과, 접종 5-7일 뒤에 유사한 병징을 확인할 수 있었고 병징 부위에서 동일한 균을 재분리하였다. 분리한 균주의 ACT-CHS-1-GAPDH-ITS-TUB2 영역을 이용하여 동정 및 유연관계 분석을 수행한 결과 C. sojae임을 확인하였다. 따라서 본 연구는 국내 최초로 C. sojae에 의해 발생한 땅콩 탄저병을 보고하고자 한다.



