서 론
클로탈라리아(Crotalaria juncea L.)는 brown hemp, sunn hemp, Indian hemp 혹은 Madras hemp로 불리며, 인도와 네팔, 파키스탄, 스리랑카, 남아프리카 등 열대 및 아열대 지역에서 섬유작물 혹은 풋거름작물로 재배되어 왔다[1]. 국내에서는 2005년부터 풋거름작물로 이용하기 위한 연구가 시작되었고, 특히 토양 내 뿌리혹선충 억제 효과가 뛰어난 것으로 알려지면서 오이, 참외, 배추 재배 농가에서 그 이용이 증가하고 있다[2]. 클로탈라리아에 발생하는 곰팡이병으로 인도에서는 Fusarium udum에 의한 시들음병과 Colletotrichum curvatum에 의한 탄저병의 피해가 심각하다고 하였으며, 그 외에 녹병, 균핵병 및 흰가루병 등을 언급한 바 있다[3]. 대만에서도 클로탈라리아 시들음병균으로 F. udum과 F. moniliforme 2종을 보고하면서 F. udum의 병원성이 더 강하다고 하였다[4]. 그밖에 여러 바이러스에 대한 클로탈라리아의 높은 감수성이 보고된 바 있다[5].
그러나 국내에는 클로탈라리아를 풋거름작물로 이용하기 때문에 이 작물에 발생하는 병에 대한 연구는 전무한 실정이다. 2014년 8월경, 클로탈라리아를 풋거름작물로 재배하는 시험포장에 발생한 시들음 증상으로부터 F. udum균을 분리하였다. Fusarium udum은 국내에 나무콩으로 알려진 pigeonpea에 심각한 병원균으로 알려져 있는데 특히 인도, 아프리카 등 열대 및 아열대 지역에서 그 피해가 크다[6, 7]. 풋거름작물 재배는 후작물에 비료 및 유기물을 공급하고, 토양 특성을 개선하거나 병해충 억제하는 등의 효과를 얻을 수 있으나, 경우에 따라 토양 서식 병원균의 기주 식물이 될 가능성이 존재한다. 그러므로 국내에서 풋거름작물로 활용되는 여러 작물을 대상으로 병 발생 모니터링과 병원균 동정, 기주범위 조사 등의 연구가 필요할 것으로 생각된다.
본 연구에서는 국내에서 처음으로 클로탈라리아 시들음병균을 분리하였고, 균학적 특성과 DNA 염기서열분석을 이용하여 동정하였으며, 클로탈라리아를 비롯한 국내 재배 콩 품종을 대상으로 병원성 검정을 수행한 결과를 보고하고자 한다.
재료 및 방법
병원균 분리 및 보존
2014년 8월, 클로탈라리아를 풋거름작물로 재배하는 전북 완주군 소재 시험포장에서 병든 시료를 채집하였다. 채집한 클로탈라리아 줄기에 형성된 자낭각을 수거하여 멸균수에 넣고, 자낭각을 파쇄하여 자낭포자를 현탁시키고, 물한천배지(water agar, WA)를 이용하여 단포자 분리하였다. 순수 분리된 균주는 감자한천배지(potato dextrose agar, PDA) 사면배지에 이식하여 배양하였고, 10°C에 보존하면서 실험에 사용하였다.
균학적 특성 조사
채집한 클로탈라리아 줄기에 형성된 자낭각을 직접 현미경으로 관찰하면서 자낭포자의 크기와 모양을 조사하였다. 또한 Fusarium균의 균학적 특성은 단포자 분리한 후 PDA 배지와 carnation leaf agar (CLA) 배지에 배양하여 조사하였다. 대형 분생포자의 모양과 크기, 소형 분생포자의 형성 유무, 모양 및 크기, 후막포자의 형성 유무, 경자(phialide)의 형태 등 형태적 특성은 이전 연구자들의 기준에 따라 조사하였다[8, 9].
분리 균주의 DNA 추출 및 염기서열 분석
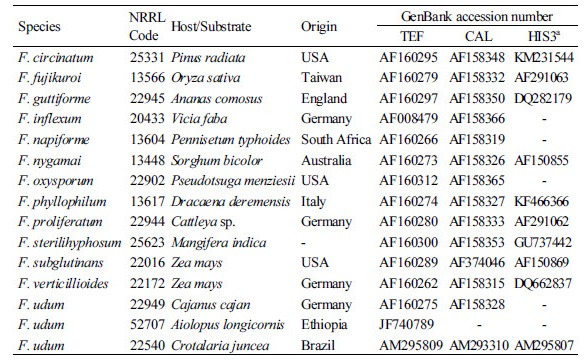
단포자 분리한 균주의 genomic DNA를 추출하기 위하여 총 6개 균주를 선발하여 각각 potato dextrose broth (PDB) 배지에 접종하고, 25°C에서 7일간 정치배양 하였다. 균사체를 miracloth로 회수하고 동결 건조시킨 후, 곱게 마쇄하여 Choi 등[10]의 방법으로 genomic DNA를 추출하고, ‒20°C에 보관하면서 실험에 사용하였다.
DNA 염기서열 분석은 O’Donnell 등[11]의 방법을 참고하여 translation elongation factor 1 alpha (TEF), calmodulin (CAL), histone H3 (HIS3) 유전자를 대상으로 실시하였다. 각 유전자의 PCR 증폭을 위하여 TEF 유전자는 EF1/EF2, CAL 유전자는 CL1/CL2A, HIS3 유전자는 H3-1a/H3-1b 프라이머쌍을 사용하였다[11, 12]. PCR 반응액은 100 ng/µL의 template DNA를 포함하여 10×Taq buffer, 2 mM dNTPs, 10 pmole/µL의 프라이머쌍, 0.5 unit의 Taq DNA polymerase를 총량 50 µL로 만들었다. 각 유전자의 annealing 온도의 경우, TEF 유전자는 60°C, CAL 유전자는 56°C, HIS3 유전자는 68°C에서 PCR 증폭하였다. 증폭된 밴드는 1% agarose gel에 전기 영동한 후 염색하여 관찰하였다. 밴드가 확인된 PCR 산물은 Wizard SV Gel & PCR Clean-up System kit (Promega, San Luis Obispo, CA, USA)를 사용하여 정제한 후, direct sequencing을 통해 염기서열을 분석하였다. 분석된 염기서열은 Clustal W 소프트웨어를 이용하여 정렬하였고, nucleotide의 유사도를 계산하여 MEGA 6.0 프로그램을 이용하여 neighbor-joining법으로 계통수를 작성하였다. 계통수 작성을 위해 사용된 reference 균주와 유전자 정보는 Table 1에 기재하였으며, 계통분석을 위한 outgroup으로 TEF와 CAL 유전자는 F. oxysporum과 F. inflexum, HIS3 유전자는 F. fujikuroi를 사용하였다.
병원성 검정
클로탈라리아와 우람콩, 태광콩 등 3개 식물을 대상으로 병원성 검정을 수행하였다. 플라스틱 포트에 클로탈라리아와 콩 종자를 파종하여 온실에서 1~3주간 재배하였다. 접종원으로 선발된 NC266-5 균주를 PDA에 7일간 배양한 후, 멸균수를 붓고 포자를 긁어 2겹의 miracloth로 걸러내었으며, 포자현탁액의 농도는 hemocytometer를 이용하여 3 × 106 spores/mL로 조정하였다. 클로탈라리아와 콩 뿌리 주변에 포자현탁액 100 mL를 관주하였고, 대조구는 같은 양의 멸균수를 관주하였다. 각 7개 식물체를 접종하였고, 접종 2~3주 후 시들음 및 황화 증상을 관찰하면서 병 발생 정도를 조사하였다. 또한 병든 식물체 지제부로부터 병원균의 재분리 여부를 조사하였다.
결과 및 고찰
병징
초기에는 클로탈라리아의 아래 잎부터 서서히 시들면서 황화되기 시작한다(Fig. 1A). 병이 계속해서 진전되면 위쪽 잎도 시들어 황화되고, 점차 식물체 전체가 시들다가 결국 말라 죽는다(Fig. 1B). 말라 죽은 줄기의 아래 부분에는 짙은 보라색 내지 검정색의 자낭각이 관찰되기도 한다(Fig. 1C). 이 병원균은 pigeonpea에도 시들음병을 일으키는데, 어린 유묘에서도 발병되지만 주로 개화기 이후에 전형적인 시들음 증상을 관찰할 수 있는 것으로 알려져 있고, 클로탈라리아 시들음병과 유사하게 잎이 황화되기 시작하다가 말라 죽게 되며, 줄기 안쪽의 도관부가 갈변되는 특징이 보고된 바 있다[13].
균학적 특성
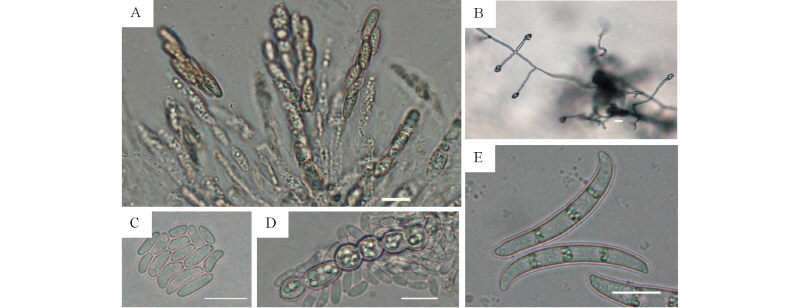
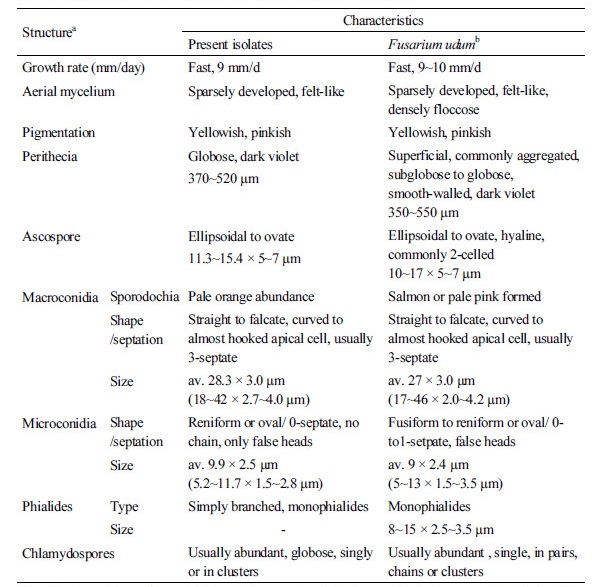
클로탈라리아 줄기에 형성된 자낭각은 구형으로 짙은 보라색을 띠며, 응집되어 형성되었으며, 크기는 직경이 370~520 µm였다. 자낭 안에 8개의 자낭포자를 형성하였고, 자낭포자는 일반적으로 1개의 격막을 가지고, 타원형이며, 크기는 11.3~15.4 × 5~7 µm였다(Fig. 2A, Table 2). 자낭포자를 단포자 분리하여 PDA 배지에 배양한 결과, 생육속도는 9 mm/day로 빠른 편이며, 기중균사는 드물게 형성하고, 황색 내지 분홍색의 색소를 형성하였다(Table 2). CLA 배지상에서 소형포자는 단경자(monophialides)에서 false heads 형태로 형성되며, 난형(oval) 혹은 신장형(reniform)으로 크기는 5.2~11.7 × 1.5~2.8 µm였다(Fig. 2B, 2C, Table 2). 대형포자는 보통 3개의 격막을 가지며, 주로 낫형으로 정단세포가 갈고리 모양으로 굽어 있으며, 크기는 18~42 × 2.7~4.0 µm였다(Fig. 2E, Table 2). 후벽포자는 매우 풍부하게 형성되며, 주로 구형으로 단일 혹은 쌍으로 형성되거나 무리 지어 형성되었다(Fig. 2D, Table 2). 이와 같은 균학적 특성에 의해 클로탈라리아 분리균을 F. udum으로 동정하였다[14].
Fusarium udum은 1910년 Butler에 의해 처음 보고 되었고, 1940년 Snyder & Hansen에 의해 F. oxysporum f. sp. udum으로 재명명되었다. 그러나 Booth [15]는 F. oxysporum에 비해 대형포자가 훨씬 굽어 있는 특성 때문에 이 균을 F. udum으로 다시 명명하였고, 지금까지 사용되고 있다[8]. 한편, 1982년 인도에서 병든 pigeonpea 줄기에 형성된 자낭각이 F. udum의 완전세대로 밝혀지면서 Gibberella indica로 기술되었으며, 서로 다른 교배형이 필요한 자웅이체성(heterothallic)으로 알려져 있다[14]. 따라서 본 연구에서 완전세대를 형성하는 것으로 확인된 국내 균주에 대해서도 교배형(mating type) 조사에 의한 다양성 분석이 필요할 것으로 생각된다. 분리된 6개 균주 중 NC266-5 균주는 국립농업과학원 미생물은행(Korean Agricultural Culture Collection)에 등록번호 KACC48401로 기탁되었다.
염기서열분석
균학적 특성에 의해 F. udum으로 확인된 클로탈라리아 분리균의 정확한 동정을 위하여 TEF, CAL, HIS3 유전자 염기서열을 분석하였다. 이들 TEF, CAL, HIS3 유전자는 Fusarium속 균의 계통학적 분류 연구에 널리 사용되는 marker로서 이미 여러 Fusarium종 복합체(species complex)를 대상으로 연구된 바 있으며[11, 16, 17], 특히 최근에는 이들 유전자 외에 largest subunit of RNA polymerase (RPB1)와 second largest subunit of RNA polymerase (RPB2) 영역이 Fusarium 계통학적 연구에 사용되고 있다[18, 19]. 그러나 F. udum의 경우, RPB1과 RPB2의 염기서열 정보가 NCBI에 등록되어 있지 않아서 본 연구에서는 TEF, CAL, HIS3 유전자를 대상으로 염기서열 분석을 수행하였다.
TEF 유전자 염기서열 분석 결과, 클로탈라리아 분리균주는 F. phyllophilum NRRL13617과 95.9~96.4%의 상동성을 나타내었고, F. udum NRRL22949, NRRL52707, NRRL22540과는 각각 95.9~96.2%, 94.4~94.7%, 94.0~94.4%의 상동성을 나타내었다(Fig. 3A). CAL 유전자 염기서열의 경우, F. udum NRRL22949, NRRL22540과는 각각 99.7%의 상동성을 보였고, F. phyllophilum NRRL13617과는 99.2%의 상동성을 나타내었으며(Fig. 3B), HIS3 유전자는 F. udum NRRL22540과 99.6~99.8%, F. phyllophilum NRRL13617과는 97.6~ 97.8%의 상동성을 나타내었다(Fig. 3C).
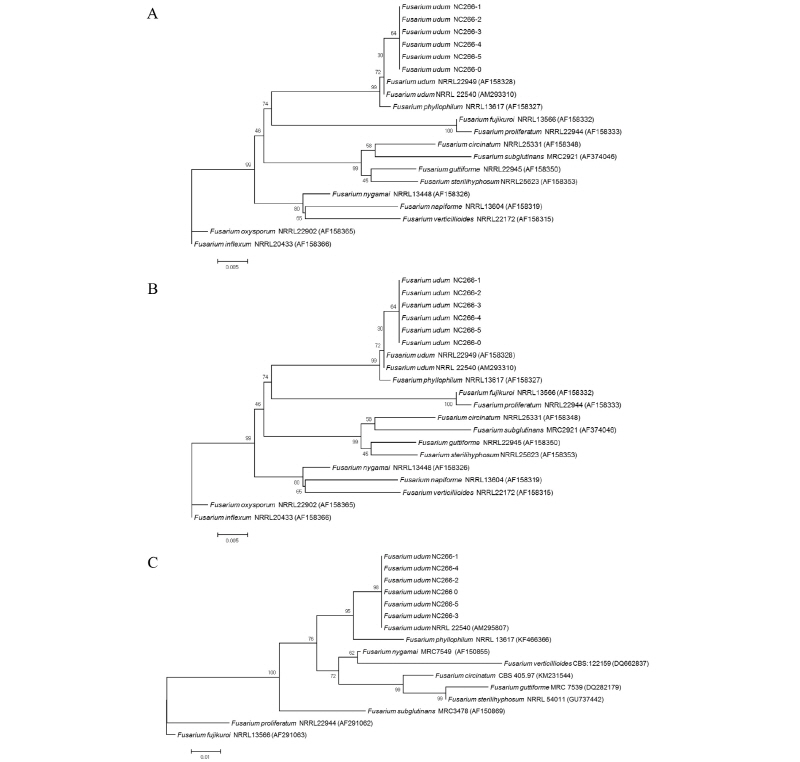
Fig. 3. Phylogenetic trees based on translation elongation factor 1 alpha (A), calmodulin (B), and histone 3 (C) genes sequence data for Fusarium udum isolated from infected sunn hemp plants and reference species. Sequence data were obtained from the NCBI GenBank database. The trees were generated using neighbor-joining analysis and Kimura 2-parameter model. The numbers in parentheses represent the sequence accession number in Genbank. Bar represents the number of nucleotide substitutions per site.
이와 같이 클로탈라리아 분리균은 CAL, HIS3 유전자의 계통수에서는 F. udum과 가장 가까운 것으로 나타났으나 TEF 유전자의 계통수에서는 F. phyllophilum과 가장 근연한 것으로 나타났는데, F. phyllophilum은 대형포자를 매우 드물게 형성하며, 소형포자는 mono-형 혹은 poly-형의 경자(phialides)에서 false heads와 짧은 사슬 형태로 형성하고, 후벽포자가 없는 것이 특징으로 완전세대 역시 밝혀지지 않아 형태적으로 F. udum과 큰 차이가 있어 구분이 가능하다[9]. 따라서 Fusarium균의 정확한 동정을 위해서는 여러 개 유전자를 사용하는 multilocus sequence typing (MLST)뿐 아니라 형태적 특성에 대한 조사가 반드시 필요할 것으로 판단된다. 특히 TEF 유전자의 분석 결과, Fig. 4에서 보는 바와 같이 국내 균주의 intron 부위에 10~27개의 nucleotide가 삽입된 것을 볼 수 있었고, 이로 인해 계통수에서 F. phyllophilum과 근연종으로 나타난 것으로 판단된다. Laurent 등[20]은 6개 F. graminearum 균주의 전체 유전자 염기서열을 비교 분석한 결과, 약 24만여개의 변이형(SNP, InDels)을 가진 다형성이 있음을 밝히면서 이는 기주-병원균 상호작용에 관여하는 유전자의 다양화 및 염색체 구조의 진화적 결과로 추정한다고 하였는데, 본 연구에서 분리된 균주도 이러한 진화 과정의 결과일 것으로 생각되며, 향후 이에 대한 연구가 필요할 것으로 생각된다. 본 연구에서 분석된 균주의 HIS3, TEF, CAL 유전자 염기서열은 NCBI GenBank에 KU599899~KU599916의 accession number로 등록하였다.

Fig. 4. Comparison of translation elongation factor 1 alpha gene sequences alignment between isolates from the infected sunn hemp plant and the reference strains of Fusarium udum and F. phyllophilum. Black box indicates that 6 Korean isolates have insertions of 10~27 nucleotides in intron site of translation elongation factor 1 alpha (TEF) gene.
병원성 검정
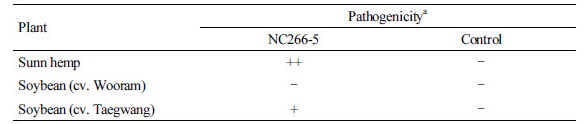
분리 균주의 병원성을 확인하기 위하여 클로탈라리아 종자를 파종하고 7일 후에 포자현탁액을 관주 처리하였다. 접종 7일 후, 유묘가 시드는 증상이 나타났고, 심한 경우 유묘가 완전히 시들어 고사하였다. 또한 약 3주간 키운 클로탈라리아 유묘에 포자현탁액을 관주 처리한 결과, 접종 2주 후부터 하엽이 시드는 증상을 관찰할 수 있었고, 3주 후에는 완전히 고사하는 개체가 나타났으며, 고사된 식물체 줄기에 흰색 또는 연분홍색의 포자가 형성되기도 하였다. 클로탈라리아는 콩과식물이며, 분리 균주인 F. udum은 pigeonpea에도 병을 일으키므로, 국내에서 재배되는 우람콩과 태광콩을 대상으로 병원성 검정을 수행하였다. 파종 2주 후, 포자현탁액을 관주 처리하여 접종한 결과, 태광콩은 일부 식물체에서 생육이 억제되고, 약하게 시드는 증상을 관찰할 수 있었으나 우람콩은 하엽의 엽맥 사이만 갈색으로 변색되었고, 시드는 증상은 나타나지 않았다(Table 3). 인도에서는 시들음병 저항성 pigeonpea를 스크리닝하기 위하여 다양한 방법으로 F. udum을 접종하였는데, 토양접종이나 수경재배 접종법 보다 포자현탁액 관주 접종이 가장 효율적이라고 하였다[21]. 현재까지 F. udum은 pigeonpea와 클로탈라리아 등에 병을 일으키는 것으로 보고되어 있으나, 본 연구에서는 콩에도 약하게 병을 일으킬 수 있음이 확인되었다. 인도에서는 32개 F. udum 균주를 유전형이 다른 12개의 pigeonpea에 접종한 결과, 5개의 병원성 변이형을 확인하였고[7], 케냐에서도 17개 F. udum을 7개 pigeonpea 품종에 접종한 결과 5개 병원형으로 그룹화하였다[6]. 이와 같이 F. udum은 다양한 병원성 변이를 나타낼 수 있고, 국내에서도 이 병원균의 분포가 확인된 만큼 다른 콩과 작물에 대한 지속적인 병 발생 모니터링이 필요할 것이다.
이상과 같이 국내 클로탈라리아 시들음 증상에서 분리한 균은 형태적, 배양적 특성과 DNA 염기서열 분석에 의해 F. udum으로 동정되었으며, 완전세대를 형성하는 것으로 확인되었다. 또한, 클로탈라리아와 태광콩에서 병원성이 확인되었으며, 국내에서 이 균의 분포가 처음으로 확인되었다. 따라서 본 결과를 바탕으로 Fusarium udum Butler에 의한 클로탈라리아 시들음병을 국내에서 처음으로 보고하고자 한다.