서 론
내생균류는 숙주식물에 해로운 영향 없이 살아있는 식물체 내에 존재하며 식물과 공생을 이루며 살아간다[1]. 내생균류는 기후에 따라 식물체 내에서 다양하게 존재하며 이차대사산물과 생물학적 조절인자 등을 생성하여 숙주식물이 외부 환경의 생물학적 또는 비생물학적 스트레스에 대한 저항성 및 영양분 획득에 더 유리하게 도와준다[2-5].
Sordariomycetes강은 Ascomycota문에서 두 번째로 큰 강으로[6], 32개 목, 105개 과, 1,331개 속, 10,000개 이상의 종으로 구성되어 있다[7]. 대부분의 종들은 육상생태계(terrestrial)에 존재하지만, 일부 종들은 수생태계(aquatic habitats)에서도 발견된다[6, 8].
본 연구에서는 제주의 습지식물로부터 분리한 내생균류 3종(Acremonium tubakii, Colletotrichum cliviae, Cylindrocarpon pauciseptatum)이 Sordariomycetes에 속하는 국내 미기록종으로 보고하고자 한다.
재료 및 방법
제주도 습지(수장동 습지, 남생이 못)에서 2016년 8월에 수생식물을 채집하였다(Table 1). 채집한 수생식물의 뿌리는 내생균류를 분리하기 위하여 뿌리의 외부를 Tween80과 1% 차아산염소나트륨(NaOCl)을 이용하여 표면소독 하였다. 세척한 뿌리는 1 cm 길이로 잘라 Hagem media (HM; 5% C6H12O6 [w/v], 5% malt extract [w/v], 1.5% agar [w/v], 0.05% KH2PO4 [w/v], 0.05% NH4Cl [w/v], 0.05% MgSO4·7H2O [w/v], 0.05% FeCl3 [w/v])에 올려 25°C에서 일주일간 배양하였다. 자른 뿌리의 단면에 균사가 나타나면 순수분리를 위하여 HM 배지에 계대배양 하였다. 분리한 균의 균사체 확보를 위하여 potato dextrose agar (PDA; Difco, Detroit, MI, USA)에 계대배양 하였다[9].
분리된 균의 동정을 위하여 균사체는 2 mL 튜브에 glass bead와 함께 넣어 균질화시킨 뒤 NucleoSpin Plant Ⅱ kit (Macherey-Nagel, Düren, Germany)를 이용하여 genomic DNA를 추출하였다. 추출된 genomic DNA는 internal transcribed spacer (ITS) 영역의 증폭을 위하여 primer로 ITS1 (5'-TCCGTAGGTGAACCTGCGG-3')과 ITS4 (5'-TCCTCCGCTTATT GATATGC-3')를 이용하여 PCR을 진행하였다[10]. PCR 산물은 Bioedit 7.2.5를 이용하여 DNA 염기서열을 정렬 및 편집하였다[11]. 비교 염기서열은 NCBI의 GenBank를 이용하여 유사도가 가장 높은 종을 기준으로 동정하였다. 계통수 작성은 neighbor-joining method로 MEGA7을 이용하였다[12]. 분리된 균은 NCBI Genbank에 등록하여 accession number를 부여 받았다.
결과 및 고찰
Acremonium tubakii W. Gams. (Fig. 1)
ITS rDNA 영역의 염기서열(KY988411)을 NCBI에 등록된 염기서열과 비교하여 분석한 결과, 이 종은 A. tubakii (Genbank accession number KF915990)와 100%의 상동성을 보였으며, 계통수 상에서도 A. tubakii group에 속하는 것을 확인하였다.
이 균주는 PDA 배지에 배양하였을 때 25°C에서 7일 동안 20 mm 정도로 느리게 생장하였으며, 균사체는 전체적으로 흰색을 띄었다. Acremonium속은 대부분 배지 상에서 느리게 생장한다고 보고된 바 있다[13].
이 균주의 포자는 PDA 배지에 배양하였을 때 14일 이후에 생성하였다. 포자 형태는 끝이 둥근 방망이 형태이며 균사와 포자가 연결된 체인 형태가 관찰되었는데 이것은 Acremonium속에서 대부분 나타나는 특징이다[13]. 크기는 길이 7~10 µm × 폭 3~5 µm이다.
Specimen examined 대한민국 제주특별자치도 제주시, 수생식물 마름, 2016년 8월 10일 채집, NNIBRFG2982, 국립낙동강생물자원관
Synonym Cephalosporium polyaleurum Tubaki
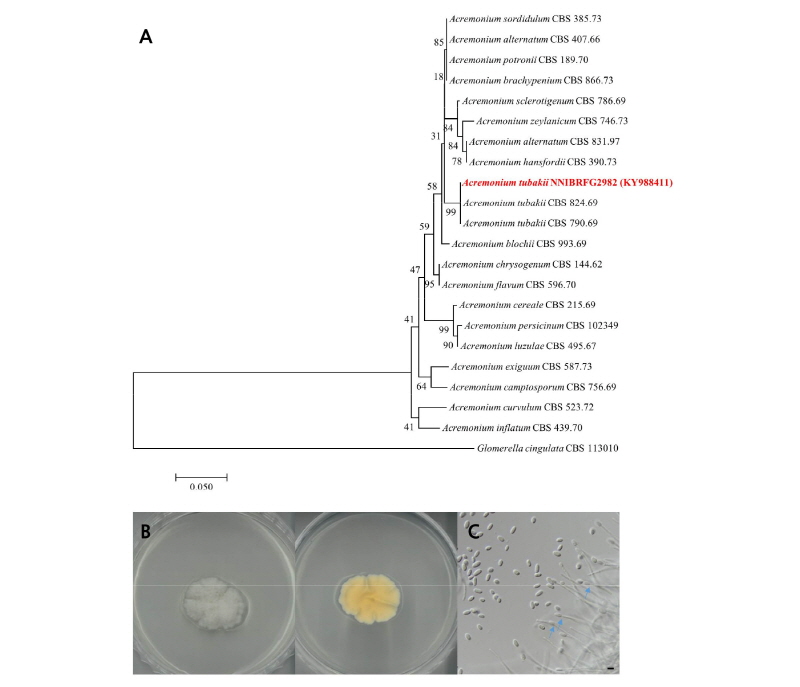
Fig. 1. Acremonium tubakii (NNIBRFG2982). A, phylogenetic analysis using neighbor-joining method based on internal transcribed spacer (ITS). The percentage of replicate trees in which the associated taxa clustered together in the bootstrap test (1,000 replicates) are shown next to the branches. Glomerella cingulate was used as outgroup; B, colony on potato dextrose agar at 7 days; C, conidia (scale bar = 10 µm).
Colletotrichum cliviae Y. L. Yang, Z. Y. Liu, K.D. Hyde & L. Cai. (Fig. 2)
ITS rDNA 영역의 염기서열(KY988414)을 NCBI에 등록된 염기서열과 비교하여 분석한 결과, 이 종은 C. cliviae (Genbank accession number KY986892)와 100%의 상동성을 보였으며, 계통수 상에서도 C. cliviae group에 속하는 것을 확인하였다. C. cliviae는 잎에 점(leaf spot)이 생기는 병원성을 띄거나 징후가 없는 내생균으로 식물체에 존재한다고 보고된 바 있다[14].
최초 보고된 바에 의하면 PDA 배지 상에서 흰색을 띄다가 5일 후부터 어두운 암녹색을 띈다고 하였다[15]. 이 균주 또한 PDA 배지에서 배양하였을 때 25°C에서 7일동안 60 mm 생장하였으며, 균사체는 전체적으로 어두운 녹색을 띄었다. 투명하고 약간 굽은 형태의 포자를 가졌으며 끝부분은 둥근 형태였다. 크기는 길이 21~23 µm × 폭 3~5 µm이다.
Specimen examined 대한민국 제주특별자치도 제주시, 수생식물 마름, 2016년 8월 10일 채집, NNIBRFG2984, 국립낙동강생물자원관
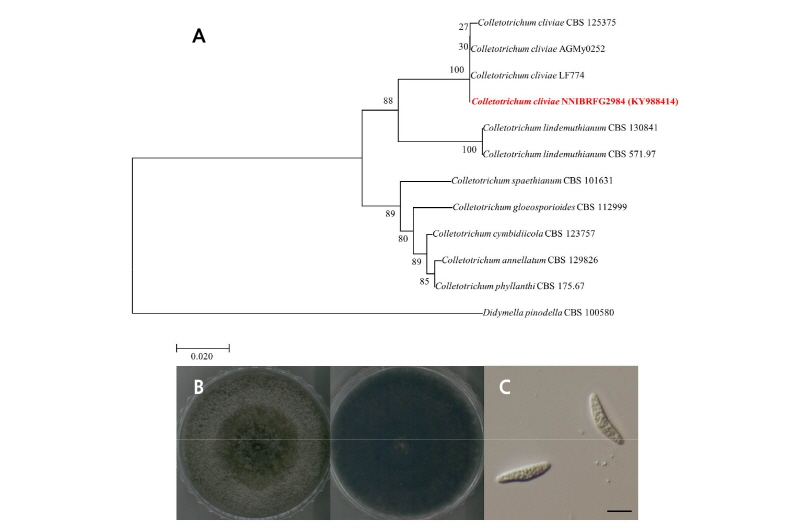
Fig. 2. Colletotrichum cliviae (NNIBRFG2984). A, phylogenetic analysis using neighbor-joining method based on internal transcribed spacer region (ITS). The percentage of replicate trees in which the associated taxa clustered together in the bootstrap test (1,000 replicates) are shown next to the branches. Didymella pinodella was used as outgroup; B, colony on potato dextrose agar at 7 days; C, conidia (scale bar = 10 µm).
Cylindrocarpon pauciseptatum Schroers & Crous, Mycol. Res. (Fig. 3)
ITS rDNA 영역의 염기서열(KY988477)을 NCBI에 등록된 염기서열과 비교하여 분석한 결과, 이 종은 C. pauciseptatum (Genbank accession number JF735305)와 99.8%의 상동성을 보였으며, 계통수 상에서도 C. pauciseptatum group에 속하는 것을 확인하였다. C. paucisep-tatum은 식물체에 내생균으로 존재하며 뿌리 등에서 분리되어 보고된 바 있다[16].
최초 보고 된 바에 의하면 이 종은 20°C 또는 25°C에서 잘 생장하며 PDA 배지에서 배양하였을 때 흰색에서 갈색으로 생장한다고 하였다[17]. 이 균주 또한 PDA 배지에서 배양하였을 때 25°C에서 7일동안 25 mm로 생장하였다. 균사체는 가운데 부분이 흰색을 띄다가 가장자리로 갈수록 황토색과 갈색, 노란색을 띄면서 생장하였다. 포자는 2개에서 4개의 격막으로 나누어져 있는 원주형(cylindronical) 모양의 포자를 가졌으며 약간 굽어져 있고 끝 부분은 둥근 형태였다. 크기는 길이 36~38 µm × 폭 4~6 µm이다.
Specimen examined 대한민국 제주특별자치도 제주시, 수생식물 좁은미꾸리낚시, 2016년 8월 9일 채집, NNIBRFG3040, 국립낙동강생물자원관
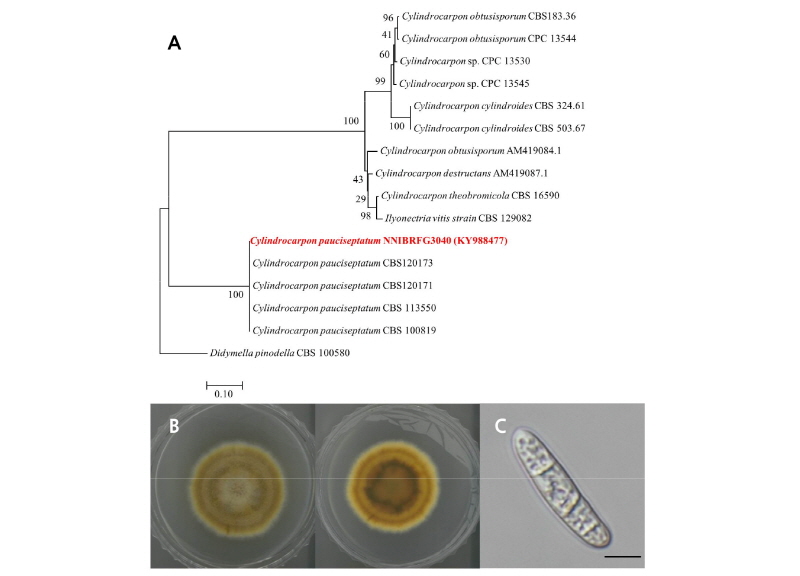
CFig. 3. ylindrocarpon pauciseptatum (NNIBRFG3040). A, phylogenetic analysis using neighbor-joining method based on internal transcribed spacer region (ITS). The percentage of replicate trees in which the associated taxa clustered together in the bootstrap test (1,000 replicates) are shown next to the branches. Didymella pinodella was used as outgroup; B, colony on potato dextrose agar at 7 days; C, conidia (scale bar = 10 µm).