서론
표고는 담자균류에 속하는 버섯으로 치마버섯(Schizophyllum commune)이나 재두엄먹물버섯(Coprinopsis cinerea)과 같이 사극성 교배계를 갖는다[1-3]. 사극성 교배계에서 두 균사 간의 교잡 화합성은 흔히 A와 B로 일컬어지는 서로 독립된 두 유전자좌에 의해 결정된다[4]. 성공적인 교잡은 두 유전자좌에 있는 대립유전자들에 의해 구분되는 교배형이 균사 간에 서로 다를 때 이루어진다[5]. A 유전자좌에는 homeodomain (HD) 전사인자, B 유전자좌에는 페로몬과 페로몬 수용체들이 암호화 되어있으며[4], 이들은 균사 간의 융합 이후에 일어나는 핵 이동(nuclear migration), 핵 쌍(nuclei pairing), 꺾쇠세포(clamp cell)의 형성 및 융합과 같은 일련의 과정을 조절하는 것으로 알려져 있다[6].
표고의 A 교배형은 A 유전자좌에 있는 한 쌍의 HD 전사인자들에 의해 결정된다[7,8]. 표고 A 유전자좌의 구조는 표고 유전체 서열에서 재두엄먹물버섯(Coprinopsis cinerea)의 A 유전자좌에 있는 HD1 및 HD2 유전자와 상동성이 높은 유전자를 탐색하는 과정에서 2014년 처음 밝혀졌다[7]. 이 구조에는 다른 담자균들에서 보고된 바와 같이 두 개의 HD 전사인자들이 N말단을 마주하고 있는 형태로 암호화 되어있고, 각각의 대립유전자에는 dimerization motif와 핵 위치 신호 및 HD 도메인이 보존되어 있다[7-10]. 또한 HD 전사인자의 대립유전자들 간에 N말단의 변이가 크게 나타났다[8]. 이 영역은 HD 전사인자들 간의 이종 이합체(heterodimer) 형성 및 그에 따른 전사인자 활성화에 기여함으로써 서로 다른 A 교배형을 갖는 균사 간의 교잡이 가능한 것으로 보고된 바 있다[11-13]. 또한 표고에서 이 영역의 염기서열에 대한 정보로 A 교배형을 구분하는 것이 가능함이 교잡 실험을 통해 증명되었다[8].
현재까지 표고의 A 교배형을 구분하는 분자마커로는 두 가지가 보고되었다. 하나는 대립유전자들 간의 인트론 (intron) 길이 차이를 통한 구분 방법이다. 이 방법은 프라이머 두 쌍을 이용하여 6종의 A 교배형을 서로 구분하는 것으로 A 유전자좌에 있는 두 HD 전사인자 각각의 인트론을 포함하는 영역을 증폭한 뒤 길이를 비교하여 구분한다[7]. 다른 방법은 HD 전사인자의 N말단의 다양성에 기초한 방법이다. 하나의 A 유전자좌에 있는 두 HD 전사인자의 N말단과 두 유전자 사이 구간을 포함하는 약 1 kb의 영역을 한 쌍의 프라이머를 이용하여 증폭 및 염기서열을 밝힘으로써 A 교배형을 구분한다[8]. 이 방법은 첫 번째 방법에서 구분한 6종의 A 교배형을 포함해 63종의 A 교배형을 구분할 수 있음이 보고되었다[8]. 첫 번째 방법은 적용 가능성이 증명된 교배형의 종류가 6가지뿐으로 그 수가 적다는 한계가 있다. 두 번째 방법은 현재까지 보고된 방법 중 가장 정확하며 다양한 A 교배형을 구분할 수 있다는 장점이 있으나, 과정상 염기서열 분석이 반드시 수반되어야 한다는 점과, 이로 인해 이핵균주의 A 교배형을 분석할 때에 두 개의 핵에서 유래한 서로 다른 증폭산물을 분리하기 위해 클로닝 과정이 추가로 포함되어야 한다는 한계가 있다.
본 연구에서는 이러한 한계를 극복하기 위하여 표고의 A 교배형을 구분할 수 있는 cleaved amplified polymorphic sequence (CAPS) 마커를 개발하고자 하였다. CAPS 마커는 DNA 염기서열의 변이로 인한 제한효소 인식부위의 차이를 이용한 방법으로, 염기서열 분석에 비해 비용이 저렴하고, 절차가 간단하며, 특별한 장비를 필요로 하지 않는다는 장점이 있어 다양한 생물 종에서 널리 쓰이고 있다[14-17]. 나아가 CAPS 마커는 공우성 마커로 기능할 수 있기 때문에 이핵균주의 두 가지 A 교배형을 동시에 감별할 수 있다는 장점이 있다. 개발된 CAPS 마커를 활용하여 표고의 품종 및 야생종을 통틀어 가장 많이 발견되는 11가지의 A 교배형(A1, A2, A3, A4, A5, A7, A11, A15, A17, A23, A27)이 구분 가능함을 확인하였다. 또한 해당 방법이 이핵균주의 교배형을 확인하는 데에도 적용 가능함을 확인하였다. 본 연구 결과는 비교적 간편하게 A 교배형을 구분함으로써 표고 신품종 육종을 위한 교배형 분석에 활용하여 교잡 확률을 높이는 데 기여하거나, 표고 담자포자의 교배형의 불균일 분포[18,19]와 같은 교배형 관련 생명현상을 연구하는 데에도 활용될 수 있을 것으로 기대된다.
재료 및 방법
균주 배양 및 DNA 추출
실험에 사용한 표고 균주는 국립산림과학원에 보존되어 있는 균주를 이용하였다(Table 1). 보존된 균주는 potato dextrose agar (PDA; BD Dfico, MD, USA) 평판배지에 계대하여 25℃에서 2주간 암배양한 뒤 DNA 추출에 사용하였다.
배양된 균사체는 cell scraper (SPL, Seoul, Korea)를 이용하여 모은 뒤 액체질소에 얼려 막자사발로 곱게 간 후, 제조사에서 제공하는 표준 지침에 따라 DNeasy Plant mini kit (Qiagen, CA, USA)를 이용하여 gDNA 추출에 이용하였다.
PCR 및 DNA 염기서열 분석
PCR은 추출된 gDNA 10 ng을 주형으로 C1000 thermal cycler (Biorad, CA, USA)를 이용하여 94℃에서 5분 변성 후, 94℃에서 30초, 55℃에서 30초, 72℃에서 45초로 35회 반복하고, 72℃에서 7분간 추가로 반응을 진행하였다. 이때 PCR 반응액은 AccuPower Pfu PCR premix (Bioneer, Daejeon, Korea)를 사용하였으며, 프라이머는 A_mat_F: 5’-GTGCTTCGTATGCTGGG-3’, A_mat_R: 5’-AGGAGCCAGGTGTAGGA-3’를 사용하였다[8]. 증폭된 PCR 산물은 1% 아가로스 젤 전기영동으로 약 1 kb의 사이즈로 증폭되었는지 확인한 뒤 해당 사이즈의 밴드만 AccuPrep PCR/Gel DNA purification kit (Bioneer, Daejeon, Korea)를 이용하여 정제하였다.
정제된 PCR 산물은 TOPcloner blunt kit (Enzynomics, Daejeon, Korea)를 이용하여 플라스미드에 클로닝한 뒤 염기서열을 분석하였다 (Macrogen, Seoul, Korea). 분석된 염기서열은 미국 국립생물정보센터(NCBI)의 BLAST search tool (https://blast.ncbi.nlm.nih.gov/Blast.cgi)을 이용하여 A 교배형의 염기서열이 맞는지 확인하였다. 염기서열이 확인된 플라스미드를 주형으로 다시 PCR하여 원하는 A 교배형을 증폭한 후 제한효소 처리에 사용하였다.
제한효소 선정
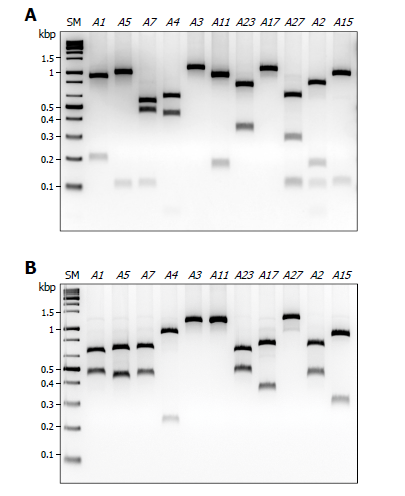
표고의 품종 및 야생종에서 가장 빈번하게 발견되는 11가지 A 교배형(A1, A2, A3, A4, A5, A7, A11, A15, A17, A23, A27)의 염기서열을 query로 CAPS Designer (https://solgenomics.net/tools/caps_designer/caps_input.pl)를 이용해 적합한 제한효소를 선정하였다. 전기영동을 통한 DNA 단편(DNA fragment)들의 구분을 명확하게 하기 위해, DNA 단편의 개수가 4개 이하이면서 가장 많은 종류의 교배형을 구분할 수 있는 제한효소를 우선으로 선별하였다. 그 결과, HaeIII가 가장 적합한 것으로 확인되었다. 하지만 HaeIII는 제한효소 인식부위가 없는 A3와 A17 및 DNA 단편의 크기와 양상이 서로 유사한 A1과 A11, A5와 A15의 구분에 한계가 있어 이를 보완하기 위해 해당 교배형들을 구분하는 데 적합한 추가 제한효소로 EcoRI을 선정하였다.
제한효소 처리 및 전기영동
HaeIII와 EcoRI 제한효소는 제조사인 Enzynomics (Daejeon, Korea)의 표준 지침에 따라 처리하였다. 약 300 ng의 DNA를 1× FastCut buffer에서 30분동안 37℃로 5 unit의 HaeIII와 반응시켰다. 마찬가지로 약 300 ng의 DNA를 1× EzBuffer EcoRI에서 30분동안 37℃로 10 unit의 EcoRI과 반응시켰다. 반응 결과물은 2.5% 아가로스 젤 전기영동을 통해 확인하였다.
결과 및 고찰
CAPS 마커를 통한 A 교배형의 구분
표고의 A 교배형은 96종의 재배품종 및 31종의 야생종에 대한 염기서열을 분석 결과 63종이 확인된 바 있다[8]. 본 연구에서는 이 가운데 재배품종 및 야생 표고를 통틀어 가장 빈번하게 존재하는 것으로 보고된 11가지 A 교배형(A1, A2, A3, A4, A5, A7, A11, A15, A17, A23, A27)을 구분할 수 있는 CAPS 마커의 개발을 목표로 하였다. 선정된 제한효소로 11가지 A 교배형을 구분할 수 있는지 확인하기 위해, 기존에 보고된 프라이머를 이용하여[8] 각각의 A 유전자좌의 변이 영역을 증폭한 뒤 HaeIII를 처리하였다. 그 결과 예상한 바와 같이 제한효소 인식부위가 없는 A3와 A17을 제외한 나머지 9종의 A 교배형에서 서로 다른 양상을 확인하였다(Fig. 1A). 특히 가장 많이 발견된 네 개의 A 교배형(A1, A5, A7, A4)을 명확히 구분할 수 있음을 확인하였다. 하지만 A1과 A11, A5와 A15는 DNA 단편들의 크기가 차이가 없어 일반적인 전기영동만으로 구분하기 어려웠다(Fig. 1A).
따라서 HaeIII만으로 구분이 어려운 세 쌍의 교배형을 구분하기 위하여 추가적인 제한효소로 EcoRI을 선정하였으며, EcoRI 처리 결과 A3과 A17, A1과 A11, A5와 A15를 구분할 수 있었다(Fig. 1B). 즉, HaeIII와 EcoRI 두 가지 제한효소의 처리만으로 11가지의 A 교배형을 구분할 수 있음을 확인하였다. 본 방법은 단핵균사의 A 교배형을 판별할 때 유용할 것으로 기대된다.
이핵균주의 A 교배형 판별에 대한 적용
CAPS 마커는 공우성 마커이기 때문에 여러 대립유전자가 섞여있는 DNA에 대해서도 적용이 가능하다. 이에 본 연구에서 개발한 CAPS 마커가 이핵균주의 A 교배형 판별에 적용될 수 있는지 살펴보았다. 품종명 수향고(Suhyang)의 A 유전자좌 증폭 산물에 HaeIII를 처리한 결과, 5개의 DNA 단편을 확인할 수 있었다(Fig. 2A). 이를 교배형 별 DNA 단편(Fig. 1A)과 비교하였을 때, 500 bp 부근의 두 밴드를 통해 수향고는 A7을 반드시 포함함을 알 수 있다. 따라서 나머지 하나의 교배형에서 약 1 kb 부근과 200 bp 부근의 밴드가 생성됨을 알 수 있으며, 이에 적합한 교배형은 A1뿐이므로 수향고의 A 교배형은 A1 A7으로 예상할 수 있었다. 이러한 예상은 EcoRI 결과와도 유사하였으며, 실제로 염기서열 분석을 통해 확인된 수향고의 A 교배형과 일치하였다[8]. 따라서 본 방법을 이핵균주의 A 교배형 판별에도 적용할 수 있음을 확인하였다.
활용 방안 및 한계
본 연구에서는 기존에 보고된 프라이머를 이용하여[8] A 유전자좌의 변이 영역을 증폭한 뒤 HaeIII와 EcoRI 제한효소를 처리함으로써 다양한 표고의 품종 및 야생종에서 가장 빈번하게 발견되는 11가지의 A 교배형을 구분할 수 있음을 확인하였다. 또한 해당 방법은 이핵균주에도 적용이 가능하므로 염기서열 분석 및 클로닝의 과정 없이도 특정 표고의 교배형을 보다 쉽게 판별하는 데 도움이 될 것으로 기대된다.
한편, 교잡 과정에서의 꺾쇠세포 형성 및 대치선 관찰을 통해 예상된 표고의 A 교배형은 염기서열 분석을 통해 확인된 63가지보다 많을 것으로 보고되었다[20,21]. 특히 야생 표고에서 A 교배형이 다른 담자균들에서와 같이 빠르게 진화하여 더욱 다양해질 수 있음이 알려져 있다[8,22]. 따라서 11가지의 A 교배형만을 구분할 수 있는 CAPS 마커는 그 적용 범위에 분명한 한계가 있다. 하지만 이들 11가지 교배형은 조사된 96종의 재배품종에서 90%의 빈도로 확인된 바 있으므로[8], 재배품종의 교배형을 밝히는 데에 특히 유용할 것으로 기대된다. 이렇게 밝힌 교배형은 서로 다른 교배형을 가진 균사 간의 교잡만 시도하게 함으로써 교잡 확률을 높이고, 교잡에 필요한 노동력과 시간을 절약하는 데 기여할 수 있을 것이다. 특히 형질의 우수성이 확인된 재배품종이 신품종 육종의 재료로 활용됨을 고려할 때, 그 유용성을 예상할 수 있다. 나아가 대량의 A 교배형을 비교적 간편하게 분석할 수 있으므로, 표고 담자포자의 교배형 불균일 분포 현상[18,19]과 같이 교배형과 관련된 생명현상을 연구하는 데에도 활용할 수 있을 것으로 기대된다.
적요
표고는 한국에서 가장 많이 소비되는 식용버섯 중 하나이다. 표고는 사극성의 교배계를 따르며, 표고의 교배형은 자웅이주성의 다른 담자균류와 마찬가지로 서로 독립적인 두 유전자좌, A와 B에 의해 결정된다. 표고의 A 유전자좌에는 한 쌍의 homeodomain (HD) 전사인자가 암호화되어 있으며 이들의 N말단에서 나타나는 높은 변이가 A 교배형의 다양성에 중요한 것으로 알려져 있다. 본 연구에서는 표고 품종과 야생종에서 많이 발견되는 11종의 A 교배형을 구분할 수 있는 CAPS 마커를 개발하고자 하였다. A 유전자좌에서 변이가 큰 부분을 PCR을 통해 증폭한 뒤 두 가지 제한효소 HaeIII와 EcoRI로 절단하여 DNA 단편의 크기 및 양상을 살펴봄으로써 11종의 A 교배형을 서로 구별할 수 있었다. 또한 해당 방법이 이핵균주의 교배형을 확인하는 데 활용할 수 있는지도 살펴보았다.
Acknowledgements
This work was supported by the Golden Seed Project of ‘Breeding of new strains of shiitake for cultivar protection and substitution of import [213007-05-4-SBH10]’ provided by the Ministry of Agriculture, Food and Rural Affairs, Ministry of Oceans and Fisheries, Rural Development Administration and Korea Forest Service.