서론
Aspergillus 속 곰팡이는 자연계에 널리 분포하는 부생성, 기회감염성 곰팡이로 Penicillium 속과 함께 다양한 작물의 저장 중 부패를 유발하는 것으로 알려져 있다[1]. 그러나 저장 중인 곡물 뿐만 아니라 재배 중인 밀, 벼, 옥수수, 수수, 견과류, 목화, 고추 등의 작물들과 가공식품에서도 발견된다[2,3]. A. flavus와 A. parasiticus를 비롯한 18여 종의 Aspergillus 속 곰팡이는 인간과 동물에 유해한 아플라톡신 독소를 생성한다[4]. 아플라톡신은 B1, B2, G1, G2를 포함하여 20여 종이 알려져 있는데, B1의 독성이 가장 강하며 발암, 돌연변이, 기형유발, DNA 손상, 면역력 억제 등을 일으킨다[5,6]. 아플라톡신은 Group 1 발암물질로 분류되어 대부분의 국가들이 강력히 규제하고 있다[1].
전 세계적으로 쌀, 옥수수 등 곡물류의 최대 37.6%가 아플라톡신에 오염되어 있으며[7], 특히 땅콩 등의 견과류, 무화과 등의 건조과일, 고춧가루의 오염률이 높은 것으로 보고되고 있다[3]. 땅콩의 주요 생산국인 중국에서 저장 땅콩의 19%가 0.01–1,602 μg/kg, 땅콩 오일(427점)의 22.5%가 15.4–49.9 μg/kg의 아플라톡신에 오염된 것으로 보고되었다[8,9]. 국내의 경우 유통 중인 견과류 및 가공식품에서 아플라톡신의 오염이 보고되었다. 박[10]에 의하면, 2003년 국내에서 유통되고 있는 땅콩 및 땅콩버터의 아플라톡신 B1 오염률은 각각 25%와 17%로 국산 땅콩 28점 중 4점, 수입산 12점 중 6점에서 오염이 확인되었으며, 평균 오염수준은 수입산이 높은 것으로 보고되었다. 또한 국내 유통 중 견과류(가공식품 포함), 장류, 곡류(가공품)에 대한 아플라톡신 B1 오염도 조사 결과 식품 기준치를 초과하지는 않았으나 견과류 및 그 가공식품의 검출빈도가 가장 높았다[11].
국내에서 땅콩의 아플라톡신 오염이 일부 보고되었으나 A. flavus의 발생에 대한 연구는 전무하다. 이에 본 연구에서는 아플라톡신 오염 빈도가 높은 땅콩의 저장 중 독성 곰팡이 및 아플라톡신의 오염도를 조사하였다. 또한 땅콩에서 분리된 A. flavus의 다양성 및 아플라톡신 독소의 생성능을 평가하였다.
재료 및 방법
땅콩 시료 수집 및 균 분리
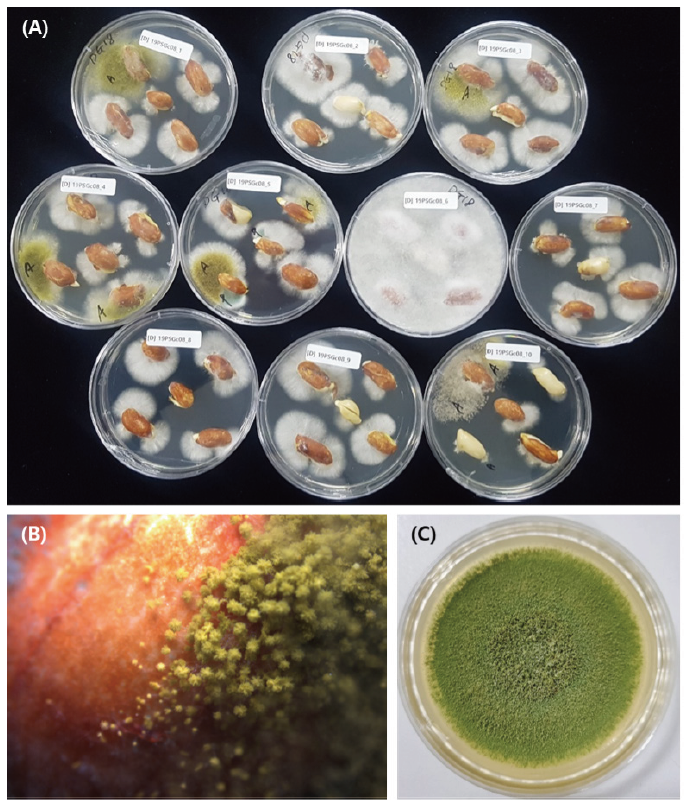
2019년 10월에 고창 지역에서 수확되어 저장 중인 피땅콩을 2020년 2월과 4월에 수집하여 독성곰팡이 오염현황을 조사하였다. 저장창고 2곳에서 3개의 개별 포장백(30 kg)으로부터 각각 1 kg씩 채집하였다. 시료 채집 기간 동안 저장창고의 평균온도 및 상대습도는 각각 7.9±2.5℃와 68.0±8.1%, 7.5±1.4℃와 68.2±1.0%의 조건이었다. 곰팡이 오염현황 조사 및 균 분리를 위하여 땅콩의 겉껍질을 제거하여 알땅콩을 수집하였다. 알땅콩은 속껍질을 유지한 채 1% sodium hypochlorite 용액에 3분간 침지하여 표면 소독하고 살균수로 2회 세척 후 여과지로 물기를 제거한 다음, 반으로 절단하여 potato dextrose agar (PDA) (streptomycin 600 μg/mL) 와 dichloran-glycerol (DG18) agar 배지에 각각 50개씩 치상하였다. 25℃ 항온기에서 5일간 배양한 후 현미경 하에서 형태학적 특성을 기반으로 곰팡이 오염도를 조사하였으며, 균 분리 및 동정을 위해 단일 균총의 포자현탁액을 water agar에 도말, 25℃ 항온기에서 18시간 배양 후 단포자를 분리하였다(Fig. 1). 분리된 균주는 20% glycerol 용액에 혼입하여 -80℃에 보관하였다.
DNA 추출 및 염기서열 분석
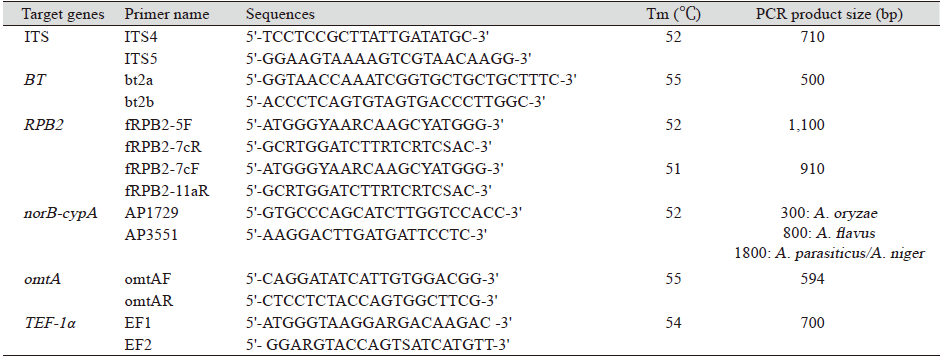
분리된 균주는 PDA 배지에 25℃에서 3–5일간 배양 후, 지 등[12]의 방법을 이용하여 균사체로부터 genomic DNA를 추출하여 -20℃에 보관하면서 실험에 사용하였다. 균 동정 및 유전적 다양성 분석을 위해 internal transcribed spacer region (ITS) rDNA, beta-tubulin (BT), RNA polymerase II subunit (RPB2), translation elongation factor 1α (TEF-1α) 유전자를 이용하였다(Table 1). Aspergillus 및 Penicillium 속 균주들은 보다 정확한 동정을 위해 ITS와 BT 유전자들을, Fusarium 속 균주는 translation elongation factor 1α (TEF-1α) 유전자의 염기서열을 분석하여 동정하였다. Aspergillus 속 균주 중 33개의 A. flavus와 A. oryzae 균주들은 아플라톡신 생합성 유전자 norB-cypA와 omtA를 추가로 분석하여 종을 구분하였다[13,14]. A. flavus와 A. oryzae는 ITS, BT 유전자는 물론 AFs 생합성 유전자 또한 높은 염기서열 상동성을 가져 구분하기 어렵기 때문에, aflT, norA 그리고 verA 세 유전자에 대한 결손 여부, omtA 유전자의 다형성 비교를 통해 A. flavus, A. oryzae 종을 동정하였다.
균 동정 및 유전적 다양성을 조사하기 위해 ITS, BT, RPB2 그리고 omtA 유전자의 염기서열을 DNASTAR (Lasergene 8, Madison, WI, USA)를 이용하여 교정하고, ClustalW 소프트웨어[15]를 이용하여 정렬하였다. ITS, BT, RPB2 유전자들의 염기서열들을 결합한 multi-locus 염기서열(2,700 bp)과, omtA 유전자 단일 염기서열(510 bp)을 MEGA-X 프로그램[16]을 사용하여 각각 maximum likelihood (ML) 법으로 계통수를 작성하였다.
땅콩의 곰팡이독소 분석
땅콩에서 자연 발생하는 아플라톡신의 정량분석은 식품공전[17]과 Trucksess 등의[18] 시험법을 변형하여 수행하였다. 땅콩의 겉껍질을 제거, 알땅콩만 수집하여 믹서로 분쇄 후 65℃에서 18시간 건조하여 실험에 사용하였다. 아플라톡신(B1, B2, G1 및 G2) 분석을 위해 균질화된 시료 3 g에 NaCl 1 g과 추출용액(MeOH: 0.5% NaHCO3=7:3, v/v) 15 mL을 가하여 300 rpm에서 1시간동안 진탕 추출하였다. 추출액은 3600 rpm에서 10분간 원심분리하여 상등액 7 mL을 취해 phosphate-buffered saline (PBS) 28 mL과 혼합하여 면역친화성컬럼(AflaTest WB, Vicam, MA, USA)에 주입하였다. 추출액이 여과된 컬럼은 0.1× PBS 3 mL과 증류수 5 mL로 순차적으로 세척하여 MeOH 2 mL로 용출 후 건조하였다. 용출액의 유도체화를 위해 트리플루오로초산(trifluoroacetic acid) 0.2 mL을 가하여 암실에서 15분간 방치하고, 20% ACN 0.8 mL을 가하여 혼합 후 0.2 μm 필터로 여과하였다. 시액들은 UPLC (Waters Acquity UPLC® H Class, Waters, Singapore)에 Xselect CSH C18 (2.5 μm, 2.1×100 mm, Waters, Dublin, Ireland) 컬럼을 장착하여 분석하였고, 유속 0.2 mL/min, 시료 주입량 10 μL, 분석시간 10 min, 이동상 DW:ACN:MeOH (65:15:20), 형광검출기(fluorescence detector, FLD) 파장 Ex=362 nm와 Em=455 nm 조건에서 분석하였다.
Aspergillus
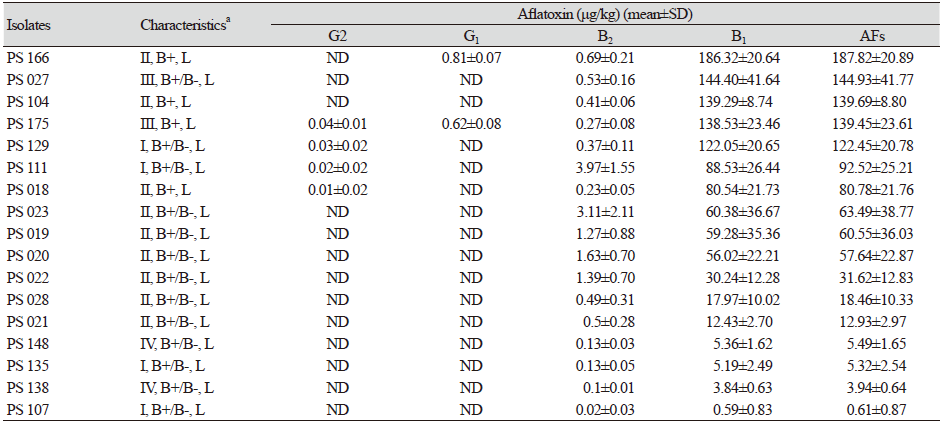
분리된 Aspergillus 속 균주들 중에서 아플라톡신 생성 가능성이 있는 균주들을 선발하기 위해 PCR을 실시하였다. 아플라톡신은 생합성 유전자 중 norB-cypA 유전자 부위를 증폭하기 위해 AP1729와 AP3551 프라이머를 이용하였다. 선발된 17점의 아플라톡신 생성 가능 균주들은 A. flavus-A. parasiticus agar (AFPA, Oxoid) 배지에 배양하여 배지 뒷면의 orange-yellow 발색 양성을 확인한 후[19], 독소생성능 실험에 이용하였다. 균주들은 20 mL로 정량하여 제작된 PDA 배지에 접종하여 25℃ 항온기, 암조건에서 배양하였다. 배양 7일 후 균사를 포함하는 배지 3 g을 취하여 땅콩의 곰팡이독소 분석법과 동일하게 분석하였다.
결과 및 고찰
저장 땅콩의 곰팡이 및 곰팡이독소 오염
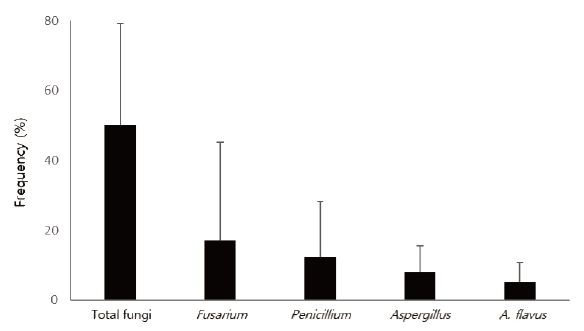
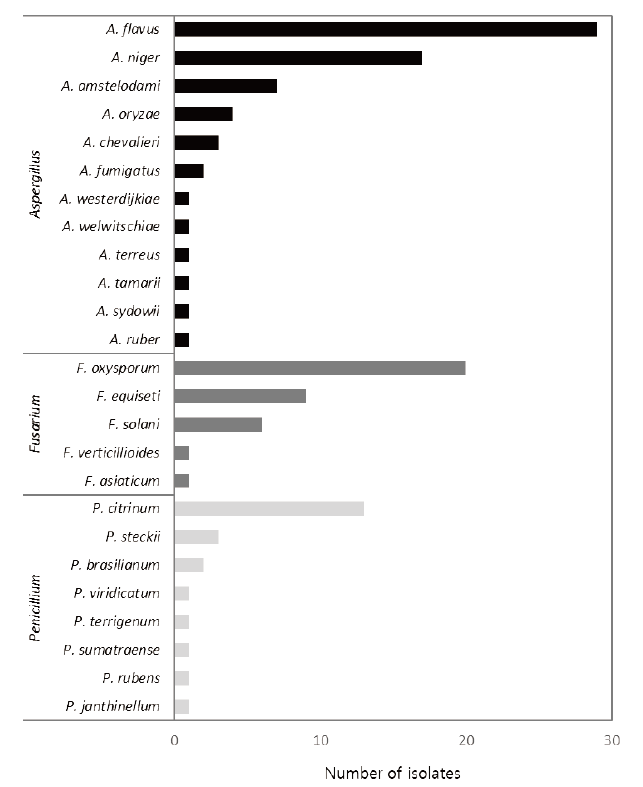
겉껍질이 있는 상태로 저장창고에서 보관되고 있는 땅콩은 Fusarium (17.2±28.0%), Penicillium (12.4±28.0%) 그리고 Aspergillus (8.0±7.6%) 속 곰팡이에 주로 오염되어 있었으며(Fig. 2), 그 외 Talaromyces, Rhizopus, Rhizoctonia, Trichocladium, Clonostachys, Mucor, Chaetomium, Trametes, Epicoccum, Humicola 속 곰팡이도 검출되었다. 땅콩의 주요 독성곰팡이로 알려진 Aspergillus 속을 주로 분리하였으며, 또한 다양한 작물들에서 독성곰팡이로 잘 알려진 F. graminearum species complex, F. fujikuroi species complex, Penicillium 속 곰팡이도 분리하였다. 분리된 총 145점의 균주를 동정한 결과, Aspergillus (68점), Fusarium (37점), Penicillium (23점), Talaromyces (11점) 속으로 나타났다. Aspergillus 속에서는 아플라톡신을 생성하는 것으로 알려진 A. flavus (29점, 42.6%)가 가장 많았으며, 다음으로 A. niger (25.0%), A. amstelodami (10.3%), A. oryzae (5.9%), A. chevalieri (4.4%), A. fumigatus (2.9%), 순이었다(Fig. 3). Penicillium 속은 P. citrinum (56.5%)이, Fusarium 속은 F. oxysporum (54.1%)이 가장 많이 동정되었다. 브라질에서 저장 땅콩은 Fusarium (25.8%), Aspergillus (21.8%)에 주로 오염되었고, 케냐에서는 Rhizopus, Fusarium, Aspergillus가 각각 80, 45, 24% 오염된 것으로 보고되었으며[20,21], 남아프리카에서 유통 중인 땅콩도 Aspergillus가 40% 이상의 발생률을 나타내었다[22]. 중국의 저장 땅콩에서는 지역간 차이는 있으나, Rhizopus, Emericella, Clonostachys, Penicillium, Eurotium, Aspergillus 순으로 우점하는 것으로 나타났고, Aspergillus의 오염률은 0.53–25.75%였다[23].
고창지역 저장 땅콩에서 A. flavus는 6–10%의 오염률을 나타내었으나 아플라톡신은 전혀 검출되지 않았다. 브라질의 A. flavus 오염률 및 아플라톡신 B1 오염농도는 각각 3.0–31.0%와 7.0–116 μg/kg이었고, 중국의 경우 0.12–6.39%와 1.02–4.03 μg/kg인 것으로 보고되었다[21,23]. 브라질의 경우 저장 온도는 19–27℃, 상대습도는 59–87% 이었는데, 시료의 33%가 아플라톡신에 오염되었고 대부분 기준치 이상의 농도였다. 중국의 경우 저장 초기(0–6개월)에는 온도가 -1–15℃ 였으나 저장 후기(7–12개월)에는 30℃까지 증가하였고, 상대습도는 48–83%까지 증가하였다. 이에 온도가 낮은 저장 초기(0–6개월)에는 Aspergillus 속 곰팡이 오염률이 낮고 아플라톡신 B1은 검출되지 않았으나 저장 후기 온도가 상승하면서 Aspergillus 속 곰팡이와 아플라톡신 B1의 오염률이 증가하였다. A. flavus의 성장과 그에 따른 아플라톡신의 생성은 여러가지 요인에 의해 영향을 받지만 특히 온도의 영향이 큰 것으로 알려져 있다. Schindler 등[24]에 따르면 A. flavus는 18℃ 이하 또는 35℃ 이상의 온도에서는 아플라톡신을 생성하지 않는 것으로 보고하였다. 고창 지역에서는 땅콩이 10월경에 수확되어 다음해 4월까지 상온으로 저장되는데 평균온도 7℃, 상대습도 68% 내외로 유지된다. 고창지역 땅콩에서 상대적인 A. flavus의 오염률이 낮고 아플라톡신이 검출되지 않은 것은 저장기간 동안의 온도와 습도가 낮아 A. flavus의 성장과 그에 따른 아플라톡신의 생성에 적합하지 않기 때문으로 판단된다.
A. flavus의 유전적 다양성
저장 땅콩에서 분리한 17점의 A. flavus 균주는 multi-locus sequence analysis 결과 2개의 유전적 그룹으로 나뉘었다(Fig. 4). PS 138과 PS 148 두 균주가 단일 그룹(Cluster IV)을 형성하였으며, 다른 그룹은 3개의 서브 그룹을 형성하였다. 3개의 서브 그룹에는 A. flavus NRRL 3357 균주와 근연관계를 가지는 균주 2점(Cluster III), A. flavus A9 균주와 근연관계를 가지는 균주 4점(Cluster I), 그 외 9점의 균주(Cluster II)가 각각 속하였다. 그룹간의 염기서열 유사도는 99.9%로 6 bp의 차이를 나타내었다. Chang 등[14]은 아플라톡신 합성 유전자 omtA의 다형성, norB-cypA의 결손 정보를 이용한 아플라톡신 독소 생성능, 그리고 morphotypes (S-와 L-type)의 비교를 통해 독성 및 비독성 A. flavus와 비독성 A. oryzae을 구분할 수 있음을 보고한 바 있다. 이에 본 연구에서 아플라톡신 생성 관련 omtA 유전자의 계통수 분석 결과, 분리된 A. flavus 균주들은 모두 L-type의 형태학적 특징을 가지며 아플라톡신을 생성할 수 있는 2개의 유전적 그룹으로 구분되었다(Fig. 5). 균주 PS 018, PS 104, PS 166, PS 175는 L-type, B+ 특성을 가지는 A. flavus NRRL 3357 및 A. parasiticus NRRL 3357 균주와 상동성을 보였고, 그 외 13점의 균주는 L-type, B+/B- 특성을 가지는 A. flavus A9과 A. flavus NPL VA2-9 균주와 100% 상동성을 보였다. A. flavus는 균핵의 형성 타입에 따라 2개의 morphotypes으로 구분되며, 일반적으로 L-type이 S-type보다 흔하게 발견된다. S-type의 A. flavus는 L-type에 비해 작은 크기(400 μm 이하)의 균핵을 만들며 3.5-4배의 아플라톡신을 만드는 것으로 보고되었다[25].
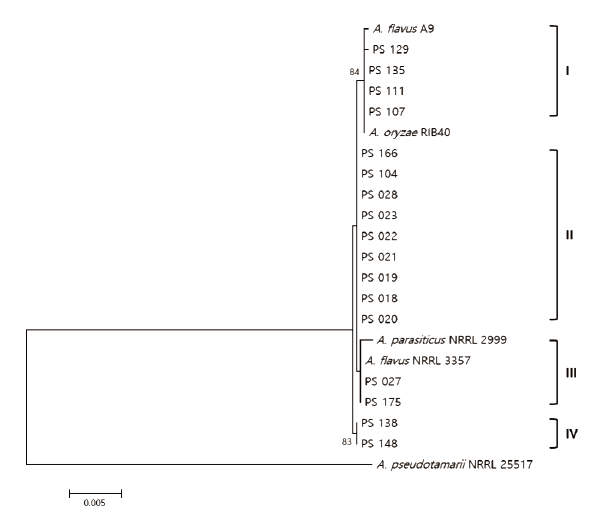
Fig. 4. Maximum-likelihood phylogenetic tree based on combined sequences of internal transcribed spacer (ITS) region , beta-tubulin (BT ), RNA polymerase II subunit (RPB2) genes of A. flavus isolates. The numbers above the nodes represent bootstrap values of >70% out of 1,000 bootstrap replication. A. pseudotamarii NRRL 25517 was used as an outgroup.
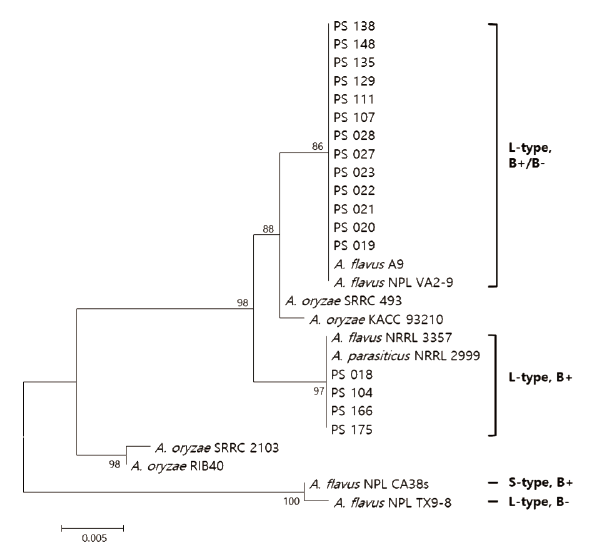
Fig. 5. Maximum-likelihood tree based on sequences of omtA gene of Aspergillus flavus isolates and related species. The numbers above the nodes represent bootstrap values of >70% out of 1,000 bootstrap replication. L- and S-type, morphotype; B+ and B-, aflatoxin B producing and non- producing strains.
A. flavus의 아플라톡신 생성능
분리된 A. flavus 균주는 시료 채집시기, 저장고, 저장백(bag)에 따라 1–2 균주씩 총 17점의 균주를 선발하였다. 선발 균주의 독소생성능을 평가한 결과, 모든 균주가 아플라톡신 독소를 생성하는 것을 확인하였다(Table 2). 총 아플라톡신 생성 농도는 최대 187.82±20.89 μg/kg, 최저 0.61±0.87 μg/kg, 평균 68.69±62.16 μg/kg 이었고, 이 중 4점의 균주는 100 μg/kg 이상의 아플라톡신 독소를 생성하였다. 이 균주들은 모두 아플라톡신 B1과 B2를 생성하였고, 그 중 1점은 G1, 3점은 G2, 1점(PS 175)은 G1과 G2를 모두 생성하였다. A. flavus 균주의 유전적 다양성과 아플라톡신 생성능의 관계를 살펴본 결과, Cluster III 균주들이 비교적 높은 농도의 아플라톡신을 생성하는 반면 Cluster IV의 균주들은 낮은 농도의 아플라톡신을 생성하였다.
본 연구의 고창지역 저장 땅콩에서 아플라톡신은 검출되지 않았으며, A. flavus 오염률이 5–10%로 낮았다. 그러나 분리된 A. flavus 균주의 대부분이 비교적 높은 농도의 아플라톡신을 생성할 수 있는 것으로 나타나 국내 주요 땅콩 생산지에 대한 추가적인 모니터링이 필요할 것으로 판단된다. 이 연구는 국내 저장 땅콩에서 A. flavus 오염률 및 독소생성능에 대한 첫 보고이다.
적요
땅콩의 독성곰팡이 및 곰팡이독소 오염현황을 조사하기 위해 고창 지역의 저장고에서 땅콩을 수집하였다. 수집된 땅콩은 Fusarium (17.2±28.0%), Penicillium (12.4±28.0%), Aspergillus (8.0±7.6%) 속 곰팡이로 주로 오염되어 있었으며, 그 외에 Talaromyces, Rhizopus, Rhizoctonia, Trichocladium, Clonostachys, Mucor, Chaetomium, Trametes, Epicoccum, Humicola 속 곰팡이가 검출되었다. 땅콩시료에서 아플라톡신은 검출되지 않았으나, 29점의 A. flavus 균주가 분리, 동정되었다. 그 중 17점의 균주를 선발하여 potato dextrose agar (PDA) 배지 상에서 아플라톡신 생성능을 분석한 결과 0.61–187.82 μg/kg의 농도범위에서 모두 아플라톡신을 생성하였다. 이들 17 균주는 아플라톡신 B1, B2도 생성하였으며, 일부(5 균주)는 G1 또는 G2를 생성하였다. 이 연구는 국내 저장 땅콩에서 A. flavus 오염률 및 독소생성능에 대한 첫 보고이다.