서론
송이는 상업적으로 중요한 버섯이지만, 현재까지 자연채취에 의존하고 있다. 송이재배가 성공한 예는 1983년 히로시마임업시험장[1]과 2010년 국립산림과학원에서 송이감염묘를 이용한 방법이 유일하다[2]. 일본의 경우는 아쉽게도 지속적인 송이발생이 없었지만, 한국의 경우는 지속적인 송이발생이 이루어지고 있다[2].
우리나라에서 송이감염묘 연구는 1980년대부터 시작되었다[3]. 이 당시 송이감염묘를 육성하여 이식하였으나, 버섯발생까지는 성공하지 못하였다. 동일한 연구가 2000년부터 임업연구원(현; 국립산림과학원)에서 다시 시작되었고, 송이감염묘 육성의 시기별 차이, 이식한 감염묘에서 송이균의 생존, 색대를 이용한 송이균 확인[4], 송이균환 생장 패턴, 송이균 생장의 추적 조사 및 버섯 발생 조사가 이루어졌다[2,5]. 또한 송이감염묘에서 소나무의 크기가 송이균의 생존에 중요하다는 것이 밝혀졌다[6].
위의 연구에서는 송이감염묘 육성 및 이식과 송이균 생장과 버섯 발생까지 추적 조사하였으나, 분자학적인 방법을 적용한 연구가 이루어지지 않았다. 그 결과 이식한 송이감염묘로부터 발생한 송이가 감염묘에서 유래한 것인지에 대한 의구심을 제기해 왔다[7,8].
송이감염묘를 이식 및 버섯발생을 모니터링 하는 홍천 송이시험지에서 2020년 21개의 송이가 발생하였다. 본 연구는 송이감염묘 이식지역에서 발생한 송이와 송이감염묘를 육성한 지역에서 발생한 송이가 동일성 유무를 알아보고자 microsatellite 분자마커를 이용하여 분석하였으며 그 결과를 보고하고자 한다.
재료 및 방법
송이감염묘의 버섯 시료
송이감염묘용 소나무는 2000년 4월과 11월에 강원도 양양군 현북면 어성전리 소재 송이산에 608본[4]을 식재하였고, 2002년 3월에 홍천군 동면 노천리 소재 송이산에 246본을 식재하였다. 홍천 송이 시험지에는 2001-2003년에 양양군 송이산에서 만들어진 송이감염묘 총 99본과 2004년에 홍천군 송이산에서 만들어진 송이감염묘 총 51본을 옮겨심었다. 송이감염묘는 송이균 생존, 생장과 버섯 발생을 추적 조사하였다[2,4,5].
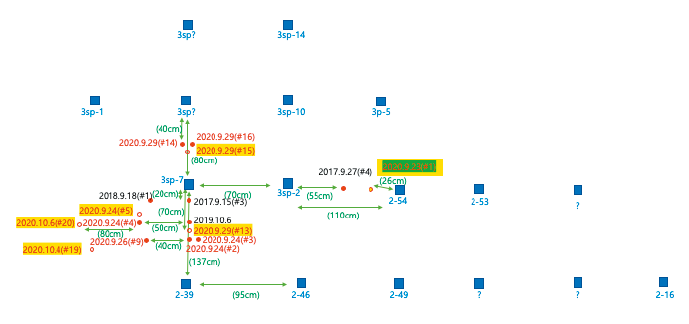
양양군 송이산은 현재 관리하고 있지 않아, 버섯 시료를 확보하지 못하였다. 홍천의 송이 감염묘 육성 지역도 현재 관리되지 않아 대부분의 송이균환이 쇠퇴하거나 소멸되어 가고 있다. 홍천의 송이감염묘 육성 지역은 5개 조사구(1번과 2번 20 m×20 m, 3번 20 m×40 m, 4번과 5번 20 m×30 m)를 선정하였고, 2번과 3번 조사구 사이에 1개 송이균환을 3sp로 명명하였다. 2002년 송이 감염묘 육성은 5개 조사구와 3sp 균환에서 이루어졌다. 홍천 송이감염묘 육성지역의 송이 시료는 2020년 2번 조사구와 3sp 균환에서 확보하였다(Table 1). 이식한 송이감염묘로부터 발생한 송이는 2020년 21개였고(Fig. 1), 이중 일부를 분석에 이용하였다(Table 1).
Microsatellite marker를 이용한 유전형 분석
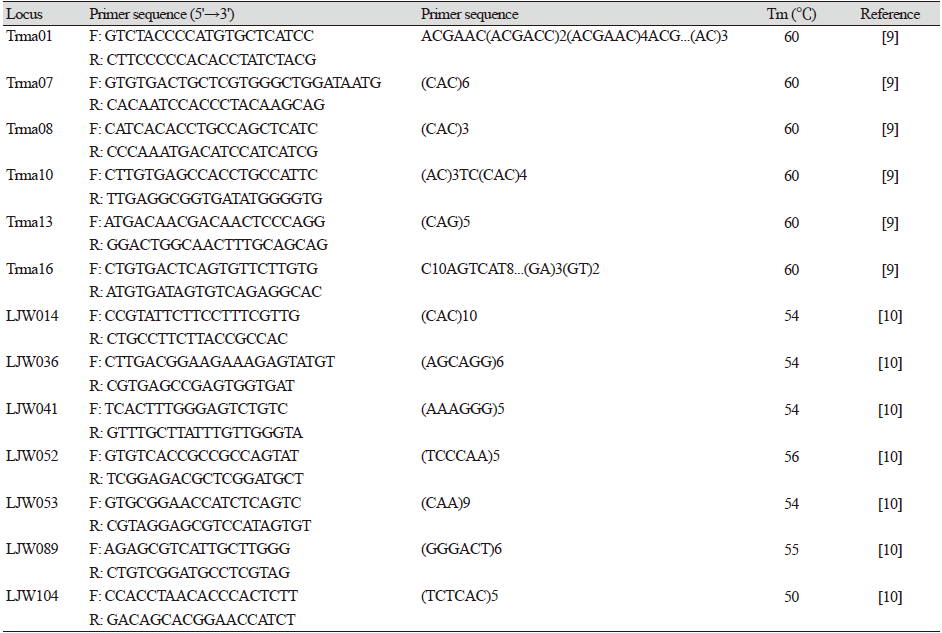
유전형 분석을 위한 DNA는 자실체로부터 추출하였다. DNA 추출은 ExgeneTM Plant SV(Cat. No. 117-152, GeneAll Biotechnology, Seoul, Korea) DNA 추출 키트를 이용하였다. 자실체는 DNA 추출 전 동결건조하여 -20℃ 냉동고에 보관하였으며, FastPrep-24TM (MP Biomedicals, California, USA) 장비와 스텐리스 비드를 이용하여 자실체를 마쇄하여 DNA를 추출하였다. DNA 추출 과정은 DNA 키트 제조사의 매뉴얼을 참조하였다. 추출된 DNA는 NanoDrop 2000 (Thermo Fisher Scientific, Massachusetts, USA)을 이용하여 농도와 순도를 측정하였다. Microsatellite markers는 이미 송이 분석을 위한 마커로 개발된 것을 이용하였다[9,10]. 이중 국립산림과학원에 보존된 송이 수집 균주에 대하여 다형성을 나타내는 13개의 microsatellite 마커를 선발하여 본 연구에 적용하였다(Table 2). PCR 분석은 총 20 μL 용액에 20 ng DNA, 1× PCR 반응 완충용액, 0.2 mM dNTP, 0.2 μM 순방향 및 역방향 프라이머, 0.5 U Taq DNA polymerase (BIOFACT, Daejeon, Korea)를 첨가하였다. PCR 반응은 Lian 등[9]과 Li 등[10]의 조건을 참조하였다. PCR 증폭 산물은 ABI 3730xl DNA Analyzer (Thermo Fisher Scientific, Massachusetts, USA) 장비를 이용하여 전기영동 하였으며, Gene Mapper v5.0 (Thermo Fisher Scientific, Massachusetts, USA) 소프트웨어를 이용하여 각 시료의 유전형을 결정하였다. GenAlEx 6.5 프로그램의 Multilocus Match 방법을 이용하여 각 시료의 유전형 간 동일성을 비교하였으며, 각 시료 간 유전적 유사도는 Codom-Genotype distance 방법을 이용하여 유전적 거리를 산출하고 주좌표 분석을 실시하여 시각화 하였다[11].
결과 및 고찰
13개의 Microsatellite 마커 분석결과, 분석된 15개의 자실체에서 총 31의 대립유전자가 관찰되었다. LJW036 마커에서 가장 많은 4개의 대립유전자가 관찰되었으며, Trma01, Trma08 및 Trma13 마커에서 1개의 대립유전자가 관찰되었다. 각각의 자실체에 대하여 13개 마커에서 관찰된 유전형의 조합을 비교한 결과, 일부 자실체간 동일한 유전형이 나타났으며, 총 6개의 유전형이 확인되었다. 홍천 송이감염묘 육성지역 3sp에서 발생한 2개의(시료번호 3sp-1, 3sp-2번) 자실체와 송이감염묘 이식지역에서 발생한 5개의(시료번호 5, 13, 15, 19, 20번) 자실체가 동일한 유전형을 나타내었다. 즉, 송이균환 3sp에서 발생한 송이와 그곳에서 만들어진 감염묘를 2004년에 옮겨 심어 2020년 발생한 송이가 일치하는 것으로 나타났다(Table 1; Fig 2 and 3). 또한 2번조사구에 3개 균환이 있었는데, 그중 한 곳에서 발생한 송이 자실체(시료번호 2-1번)와 그곳에서 만들어진 송이감염묘를 2004년에 옮겨 심은 곳에서 2020년 발생한 송이 자실체(시료번호 1번)의 유전형이 동일하게 나타나 감염묘를 통해 동일한 유전형을 갖는 송이가 발생한 것을 알 수 있었다(Table 1; Fig 2 and 4). 결국 송이감염묘가 송이균을 옮겨주어 버섯발생까지 이어졌다고 할 수 있다. 그리고 송이감염묘 육성지역 2번 조사구의 3개 송이가 다르게 분석된 것은 서로 다른 균환임을 가리킨다. 일본 연구자들이 한국의 송이감염묘 연구에 대해 분자학적 뒷받침이 없는 것에 대해 언급하고 있다[7,8]. 이번 결과는 한국의 송이감염묘 연구를 통해 발생한 송이가 송이감염묘로부터 기원했다는 명확한 답을 줄 수 있는 결과이다. 한편, 분석된 송이 자실체간 유전적 거리 분석결과에서 양양지역에서 발생한 2개의 자실체(시료번호 17, 21번)와 홍천의 3sp 지역에서 유래된 자실체(3sp-1, -2번 및 5, 13, 15, 19, 20번)간 유전적 거리가 가장 멀었으며, 기타 홍천지역 송이 사이에도 유전형의 차이에 따라 유전적 거리가 상이한 것으로 나타났다(Table 3). 이러한 결과는 양양에서 옮겨온 송이감염묘에서 발생한 송이는 홍천 것과 유전적으로 상당부분 이질적인 것으로 추정할 수 있으며, 홍천의 감염묘에서 발생한 송이도 모두 동일하지 않아 송이균환에 따라 다른 유전자풀을 갖고 있고 그로부터 만들어진 송이감염묘 임을 알 수 있다(Table 1; Fig 2). 한편, 양양의 송이감염묘 육성지역에서 송이 버섯시료를 확보할 수 없어 본 연구에서는 비교할 수 없었다.
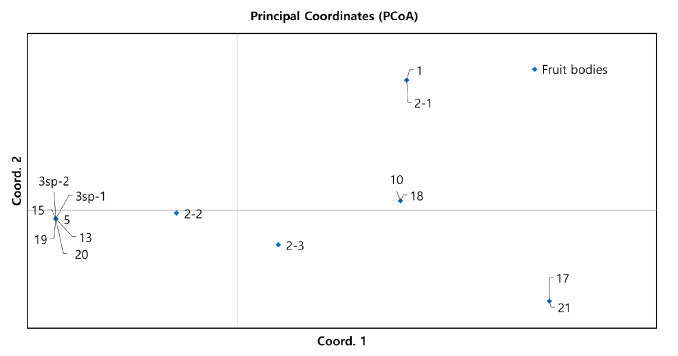
Fig. 2. Principal coordinates analysis according to genetic distance between the fruiting body of existing production area and the fruiting body of transplanted area of matsutake-infected pine trees. The fruiting bodies with the same genotype are located at the same point. Number indicates the number of tested fruiting body used in this study (Table 1)
Murata 등[12]은 같은 송이균환에서도 유전적 변이를 갖고 있어 매우 다양한 송이균환이 존재할 수 있음을 제시하였다. Kurokochi 등[13]은 10개 송이 개체군 중 단지 1개 개체군에서 많은 대립유전좌가 관찰되어 개체군의 변이가 크지 않다고 하였다. 본 연구에서는 낱개 송이의 동질성 유무만을 분석하다 보니 한 지역적 개체군의 변이를 언급하기는 어려웠으나, 동일지역의 다른 송이균환이 사용한 microsatellite 마커에 의해 구별성을 갖는 것으로 보아 유전적 다양성이 높게 관측될 수 있음을 예상할 수 있었다. 그리고 우리가 적용한 microsatellite 마커는 송이균을 구분할 수 있는 매우 분별력이 좋은 마커로 생각된다[9,10]. 아울러 송이감염묘 시험지가 지속적으로 모니터링과 산림관리를 통해 유지되었기에 송이발생의 결과를 얻을 수 있었다고 판단된다[2].
한편, 홍천 송이감염묘 시험지는 2009년 송이균이 23곳이 생존한 것이 확인되었고[5], 버섯 발생은 2010년 1개, 2017년 5개, 2018년 1개, 2019년 1개, 2020년 21개 이루어졌지만, 분자학적 방법을 이용한 동일성 유무를 판별하지는 않았다. 따라서 본 연구를 통해 2020년 발생한 송이가 동일한 송이균환에서 유래한 것인지를 파악하는 계기가 되었고, 생존한 송이균환과 발생한 버섯과의 관계 확인에 도움이 되었다. 2010년에 발생한 곳의 송이균환은 현재 매우 미약한 상태이며, 지속적인 송이발생이 이루어지지 않고 있다. 송이 발생은 외부 환경 요인에 좌우되기에[14] 기대하는 수준의 지속적 송이 발생은 이루어지지 않고 있다.
적요
송이는 상업적으로 중요한 야생 식용 버섯이다. 현재까지 송이 재배는 송이감염묘를 이용한 것이 유일한 방법이다. 2020년 송이감염묘로부터 21개의 송이가 홍천 송이시험지에서 발생하였다. 송이감염묘 생산지역의 송이와 이식한 송이감염묘로부터 발생한 송이는 모니터링을 통해 확인하였고, 13개의 microsatellite 마커를 이용하여 분석한 결과 동일한 것이 확인되었다. 송이감염묘는 송이균환 형성과 송이 발생에 성공하였고, 송이발생은 2010년과 2017년부터 2020년까지 발생하였다. 결론적으로 송이 버섯발생은 송이감염묘의 송이균에서 기원한 것이다.