서론
버섯 바이러스는 양송이 재배농가에서 최초로 보고되었으며, 버섯이 바이러스에 감염되면 자실체의 기형 현상이 발생하고 균사 생장이 불량해지는 것으로 보고되었다 [1,2]. 예컨대 Flammulina velutipes browning virus (FvBV)는 팽이버섯 자실체의 갈변현상을 유발하여 버섯 품질에 영향을 줄 수 있음이 보고되었다 [3]. 특히 양송이에서 바이러스에 의한 La France disease로 인해 버섯의 생산에 막대한 피해를 입게 되면서, 버섯 바이러스에 대한 연구가 활발하게 이루어지게 되었다 [4]. 초기에는 버섯에서 새로운 바이러스를 검출하고 동정하는 연구가 주를 이루었으며, 그 과정에서 일부 버섯은 바이러스 입자가 포함된 균주라도 재배 환경에 따라 문제가 되지 않을 수 있음이 보고되었다[5,6]. 바이러스 입자가 발견되었지만 병징이 나타나지 않으면 바이러스 감염의 위험성을 간과할 수 있지만, 특정 환경 조건 또는 특정 돌연변이를 가진 균주에서 바이러스 감염이 여전히 문제가 될 수 있음이 보고되었다 [7]. 이처럼 바이러스 감염이 버섯의 품질과 생산성에 상당한 영향을 미칠 수 있기 때문에 바이러스에 감염되지 않은 균주를 선별하는 것은 매우 중요하다.
한편, 버섯에 감염된 바이러스를 검출 및 동정하는 연구와 병행하여 버섯 바이러스를 제거시키는 연구도 주목을 받고 있다. 기존의 연구에서 AIDS 치료제이자, 균사 생장에 영향을 미치는 것으로 알려진 adenosine 3’,5’-cyclic monophosphate (cAMP)가 첨가된 배지에서 균사를 계대 배양하는 방법과 균사체 단편화 방법을 이용하여 바이러스를 제거하는 방법이 보고되어 있다 [8-10]. 최근에는 표고에 항바이러스제인 ribavirin을 처리하여 바이러스의 제거를 시도한 결과, Lentinula edodes mycovirus HKB (LeV-HKB) 제거는 성공하였으나 Lentinula edodes partivirus 1 (LePV1) 제거는 실패함을 통해, ribavirin이 특정 바이러스에 대한 제한적인 제거에 유용할 수 있음이 보고되었다 [11]. 이러한 바이러스 제거 방법 개발은 바이러스 감염 균주로부터 바이러스만 제거되고 유전적으로는 동일한(isogenic) 균주를 얻게 함으로써, 버섯과 바이러스 상호작용에 대한 연구에 필수적이다. 하지만 현재까지 mycovirus는 식물 및 동물 바이러스와 달리 연구 기술적 측면에서 인공접종과 형질전환의 방법이 부족하며, 버섯에 감염된 바이러스를 제거하는 방법도 한정된 종류의 버섯과 버섯 바이러스에 대해서만 확인되어서, 버섯과 바이러스의 상호작용을 이해하는 데 아직 많은 어려움이 있는 상황이다 [12,13].
표고는 전국에서 널리 재배되고 있고 생산량과 경제적 가치 측면에서 중요한 상업용 버섯 중의 하나이다 [14,15]. 팽이버섯 및 느타리에 비해 표고는 재배 기간이 비교적 길고 동일한 배지에서 자실체를 5회 이상 수확이 가능하다. 특히 농장에서 표고를 재배할 때 종종 자연 환경에 노출되어 있기 때문에 바이러스 감염에 취약하다는 특징이 있다 [16]. 실제로 본 연구팀의 선행 연구 결과, 한국의 야생 표고 112균주 가운데 약 90%가 바이러스에 감염된 것으로 나타났다 [17]. 게다가 국내에서 표고의 실내 재배가 계속 증가함에 따라 배지 표면의 불완전한 갈변, 균사체 이상성장, 자실체 기형 등의 다양한 이상 증상이 관찰되고 있어 바이러스 감염에 대한 우려도 높아지고 있다 [18].
지금까지 여러 종류의 바이러스가 표고에서 발견되었는데 그 중에서 LeV-HKB는 이중 가닥 RNA 바이러스의 일종으로, 일본에서 전체 유전체 염기서열 분석을 완성한 최초의 표고 바이러스이며, 국내 야생 표고에서 가장 높은 감염률을 보이는 것으로 알려져 있다 [17,18]. Lentinula edodes negative-strand RNA virus 1 (LeNSRV1)은 단일 가닥 RNA를 게놈으로 가지며, 담자균을 감염시키는 mymonavirus로는 첫 번째로 알려졌으며, 알려진 mymonavirus들 가운데 가장 큰 게놈을 가지고 있는 것으로 보고되었다 [19].
한편, 버섯 바이러스는 균사융합(anastomosis)에 의해 세포내용물이 이동하는 과정에서 전이(수평 감염, horizontal transmission)되거나, 포자 생성 과정에서 후대로 전이(수직 감염, vertical transmission)되는 것으로 알려져 있다 [20,21]. 버섯 신품종 육성 과정에서 우량 품종의 담자포자 유래 단핵균사를 활용한 단핵균사-단핵균사 교잡(mono-mono cross) 혹은 이핵균사-단핵균사 교잡(di-mono cross)이 널리 쓰이는 것을 고려할 때, 우량 품종에서의 버섯 바이러스 수직 감염에 대한 연구는 버섯 바이러스의 수직 감염 기작을 이해하기 위한 학문적 측면 뿐 아니라 신품종 버섯의 바이러스 감염 관리를 위해서도 필요할 것이다.
이에 본 연구에서는 표고 품종 산백향(NIFoS 2778)과 태향고(NIFoS 4317)의 바이러스 감염 여부를 확인하였고, 이들의 담자포자 유래 단핵균사들이 바이러스에 감염되었는지 확인함으로써 수직감염에 대한 조사를 수행하였다. 나아가 각각의 담자포자 유래 단핵균사들에 대한 생장속도를 확인하였다. 본 연구는 표고의 무바이러스 종균의 선발 및 바이러스에 감염되어 있지 않은 포자를 활용한 교배를 통해 새로운 무바이러스 종균을 개발하는 데 도움이 될 것으로 기대된다.
재료 및 방법
균주 및 배양조건
실험에 사용한 표고 균주는 국립산림과학원 균주보존실에 potato dextrose agar (PDA)배지에 4℃로 보존되어 있는 산백향(NIFoS 2778), 태향고(NIFoS 4317) 균주를 사용하였다. 이 균주들은 PDA 평판배지에 25℃에서 배양하여 접종원으로 사용하였다.
담자포자 분리
페트리 디쉬에 버섯 자실체를 올려놓고 2-3시간 두어 포자가 떨어지면 멸균된 증류수를 넣고 순차 희석 후 PDA배지에 50 μL 도말하여 25℃에서 5-7일간 배양하였다. 균사 콜로니가 생기면 하나씩 계대배양을 하여 단포자 균주를 얻고 광학 현미경으로 클램프의 발생유무를 확인하였다[22].
교배형 분석 및 단핵균주 선정
단핵균사의 교배형 분석은 선행보고의 조건을 따라 수행하였다 [23]. 균주별로 DNeay Plant Pro kit (Qiagen, CA, USA)를 사용하여 제조사에서 제공하는 표준 지침에 따라 gDNA를 분리한 뒤, 각각의 교배형을 선택적으로 증폭할 수 있는 프라이머로 PCR을 수행하였다. PCR 증폭 여부는 1.5% 아가로스 젤 전기영동으로 확인하였다. 교배형을 확인한 단핵균주 중에서, 각 품종 유래 단핵균주가 가질 수 있는 네 가지 교배형별로 20개의 균주를 무작위로 선정한 뒤, 이후 실험에 사용하였다.
RNA 추출 및 cDNA 합성
PDA 배지에서 성장한 균사체를 cell scraper (SPL, Seoul, Korea)로 모은 뒤, 막자와 막자사발, 액체질소를 이용해 균사를 가루 형태로 만들어서 RNeasy Mini Kit (Qiagen, CA, USA)를 이용하여 RNA를 추출하였다. RNA 정량은 Epoch Multi-volume Spectrophotometer (BioTek, VT, USA)를 사용하여 조사하였고 cDNA 합성은 Moloney murine leukemia virus (MMLV) 역전사효소(New England Biolabs, MA, USA)를 사용하여 수행하였다.
PCR 및 전기영동 확인
PCR은 합성된 cDNA를 주형으로 C1000 thermal cycler (Biorad, CA, USA)를 이용하여 95℃에서 5분 변성 후, 95℃에서 30초, 60℃에서 30초, 72℃에서 1분으로 25회 반복하고, 72℃에서 5분간 추가로 반응을 진행하였다. 이때 PCR 반응액은 AccuPower PCR premix를 사용하였으며, 프라이머는 바이러스의 RNA 의존성 RNA 중합효소(RdRp) 유전자에 대한 특이적 프라이머로 PCR을 수행하였다 [18]. 증폭된 PCR 산물은 1.5% 아가로스젤 전기영동으로 확인하였다.
균사 생장 조사
PDA 배지 1개당 25 mL씩 분주하여 동일한 조건의 배지를 준비하였다. 각 배지에 균사 플러그(직경 6 mm)를 접종한 후 25℃에서 14일 동안 배양하였다. 균사 생장률 측정을 위해 디지털 버니어캘리퍼스를 이용하여 각 균주의 균총의 크기(접종원의 직경 제외)를 측정하여 생장률을 비교하였다. 균주마다 3 반복 실험을 수행하였다.
통계분석
균사 생장속도에 대한 통계분석은 SPSS 프로그램(SPSS version18, SPSS IBM, NY, USA)을 이용하였다. 네 가지 교배형에 따른 생장속도 차이를 비교하기 위해서 일원배치 분산분석(one-way ANOVA)을 수행했으며, 감염 바이러스 종류에 따른 생장속도 차이를 비교하기 위해서 Student’s t-test를 수행하였다.
결과 및 고찰
표고 품종과 담자포자의 바이러스 검정
국내의 톱밥배지 재배용 표고 품종인 산백향(NIFoS 2778)과 태향고(NIFoS 4317)가 기존에 알려진 바이러스 중에서 2개의 mycoviruses (LeV-HKB 및 LeNSRV1)에 감염되어 있음을 확인하였다(Fig. 1). 이와 같은 여러 바이러스의 동시 감염은, 식물 및 곰팡이에서도 널리 보고되었으며, 표고에서는 중국과 일본 재배 품종들에서 보고된 바 있다 [9,16]. 또한 국내 야생 표고의 경우, 60% 이상이 2종에서 5종까지 다양한 바이러스에 다중감염 되었음이 알려졌다 [17]. 아직 국내외에서 표고 품종의 바이러스 감염 관리가 이루어지지 않고 있는 상황을 고려할 때, 이와 같은 다중감염이 여러 표고 품종에서 나타날 것으로 생각된다.
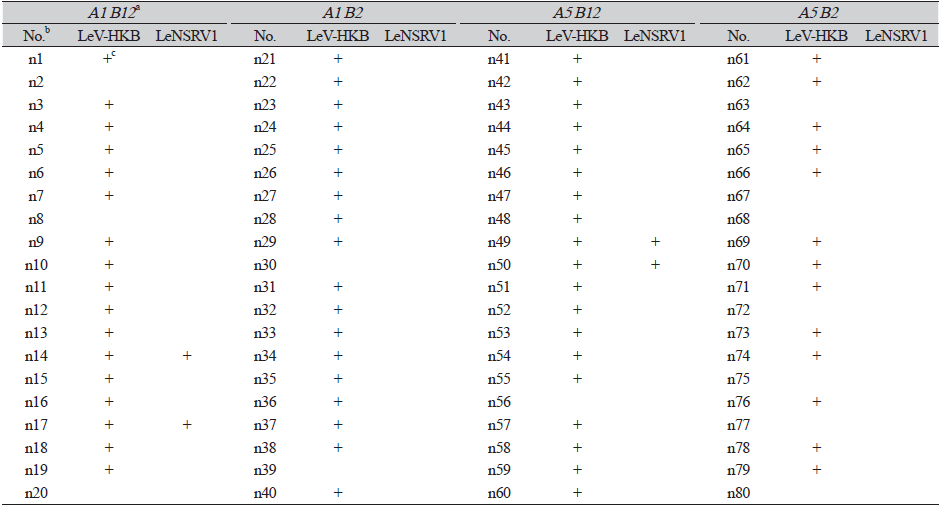
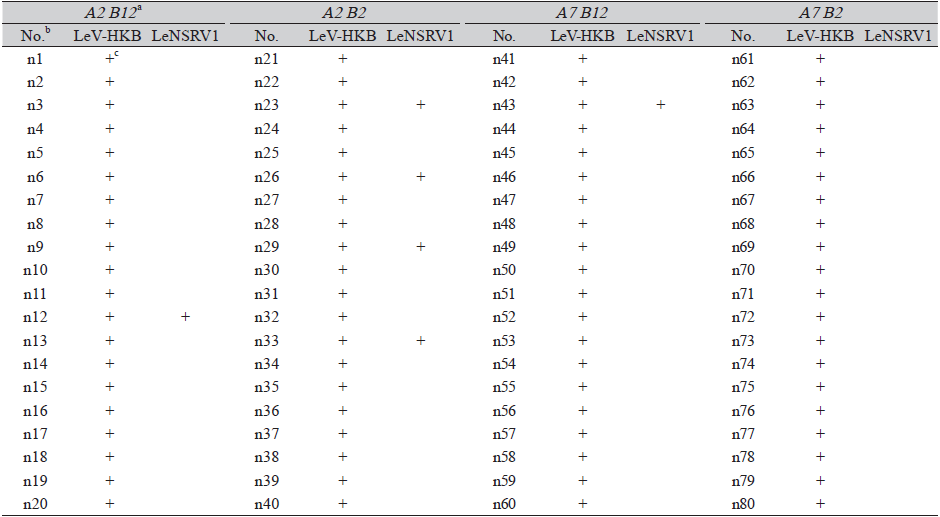
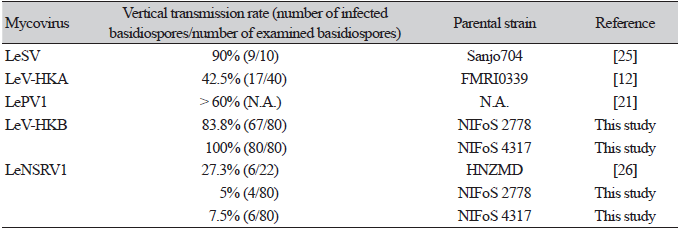
다음으로, 이 품종들에서 버섯 신품종 육성 과정의 재료가 될 수 있는 담자포자 유래 단핵균사들을 확보하여 후대로 전이되는 수직감염률을 조사하였다(Table 1 and 2). 그 결과, 산백향(NIFoS 2778)에서는 조사한 80균주 중 13균주의 무바이러스 단핵균사를 확보한 데 반해, 태향고(NIFoS 4317)에서는 무바이러스 단핵균사가 발견되지 않았다. 또한 LeV-HKB는 두 품종에서 평균 90% 이상의 감염률을 보인 데 반해(산백향 83.8%, 태향고 100%), LeNSRV1는 10% 이하의 낮은 감염률을 알 수 있었다(산백향 5%, 태향고 7.5%) (Fig. 2; Table 3). 두 가지 바이러스에 모두 감염된 단핵균사는 산백향 5%, 태향고 7.5%로 확인되었다. 기존의 연구결과들과 종합하여 봤을 때, 표고 품종에 따라 감염되는 바이러스 종류가 다르고, 바이러스 종류에 따라 수직 감염률도 다르게 나타났다. 특히 본 연구에서 확인한 LeV-HKB의 경우 보고된 표고 바이러스들 중 수직감염률이 LeSV와 더불어 높게 나타났다(Table 3). 일본에서 처음 발견된 LeV-HKB는 유전적 분석을 한 결과 외피단백질에 대한 서열정보가 확인되지 않았고 전자현미경 분석에서 초원심분리에 의한 바이러스 정제에 실패했는데 이것은 바이러스의 외피단백질이 없는 경우 바이러스의 정제에 취약하다는 것을 시사하고 있었다. 현미경 관찰을 위한 보다 안정적인 바이러스 정제를 위해 바이러스 현탁액을 여과한 후 바로 관찰하여 선형의 이중 가닥 RNA (double-stranded RNA)를 확인하였고, 다른 Totivirus와 밀접한 관련이 있는 mycovirus임을 확인하였다[24]. 바이러스의 구조는 바이러스가 숙주세포를 감염시키고 전파력을 증가시키기 위해 중요한데 외피단백질이 없는 LeV-HKB의 구조는 바이러스의 감염률과 증식률에 영향을 미칠 수 있는 요인이 될 수 있을 것으로 생각된다. 반대로 LeNSRV1는 보고된 표고 바이러스들 중 가장 낮은 수직감염률을 보였다. 중국의 표고 핵심집단에 대한 LeNSRV1 감염 여부 조사 결과, LeNSRV1에 감염된 대부분의 품종에서 LeNSRV1이 낮은 농도로 검출되었음이 보고되었다 [25]. 따라서 LeNSRV1의 낮은 발현량이 낮은 수직감염률을 일으키는 것으로 추측된다. 한편, LeNSRV1은 수직감염률이 보고된 표고 바이러스들 중 유일하게 음성 단일 가닥 RNA (negative single-stranded RNA)를 게놈으로 갖는다 [26]. 향후 연구를 통해 표고에서의 낮은 발현량과 낮은 수직감염률이 음성 단일 가닥 RNA 바이러스들의 공통된 특성인지, LeNSRV1의 고유한 특성인지 밝혀진다면, 음성 단일 가닥 RNA의 제거 및 관리에 대한 단서를 얻을 수 있을 것으로 기대된다.
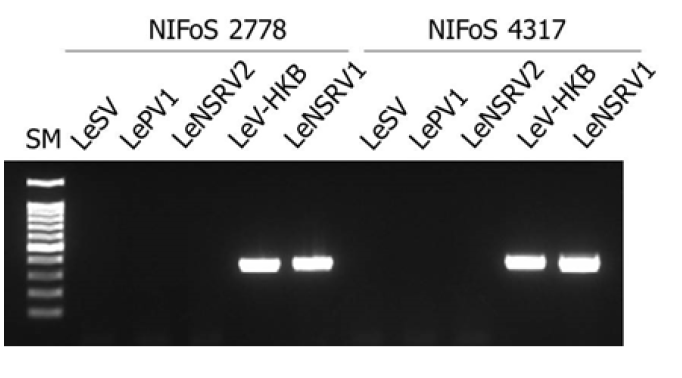
Fig. 1. RT-PCR detection of viral infection from Sanbaekhyang (NIFoS 2778) and Taehyanggo (NIFoS 4317) using specific primer sets targeting each virus-specific RNA-dependent RNA polymerase (RdRp) genes. SM, 100 bp marker; LeSV, Lentinula edodes spherical virus; LePV1, Lentinula edodes partitivirus 1; LeNSRV2, Lentinula edodes negative-strand RNA virus 2; LeV-HKB, Lentinula edodes mycovirus-HKB; LeNSRV1, Lentinula edodes negative-strand RNA virus
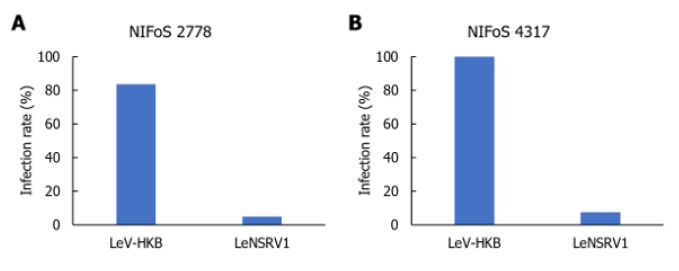
Fig. 2. Infection rate of each RNA mycovirus in the 80 monokaryotic progeny of NIFoS 2778 (Sanbaekhyang) (A) and NIFoS 4317 (Taehyanggo) (B). The viruses were indicated on the x-axis and the infection rates were indicated on the y-axis. The infection rate was calculated by the total number of progenies infected with the virus divided by the total number of progenies examined. LeV-HKB, Lentinula edodes mycovirus-HKB; LeNSRV1, Lentinula edodes negative-strand RNA virus 1.
무바이러스 종균 개발을 위해서는, 바이러스 감염 이핵균사로부터 바이러스를 제거하여 활용하는 방법 외에, 바이러스 감염 이핵균사의 자실체로부터 담자포자를 받아 무바이러스 단핵균사들을 선별하여 활용하는 방법이 가능하다. 본 실험의 결과로 볼 때, 표고에서 바이러스의 수직감염은 대체로 빈번하게 이루어지며, 바이러스 종류에 따라 큰 차이를 보임을 알 수 있다. 또한 선행 연구에서 바이러스 제거 효율도 바이러스 제거 방법 뿐 아니라 바이러스 종류에 따라 큰 차이가 있음이 알려져 있다 [11]. 따라서, 활용하고자 하는 교배 모본이 감염된 바이러스 종류에 맞춰 교잡에 활용할 무바이러스 균주를 확보하는 전략을 달리 수립하는 것이 필요할 것이다.
바이러스 감염과 단핵균사 생장속도
표고와 느타리에서는 이핵균사의 바이러스 제거 실험을 통해 유전적으로 동일한 바이러스 감염 및 미감염 균사들을 확보하고, 이들의 생장속도를 비교한 결과, 바이러스 감염이 이핵균사 생장속도를 감소시킬 수 있음이 보고된 바 있다 [9-11]. 또한 표고 단핵균사 집단 중 생장속도가 빠른 그룹에서 LeV-HKA (Lentinula edodes mycovirus-HKA)바이러스에 감염된 균사들의 비율이 급격히 감소함을 통해, 단핵균사에서도 바이러스 감염이 균사 생장속도에 영향을 줄 가능성이 있음이 보고된 바 있다[12]. 이러한 선행 보고를 바탕으로, 바이러스 감염이 단핵균사의 생장속도에 영향을 미치는지 확인하고자, 각 품종 유래 80개의 단핵균사들의 생장속도를 조사하였다(Fig. 3). 그 결과, 기존 결과들의 경향과 일치하게, 산백향(NIFoS 2778) 유래 단핵균사 집단에서는 두 바이러스에 모두 감염되지 않은 균사들이 생장속도가 빠른 그룹에 편향되어 나타남을 확인하였다. 또한 태향고(NIFoS 4317) 유래 단핵균사 집단에서는 두 바이러스에 모두 감염된 균사들이 생장속도가 느린 그룹에 편향되어 나타남을 확인하였다. 하지만, 핵융합과 감수분열의 결과 생성된 담자포자 유래 단핵균사들은 서로 유전적으로 동일하지 않기 때문에, 단핵균사들의 생장속도를 바이러스 감염 여부만으로 나누어 비교하는 데에는 한계가 있다.
한편, 본 연구팀은 최근 담자포자 유래 단핵균사들의 교배형과 생장속도에 대한 선행 연구 결과, A 교배형과 단핵균사 생장속도 사이에 상관관계가 있음을 확인하였다[23]. 해당 결과가 교배형이 아니라 서로 다른 바이러스 감염에 의한 효과였는지 확인하기 위하여, 본 연구에서 확보한 단핵균사 집단 중 가장 많은 수의 균주를 확보한 LeV-HKB 단일 감염 단핵균사들의 생장속도를 교배형별로 비교하였다. 그 결과, 기존 연구 결과와 일치하게, 두 품종 모두에서 A 교배형에 따라 단핵균사의 생장속도에 차이가 나타남을 확인하였다(Fig. 4). 따라서 A 교배형 또는 A 교배형과 연관된 유전자가 단핵균사들의 생장속도에 영향을 주는 유전적 요인으로 판단된다.
이러한 결과를 바탕으로, 바이러스 감염과 단핵균사 생장속도 사이의 상관관계를 살펴볼 때 담자포자 유래 단핵균사들의 유전적 차이에 의한 생장속도 차이를 최대한 배제하기 위해, A 교배형은 동일하고 감염된 바이러스의 종류의 수가 다른 균사들의 생장속도를 비교하였다. 이 때, 최소 5균주 이상을 확보한 집단에 대해서만 분석을 수행하였다. 단핵균사의 A 교배형별로 바이러스 감염에 따른 생장속도를 비교한 결과, 산백향(NIFoS 2778)과 태향고(NIFoS 4317) 모두에서 통계적으로 유의미한 차이를 확인하지 못했다(Fig. 5). 그러나 분석한 모든 그룹에서 감염 바이러스의 종류가 늘어날수록 평균 생장속도가 감소하는 경향을 확인할 수 있었다. 특히 태향고(NIFoS 4317)의 A2 그룹에서는 LeV-HKB와 LeNSRV1에 모두 감염된 균사들이 LeV-HKB에만 감염된 균사들에 비해 생장속도가 약 19% 감소함을 확인하였다. 따라서, 이핵균사에서 보고된 바와 같이, 바이러스 감염이 단핵균사의 생장을 느리게 하는 영향을 줄 수 있을 것으로 생각된다. 단핵균사 생장속도에 대한 바이러스 감염의 영향을 보다 정확히 알아보기 위해서, 향후 보다 많은 수의, A 교배형은 같지만 바이러스 감염 여부가 서로 다른 단핵균사들을 확보하거나, 바이러스 감염 단핵균사에 바이러스 제거를 시도하여서 바이러스 감염 균주와 유전적으로 동일한(isogenic) 바이러스 미감염 균주를 확보하여 서로의 생장속도를 비교하는 실험이 필요할 것이다.
ACKNOWLEDGEMENTs
This study was supported by a grant from the General Project (FP0800-2020-01) of National Institute of the Forest Science and the Golden Seed Project of ‘Breeding of new strains of shiitake for cultivar protection and substitution of import [213007-05-5-SBH10]’ provided by the Ministry of Agriculture, Food and Rural Affairs, Ministry of Oceans and Fisheries, Rural Development Administration and Korea Forest Service.
적요
표고는 중요한 상업용 버섯이며, 표고에서의 바이러스 감염에 대한 여러 보고가 있었다. 톱밥 재배 품종인 산백향(NIFoS 2778)과 태향고(NIFoS 4317)에 대한 바이러스 검출 결과, 이들이 2개의 mycoviruses (LeV-HKB 및 LeNSRV1)에 감염되어 있음을 확인하였다. 각 품종의 자실체에서 분리된 담자포자에서 유래한 80개의 단핵균사에서 바이러스 감염을 조사한 결과, 대부분의 단핵균사들이 바이러스에 감염되어 있었으며, LeV-HKB와 LeNSRV1의 수직감염률에 차이가 있었다. LeV-HKB가 LeNSRV1 보다 높은 수직감염률을 나타낸 것이다. 따라서 mycovirus의 수직 감염 기작이 바이러스 종에 따라 다른 것으로 보인다. 다음으로, 담자포자 유래 단핵균사들의 생장속도를 조사하여 바이러스 감염과 생장속도의 상관관계를 조사하였다. 바이러스 감염과 균사 생장속도 사이에 통계적으로 유의한 상관관계가 없었지만, 감염된 바이러스의 종류가 늘어날수록 생장속도가 감소하는 경향을 확인하였다. 본 연구는 mycovirus의 수직 감염 기작을 이해하는 데 기여하고, 바이러스에 감염되지 않은 단핵균사를 이용한 무바이러스 품종 개발을 촉진하는 데 기여할 수 있을 것으로 기대된다.