서론
자낭균 효모는 인류 역사에서 오랫동안 식품을 발효를 위해서 이용되었다. 최근에도 발효식품 뿐 이외에도 화장품소재, 바이오에너지 생산 균주, 오염된 환경을 정화하는 환경개선제 등 다양한 분야에 활용되고 있다[1]. 다양한 용도로 활용될 수 있는 효모들을 발굴하고 특성을 분석하는 일은 지속되고 있으며 최근에도 야생 효모를 발굴하고 동정하며 융복합적으로 효모를 활용하기 위한 연구가 지속되고 있다[1].
국내 토양에서는 다양한 야생 효모들이 많이 존재하며 효모들이 배양되는 일반적인 배지를 이용하여 다양한 효모들이 배양되고 있다. 해외에서는 발견이 되었으나 아직 국내에서 발견되지 못한 미기록종(unrecored species)가 꾸준히 발견되고 있으며, 전세계 어디서도 발표되지 않은 신종(new species)의 가능성이 높은 신종후보 효모들도 꾸준히 발굴되고 있다[1]. 국내에서도 다양한 야생 효모를 발굴한고 산업적으로 이용하고자 하는 노력이 필요하다.
Wickerhamomyces 속 효모들은 자낭균문(Ascomycota)에 속하며, Saccharomycetes, Saccharomycetales, Phaffomycetaceae에 속한다. 가장 일반적으로 알려진 Saccharomyces 속 효모와 같은 문(phylum), 강(class), 목(order)에 속해 있어서 Saccharomyces 속 효모와 비슷한 특징을 보이고 있다[2]. Wickerhamomyces 속 효모들은 구형의 자낭포자(ascospore) 또는 모자 모양(hat-shaped)의 자낭포자를 형성하며, 메탄올을 분해하지 못하고, 세포 내 주요 퀴논으로 유비퀴논 Q-7을 가지고 있다[3].
Archaeorhizomyces 속은 Wickerhamomyces 속 효모와 마찬가지로 자낭균문(Ascomycota)에 속하지만, Archaeorhizomycetes, Archaeorhizomycetales, Archaeorhizomycetaceae에 속한다[4]. Archaeorhizomyces 속은 Saccharomyces 속 효모나 Wickerhamomyces 속 효모와는 다른 특징을 보이는데, 성장이 느리고, 주로 식물 뿌리와 관련된 토양에서 흔히 발견되는 곰팡이류로 알려져 있다[4].
환경 시료의 메타 유전체 분석 결과에 따르면, Archaeorhizomycetes 효모는 토양의 전체 효모 군집 중에서 상당 부분을 차지한다고 알려져 있다. 그러나, Archaeorhizomycetes 효모를 분리하고 동정한 연구는 많이 진행되지 않았다[5].
이 연구에서는 국내 토양으로부터 야생 효모를 분리하였고 동정하였다. 세균과 곰팡이의 생장이 제한된 배지를 이용하여 효모를 배양하고 순수분리가 된 균총은 현미경으로 관찰하여 출아가 일어나고 있음을 확인하였다. 동정이 된 효모 중에서 신종으로 가능성이 있는 균주들을 선별하여 형태학적 특징과 생리학적 특징을 분석하였다. 선발된 신종 후보 효모들의 정확한 분자생물학적 유연관계를 확인하기 위하여 효모 동정에 기준이 되고 있는 26S rRNA 유전자의 D1/D2 영역과 ITS (internal transcribed spacer) 영역에 대해 염기서열 분석을 실시하고 유사한 종의 효모들과 서열을 비교하였다.
재료 및 방법
야생 효모의 분리 및 동정
서울시 내 불암산(37°39'41.2"N 127°04'57.0"E)에서 샘플을 채집하였다. 멸균된 튜브에 9 mL의 멸균수를 넣고, 토양 샘플을 1 g 넣어 희석, 현탁 하였다. 이 현탁액을 10배씩 순차적으로 희석하여 yeast extract peptone dextrose (YPD, BD Difco, Franklin Lakes, USA) 한천 배지(pH 3.7-5.0)에 100 μ L씩 분주하여 도말한 후 10℃에서 3-5 일 배양하고 발생한 효모 균총 중 일부를 선별하여 순수 분리하였다[6]. YPD 한천 배지는 chloramphenicol (0.4 g/mL) 0.04%, sodium propionate (0.2 g/mL) 0.02%를 첨가하여 제조하였다.
분리한 효모의 동정을 위해 26S rDNA의 D1/D2 region을 NL1, NL4 primer를 이용하여 PCR 증폭한 뒤 염기서열을 분석하였다[7]. 확보한 염기서열은 미국 국립생물정보센터(National Center for biotechnology Information, NCBI)의 Basic Local Alignment Search Tool (BLAST) 데이터베이스를 이용해 다른 효모의 D1/D2 영역 염기서열과의 상동성을 비교하였고, Clustal X 2.0 프로그램을 이용하여 주변 서열들과 비교, 정렬하였다[8]. 계통수는 MEGA 11.0 프로그램에서 neighbor-joining method를 사용하여 구축하였다[9]. 동정한 효모 중 신종 후보 효모 균주들은 신종 후보 효모임을 명확하게 파악하기 위해 5.8S rDNA 및 ITS 영역을 ITS1, ITS4 primer를 이용하여 PCR 증폭한 뒤 염기서열을 분석하였다[10]. 마찬가지로 NCBI의 BLAST 데이터베이스를 이용하여 다른 효모의 ITS 서열과의 상동성을 비교하고, Clustal X 2.0 프로그램을 이용하여 주변 서열들과 비교, 정렬하였다. 계통수는 5.8S rRNA, ITS 영역 및 D1/D2 영역을 포함하여 MEGA 11.0 프로그램에서 neighbor-joining method를 사용하여 구축하였다.
분리된 효모의 형태학적 특성
효모의 위상차 현미경(DM500, LEICA, Wetzlar, Germany) 관찰을 위해 YPD 한천 배지에서 3일간 배양한 효모의 플레이트에서 깨끗한 단일 균총을 분리하여 슬라이드 글라스 위의 멸균 증류수에 희석하고 커버 글라스로 덮어주었다. 위상차 현미경을 통하여 세포의 크기, 출아여부를 확인하였다.
효모 신종 후보종들의 탄소원 활성 측정
효모의 탄소원 활성을 측정하기 위해 API 20C AUX kit (bioMerieux, Hazelwood, Mo.)를 이용하였다. YPD 한천 배지에서 배양한 균을 모아 suspension medium (2 mL)에 넣고 2 McFarland 탁도에 맞추어 희석하였다. 탁도를 맞춘 균주 혼합물은 API C Medium에 100 μL씩 넣어 기포가 생기지 않도록 혼합하였다. API C medium의 균액을 스트립의 큐플에 기포가 생기지 않도록 피펫을 이용해 적정량 분주하였다. 이를 30℃에서 48-72시간 배양하였다. 음성 대조군과 비교하여 혼탁도가 높은 큐플을 양성 반응으로 결과를 기록하였다[11].
결과
야생 효모 분리, 동정과 신종 후보 균주 선별
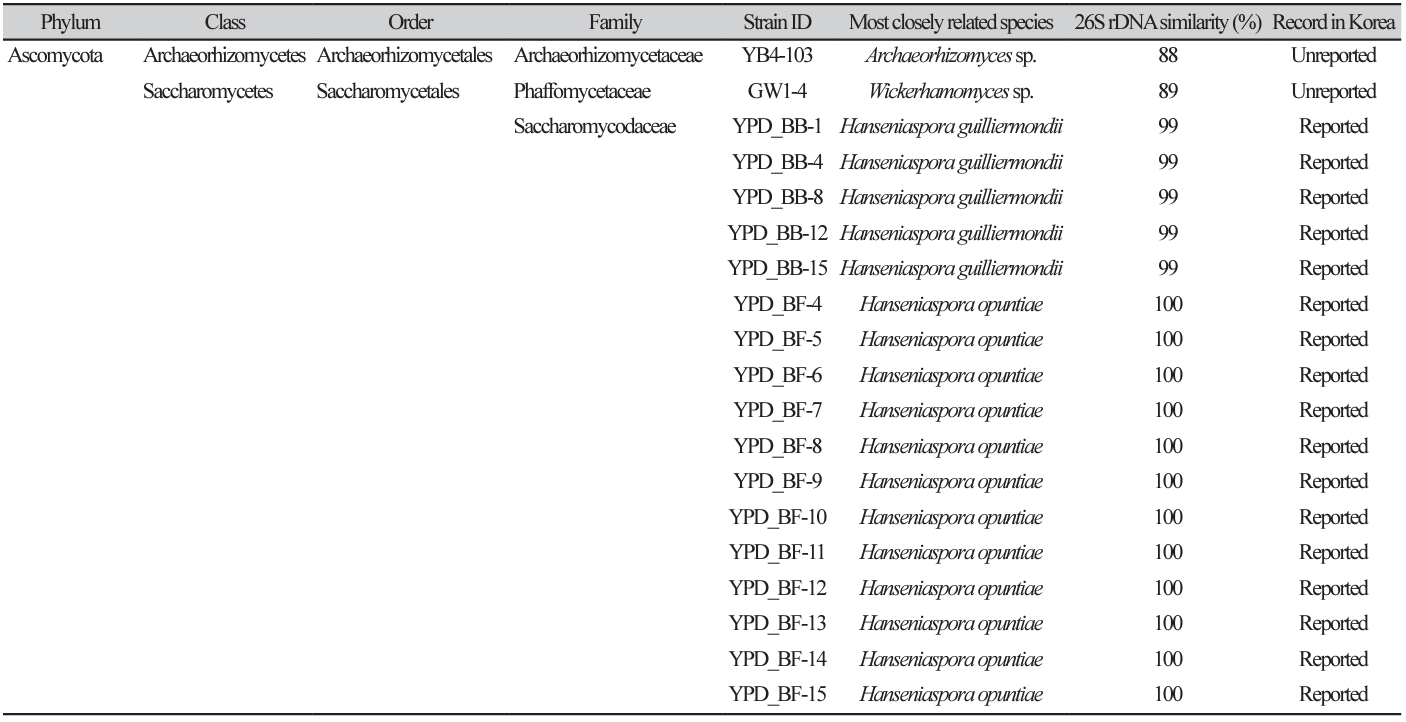
불암산의 토양에서 야생 효모들을 19종 분리하였다. 26S rRNA 유전자의 D1/D2 영역을 시퀀싱하고 얻어진 서열을 미국 국립생물정보센터(NCBI, http://ncbi.nlm.nih.gov)의 데이터베이스를 통해 동정하였다. 그 결과, 샘플로부터 Archaeorhizomycetaceae 1 개 균주, Phaffomycetaceae 1 개 균주 그리고 Saccharomycodaceae 17 개 균주, 총 19 개 균주가 분리되었다(Table1).
분리한 19 개 균주 중에서, GW1-4 균주는 Wickerhamomyces xylosivorus NBRC 111553T 균주와 88.97%의 D1/D2 영역 유사도를 보였다. 그리고, YB4-103 균주는 Archaeorhizomyces finlayi CBS 128710T 균주와 87.75%의 D1/D2 영역 유사도를 보였다. 따라서, 두 균주는 신종 후보로 판단되어 이 후 실험을 진행하였다.
분리, 선별된 효모 신종 후보 균주들의 분자계통학적 위치
신종 후보로 선별된 Wickerhamomyces sp. GW1-4 균주와 Archaeorhizomyces sp. YB4-103 균주의 D1/D2 영역을 기준으로 근연종 효모들과의 염기서열 유사도를 확인하였다. GW1-4균주의 D1/ D2 영역 염기서열은 길이가 503 bp 이었고, ITS 영역은 길이가 491 bp 이었다. 이 염기서열들은 GenBank에 등록하였으며, D1/D2 영역 accession 번호는 OR764776이고, ITS 영역 accession 번호는 OR825822이다. 균주 GW1-4와 Wickerhamomyces xylosivorus NBRC 111553T 균주를 비교한 결과, D1/D2 영역은 11.03%의 염기서열 차이를 보였으며, ITS 영역은 14.86%의 염기서열 차이를 보였다.
YB4-103균주의 D1/D2 영역 염기서열의 길이는 509 bp 이었고, ITS 영역 염기서열의 길이는 326 bp 이었다. 이 염기서열들은 GenBank에 등록하였으며, D1/D2 영역 accession 번호는 OR764775이고, ITS 영역 accession 번호는 OR825823이다.
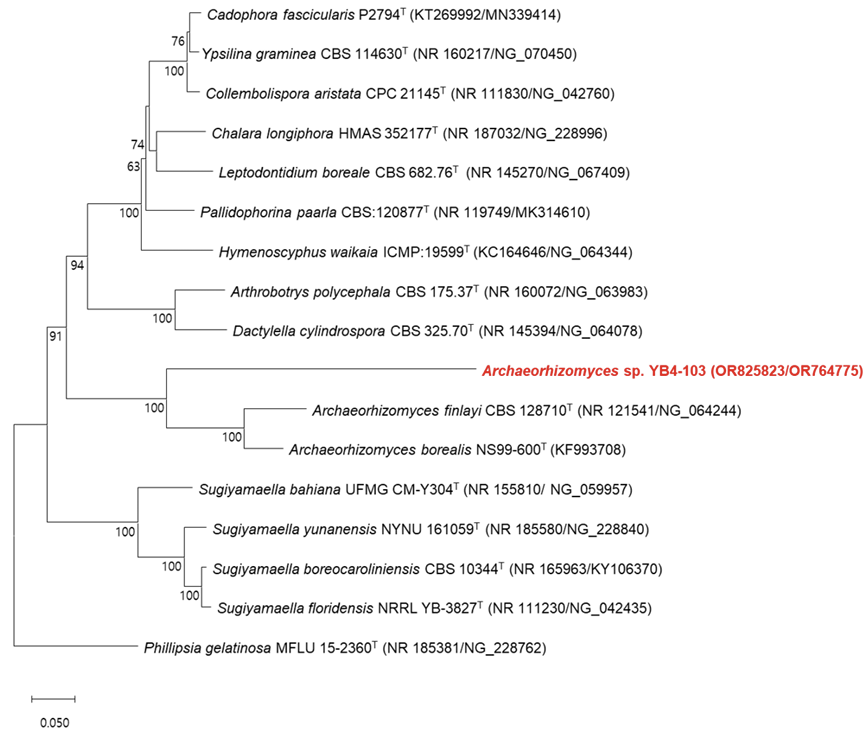
균주 YB4-103은 Archaeorhizomyces finlayi CBS 128710T 와 9.5%의 D1/D2 영역의 염기서열 유사도 차이를 보였으며, ITS 영역의 염기서열 유사도 차이는 8.83%를 보였다. D1/D2 영역에서 3 개 염기의 치환이 나타날 때 서로 다른 종(species)으로 구분되는 기준을 적용하여[12], 균주 GW1-4는 Wickerhamomyces 속에 속하는 신종 후보 균주라고 판단되었고, 균주 YB4-103는 Archaeorhizomyces 속에 속하는 신종 후보 균주라고 판단되었다. D1/D2 영역의 염기서열을 이용하여 neighbor-joining 기법으로 구성된 두 균주의 계통수는 각각 Fig. 1과 Fig. 2에 나타내었다. 또한, ITS 영역부터 D1/D2 영역까지 이어진 염기 서열 정보를 이용해 neighbor-joining 기법의 계통수를 작성한 그림은 각각 Fig. 3와 Fig. 4와 같았다.
Fig. 1
Neighbor-joining phylogenetic tree based on the nucleotide sequences of D1/D2 region of 26S rDNA showing the position of novel yeast GW1-4. The tree was generated by the neighbor-joining method, using MEGA11. Bootstrap values (>70%) are shown at the branch nodes. Bar, 0.02 substitutions per nucleotide position. Saccharomyces cerevisiae NRRL Y-12632T was used as an outgroup. Ttype strain.

Fig. 2
Neighbor-joining phylogenetic tree based on the nucleotide sequences of D1/D2 region of 26S rDNA showing the position of novel yeast YB4-103. The tree was generated by the neighbor-joining method, using MEGA11. Bootstrap values (>70%) are shown at the branch nodes. Bar, 0.05 substitutions per nucleotide position. Phillipsia gelatinosa MFLU 15-2360T was used as an outgroup. Ttype strain.
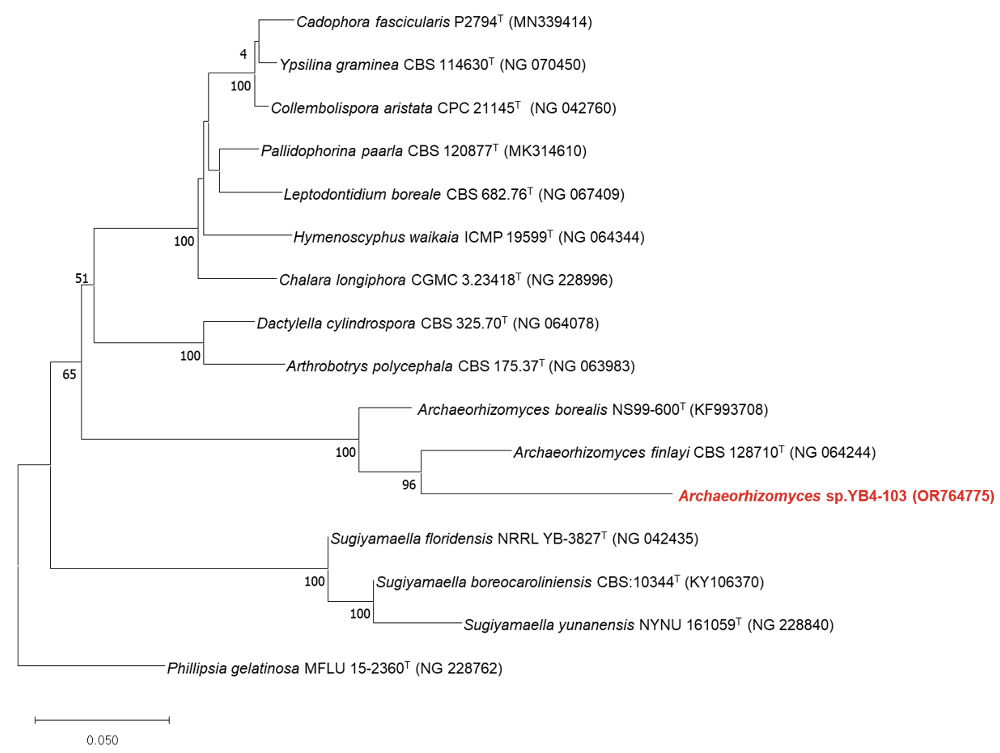
Fig. 3
Phylogenetic tree based on the concatenated sequences of the internal transcribed spacer (ITS) regions and D1/D2 region of the large subunit (LSU) rRNA gene and constructed by the neighbor-joining method, showing relationships of a novel species candidate GW1-4 and closely related species. Saccharomyces cerevisiae CBS 1171T was used as an outgroup. Bootstrap values (>70%) are shown at the branch nodes. Accession numbers were shown in parentheses. Bar, 0.05 substitutions per nucleotide position.Ttype strain.

Fig. 4
Phylogenetic tree based on the concatenated sequences of the internal transcribed spacer (ITS) regions and D1/D2 region of the large subunit (LSU) rRNA gene and constructed by the neighbor-joining method, showing relationships of a novel species candidate YB4-103 and closely related species. Phillipsia gelatinosa MFLU 15-2360T was used as an outgroup. Bootstrap values (>70%) are shown at the branch nodes. Accession numbers were shown in parentheses. Bar, 0.05 substitutions per nucleotide position.Ttype strain.
선별된 효모 신종 후보 균주들의 균학적 특성
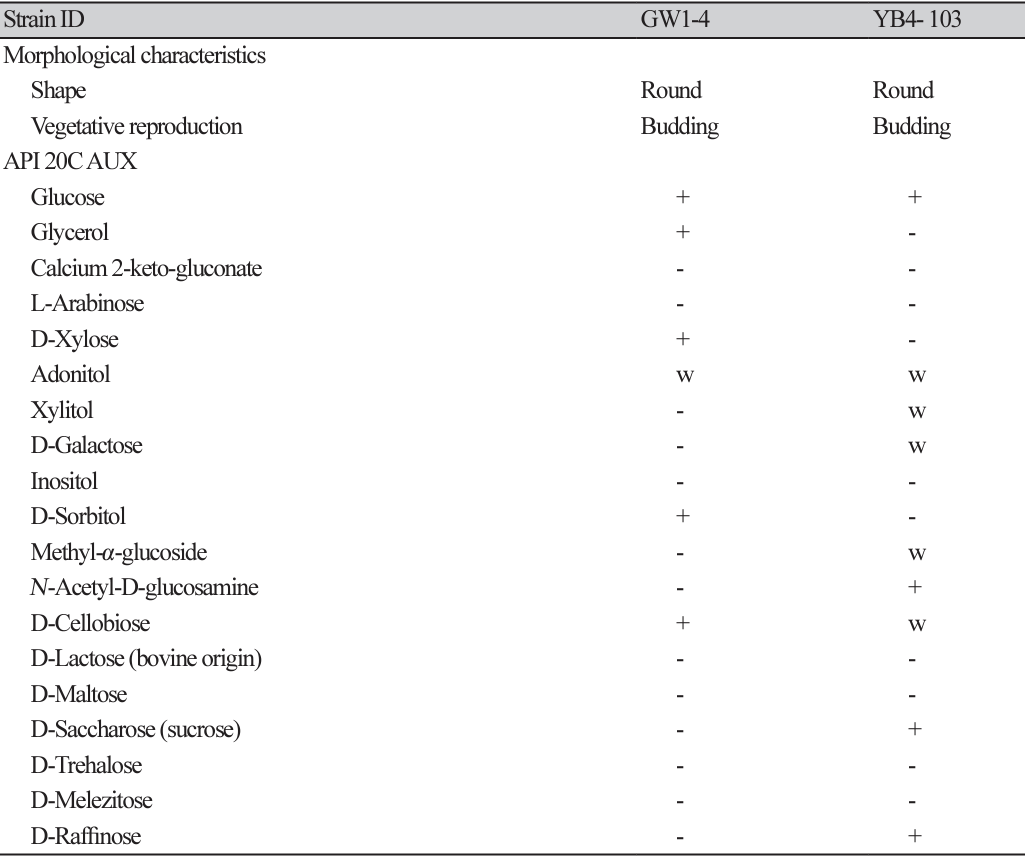
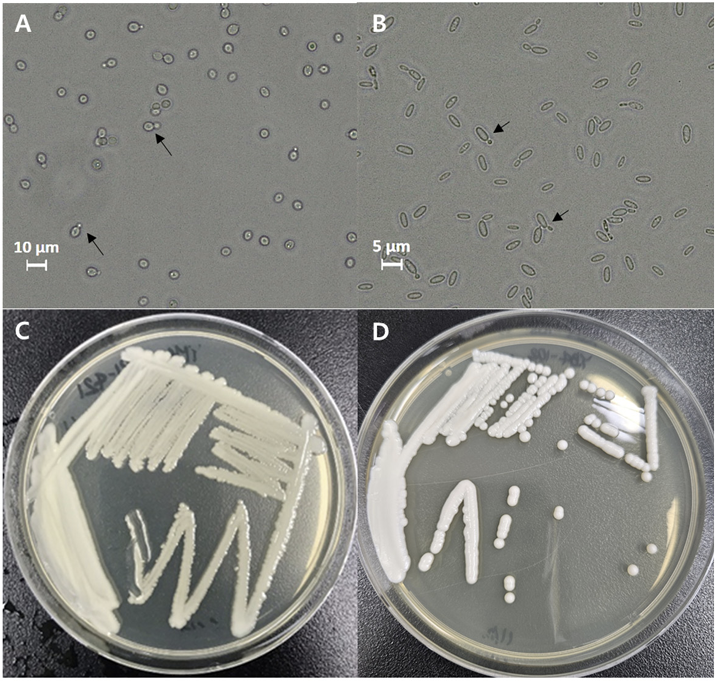
신종 후보로 최종 선별된 Wickerhamomyces sp. GW1-4 균주와 Archaeorhizomyces sp. YB4-103 균주의 균학적 특성은 Table 2에 나타내었으며, 위상차 현미경으로 관찰한 두 균주의 cell 사진은 Fig. 5에 나타내었다. 균주 GW1-4는 최근연종인 Wickerhamomyces xylosivorus NBRC 111553T 균주와 비교한 결과, 형태학적으로 동일한 특징을 보여주었다[12]. 그러나, 균주 YB4-103는 최근연종인 Archaeorhizomyces finlayi CBS 128710T 균주와 비교한 결과, 형태학적으로 다른 특징을 보여주었다. Archaeorhizomyces finlayi CBS 128710T 균주는 budding이 관찰되지 않은 반면에, 균주 YB4-103는 budding이 관찰되었다[4]. 이 신종 후보 효모들의 형태적, 배양적 특징으로 두 균주 모두 세포의 형태가 타원형이고, 출아하여 영양 증식하였다. 또한, 균주 GW1-4의 세포의 크기는 3.0-5.2 μm × 3.0-5.0 μm이며 균주 YB4-103의 세포 크기는 4.0-6.0 μm × 2.0-3.0 μm이었다. 동정된 GW1-4와 YB4-103 균주는 국립농업과학원 농업미생물은행에 각각 KACC 410359와 KACC 410360으로 기탁하였다.
Fig. 5
Morphology of cells from strain GW1-4 and strain YB4-103 incubated at 25℃. All strains were grown after 3 days on yeast extract peptone dextrose (YPD) agar. The budding cells of Wickerhamomyces sp. GW1-4 (A) and Archaeorhizomyces sp. YB4-103 (B). bars, 10 µm. The colonies of Wickerhamomyces sp. GW1-4 (C) and Archaeorhizomyces sp. YB4-103 (D).
효모 신종 후보 균주들의 탄소원 활성 측정
Wickerhamomyces sp. GW1-4의 탄소원 동화 가능 여부 측정 결과는 다음과 같았다. Glucose, glycerol, D-xylose, D-sorbitol 그리고 D-cellobiose는 양성, adonitol은 약한 양성, calcium 2-ketogluconate, xylitol, L-arabinose, N-acetyl-D-glucosamine, D-galactose, inositol, methyl-α-D-glucoside, D-lactose (bovine origin), D-maltose, D-saccharose (sucrose), D-trehalose, D-melezitose 그리고 D-raffinose는 음성이었다. GW1-4 균주는 최근연종인 Wickerhamomyces xylosivorus NBRC 111553T 균주와 탄소원 활성에서도 차이를 보였다. GW1-4 균주는 adonitol, D-sorbitol, N-acetyl-Dglucosamine, D-trehalose, D-raffinose의 탄소원 활성 측정 결과에서 차이를 나타내었다[12].
Archaeorhizomyces sp. YB4-103 균주의 탄소원 동화 가능 여부 측정 결과는 다음과 같았다. Glucose, N-acetyl-D-glucosamine, D-saccharose (sucrose) 그리고 D-raffinose는 양성, adonitol, xylitol, D-galactose, methyl-α-D-glucoside 그리고 D-cellobiose는 약한 양성, glycerol, calcium 2-keto-gluconate, L-arabinose, D-xylose, inositol, D-sorbitol, D-lactose (bovine origin), D-maltose, D-trehalose 그리고 D-melezitose는 음성이었다.
고찰
불암산 토양 샘플에서 야생 효모 19 균주를 분리하였다. 효모의 D1/D2 영역과 ITS 영역을 기준으로 야생 효모를 동정하여 지금까지 보고되지 않은 신종 후보 효모 2 균주를 선별하였다. 신종 후보 효모 GW1-4 균주와 YB4-103 균주는 위상차 현미경과 API 20C AUX kit를 통해 균학적 특성을 조사하였다. GW1-4 균주와 YB4-103 균주의 세포 모양은 타원형이며, 균총은 볼록하고, 부드러운 형태를 보여주었다. 균주 GW1-4는 glucose, D-sorbitol, 그리고 D-cellobiose를, 균주 YB4-103는 glucose, N-acetyl-D-glucosamine, D-saccharose, 그리고 D-raffinose를 탄소원으로 사용할 수 있음을 확인하였다.
Fig. 1에서 GW1-4 균주는 다른 Wickerhamomyces 속 효모들과 가까운 유연관계를 보여주었다. 분자계통학적으로 Pichia 속, Debaryomyces 속, Saccharomyces 속 효모들과 가까이 위치하였다. 그러나, Wickerhamomyces 속 효모들은 Phaffomycetaceae 과에 속하며, Pichia 속, Debaryomyces 속, Saccharomyces 속 효모들과는 다른 계통을 보이는 것을 알 수 있다. Wickerhamomyces 속 효모들은 국내 토양에서 종종 발견되며, 국내에 서식하는 곤충의 장에서도 많이 발견되고 있다[13].
GW1-4 균주는 최근연종인 Wickerhamomyces xylosivorus NBRC 111553T 와 11.0%의 염기서열 차이를 보였다. Wickerhamomyces 속 효모들은 Candia 속 효모들과 가까운 유연관계를 보이고 있다(Fig. 1). Candia 속 효모들은 오랜 기간 형태학적으로만 동정된 종이 많아서 계통수를 분석하면 여러 다른 과에 산발적으로 흩어져서 위치하고 있다. 재분류(reclassification)를 비롯한 많은 재동정이 필요한 속이다. 상당수의 Candia 속 효모들은 Wickerhamomyces 속 효모로 재분류가 되어질 것으로 예상되어진다.
GW1-4 균주는 최근연종인 Wickerhamomyces xylosivorus NBRC 111553T 균주와 탄소원 활성에서도 차이를 보였다. GW1-4 균주는 adonitol, D-sorbitol, N-acetyl-D-glucosamine, D-trehalose, D-raffinose의 탄소원 활성 측정 결과에서 차이를 나타내었다[14].
Fig. 2에서 YB4-103 균주는 Archaeorhizomycetes에 속한 효모들과 유연관계를 가지고 있음을 알 수 있다. 이 효모들은 Saccharomyces 속 효모들과는 분류학적으로 거리가 먼 효모들이며 Saccharomyces 속 효모들과는 다른 특성이 기대된다. YB4-103 균주는 최근연종인 Archaeorhizomyces f inlayi CBS 128710T와 9.5%의 염기서열 차이를 보이고 있다. D1/D2 영역의 염기 차이가 3개(0.5%) 차이를 보일 때 다른 종으로 판단되는 기준에 의하면 YB4-103 균주는 최근연종인 Archaeorhizomyces f inlayi CBS 128710T와 유연관계가 아주 멀다는 것을 생각할 수 있다.
Archaeorhizomyces 속 효모들은 균사를 만드는 것으로 알려진 바 있다. 환경 조건에 따라서 출아와 균사 형성을 선택할 수 있다. 출아(budding)가 관찰되지 않는 근연종들과 달리 YB4-103 균주는 출아를 통한 생식이 관찰되었다[4].
이에 따라 GW1-4균주와 YB4-103 균주는 신종 후보종이라고 판단된다. 그러나, 그동안 효모의 분류가 형태학적인 특징에 의해 주로 이루어져 왔고 26S rRNA 유전자 D1/D2 서열에 기준한 분류가 최근에 이루어졌기 때문에 오동정 결과를 많이 포함하고 있다[15]. 효모의 분류에서 중요한 type strain 균주들의 26S rRNA 유전자 D1/D2 서열을 체계적으로 모아 놓은 데이터베이스도 존재하지 않으며 기존의 데이터베이스가 있다고 하더라도 업데이트가 매우 늦게 이루어지고 있는 상황이므로 효모 분류의 체계화를 위한 전세계적 노력이 필요한 실정이다.