서론
1780년 미국 동부 지역에서 처음 보고된 과수화상병 (fire blight disease)은 사과, 배 등의 장미과에 속하는 식물에 큰 피해를 주는 식물 병해로써 병원세균인 Erwinia amylovora에 의해 발생한다 [1]. 이병은 다수의 유럽국가를 비롯하여 여러 아시아 국가에서도 발생이 보고되었고 국내에서는 2015년 경기도 안성과 제천에서 처음 발생하였다 [2]. 병의 전파 속도가 빨라서 2021년에는 22개 시군의 618개 농가, 2022년도에는 19개 시군에 210개 농가 등에서 발생하여 과수 농가에 큰 피해를 입히고 있다[2]. 이 병원균은 과수에 꽃이 필 때 방화 곤충에 의해 옮겨지거나, 기공과 상처를 통해 잎에 침입하여 증식하고 확산된다. 강우시에는 꽃에서 세균액이 흘러나와 전파되기도 하며, 건조할 때는 바람에 의해 전파되는 것으로 알려져 있다 [3].
화상병 방제를 위해서 개화기에 streptomycin과 구리 합성 화합물을 포함한 약제를 사용하며 감염 가지의 경우 6-12인치 정도의 가지치기를 진행하는데 병이 심할 경우 나무를 뽑아 소각하거나 매몰한다 [3]. 우리나라의 경우 환경문제와 산불 위험성을 고려하여 매몰 방제를 수행하고 있다. 이방제법은 깊이 5 m, 넓이 2 m 크기의 구덩이를 파고 생석회를 뿌린 후 병 발생 과원의 모든 기주식물을 굴취하여 매몰하는 방법이다. 이렇게 매몰처리한 과원에서는 2년간 화상병 기주식물의 식재가 금지된다 [4]. 그러나 아직 매몰방제가 최적의 방제인지 분명하지 않다. 따라서 방제방법의 안전성을 입증하기 위해서 매몰된 이병식물 및 이병식물 주변의 토양에서 화상병균 잔존 여부 조사가 필요하다. 이에 따라 본 연구팀은 화상병 이병식물 매몰지 토양에 대한 미생물 조사를 수행하여 오고 있다. 배나무 과원 화상병 매몰지 토양에 대한 미생물상 조사를 하면서 국내에 기록되지 않은 진균 8종이 발견된 바 본 연구에서는 이들 종에 대한 동정과 더불어 형태학적, 분자유전학적 특성을 기술하여 보고하고자 한다.
재료 및 방법
화상병 이병 식물 매몰지의 토양 시료 채취.
토양시료 채취 장소로서 경기도 안성 소재의 배 (Pyrus pyrifolia var. culta. (Burm. f.) Nakai.) 과수 매몰지 2곳을 선정하였다. 안성시 공도읍 마정리 (37°00'40.7"N 127°11'35.0"E)는 국내 화상병이 첫 발생한 과원의 매몰지로서 매몰한지 최소 38개월 이상 된 곳이다. 안성시 서운면 현매리 (36° 56'14.4"N 127°13'19.3"E)는 매몰한지 24개월 이상 된 곳이다. 토양 채취는 농촌진흥청의 허가를 받고 식물방역 규정을 준수하여 진행하였다. 2019년 8월과 2020년 6월에 포크레인으로 이병식물 매몰지 마다 3개 지점을 찾아 약 3 m 깊이까지 파헤친 후 이병식물을 확인하고 식물과 접촉한 주변의 토양 시료 약 300 g을 알콜로 살균한 모종삽을 이용하여 무균용기에 채취하였다. 채취한 토양 시료는 저온 쿨러에 담아 실험실로 운반하였다.
진균분리 및 동정.
채취한 토양시료는 생물안전 2등급 허가시설 실험실에서 분석하였다. 토양 시료 일부를 20 mL의 멸균 증류수를 담은 50 mL conical tube에 넣은 후 Vortex mixer를 이용하여 3분간 Vortexing 하여 토양시료를 현탁시켰다. 현탁액을 멸균 증류수를 이용하여 10-4까지 점진적으로 희석을 진행한 후 희석액 100 μL를 Dichloran-Glycerol 18% (DG18) 배지에 도말하고 25℃ 배양기에서 5일간 배양하였다. 배양된 배지에 자란 진균 균총의 크기, 모양, 색 등의 형태학적 특징을 육안으로 관찰 후 서로 달라 보이는 균총들을 취하여 Potato Dextrose Agar (PDA)에 순수분리 하였다. 분리된 균주는 DK (Dankook University) 또는 DUCC (Dankook University Culture Collection) 번호를 부여하였다. 순수분리가 완료된 진균의 미세구조는 광학현미경(BX53; Olympus, Tokyo, Japan)을 이용하여 관찰하였다. 배양된 진균의 균사체로부터 Direct DNA Prep (NAVIBIOTECH, Cheonan, Korea)을 사용하여 genomic DNA를 추출하였다. 추출한 DNA를 주형으로 하여 internal transcribed spacer (ITS) region [5], 28S ribosomal DNA gene (LSU rDNA) [6], BenA gene (BenA) [5] 프라이머를 사용하여 ITS, LSU rDNA와 BenA에 대해 PCR을 수행하여 증폭하였다. PCR은 94℃에서 5분간 pre-denaturation 한 후, denaturation 94℃ 30초, annealing 56℃ 30초, elongation 72℃ 30초 조건에서 총 35 cycle 진행하고 72℃에서 10분간 final extension하여 수행하였다. 증폭된 PCR product는 1% Agarose gel에 전기영동하여 증폭 DNA 밴드를 확인하였다. 증폭이 확인된 PCR product는 High Pure PCR Product Purification Kit (Roche, Indianapolis, IN, USA)를 사용하여 정제한 후 MACROGEN사 (Seoul, Korea)에 염기서열 분석을 의뢰하였다.
분자계통학적 분석.
결정된 ITS, LSU rDNA, BenA 염기서열은 분자적 동정을 위해 미국 National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov)에 있는 BLAST 프로그램을 사용하여 NCBI 데이터베이스에 등록되어 있는 진균들의 ITS, LSU rDNA, BenA 염기서열과 상동성을 비교하였다. 계통분석을 위해서는 분리 균주와 유의한 상동성을 나타내는 비교 taxon 균주의 염기서열을 NCBI의 GenBank에서 다운받았다. MEGA10 프로그램 [7]의 Clustal W [8]로 다중정렬을 실시하고 염기서열 유사도를 분석하였다. 계통도 분석은 Kimura 2-parameter 모델과 neighbor-joining 방법 [9]으로 분석하였고 계통도 가지의 clade 신뢰도는 1,000번의 bootstrap을 이용하여 평가하였다.
결과 및 고찰
본 연구에서 분리 및 동정된 국내 미기록 토양 진균에 대한 각 종의 형태학적 특징과 염기서열분석을 통한 계통 분석 결과를 아래와 같이 제시하였다. 모든 균주는 국립생물자원관에 기탁하여 NIBR (National Institute of Biological Resources) 번호를 받았다. 분석된 염기서열은 NCBI 데이터베이스인 GenBank에 등록하였으며 등록번호는 각 종마다 분석한 계통도의 균주 번호 앞에 표시하였다
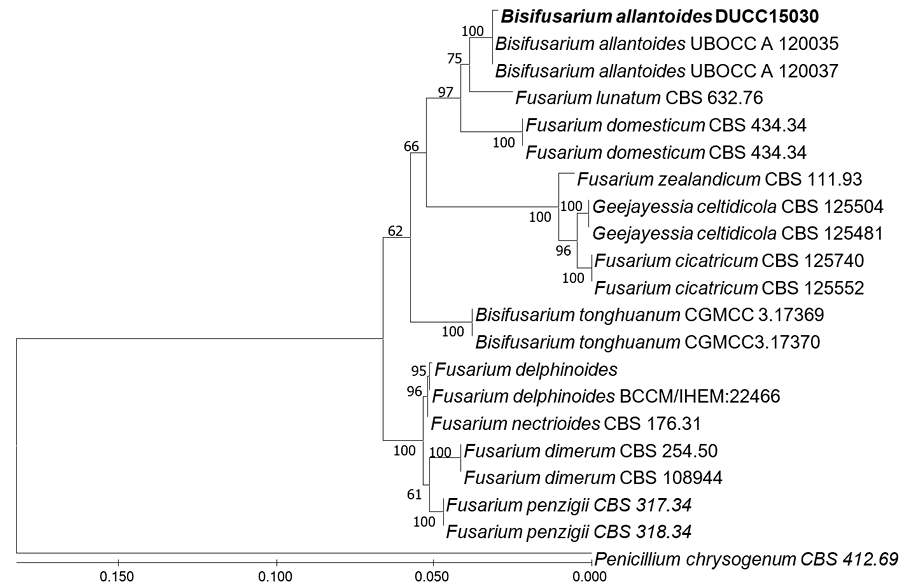
Bisifusarium allantoides O. Savary, M. Coton, E. Coton & J.L. Jany, Index Fungorum 504: 1 (2021) [MB#559338]
DUCC15030 (NIBRFGC000508320) 균주는 PDA 배지에서 7일간 배양하였을 때 균총 직경이 약 44-46 mm 정도로 균사가 배지 표면에 붙어 생육하였다. 앞면에서 봤을 때 하얀색 분말 같은 균사가 위에 겹쳐져 있는 주황색 집락을 형성하였다 (Fig. 1A). Malt Extract Agar (MEA)에서는 7일간 배양했을 때 균총의 직경은 약 22-23 mm 정도이고 PDA 앞에서 봤을 때 중앙이 짙은 주황색이며 바깥으로 갈수록 연한 주황을 나타내는 집락을 형성하였다 (Fig. 2A). Microconidia는 7-10 ×2-3 μm의 크기로 반달 모양이었으며 격벽이 있는 것과 없는 것이 존재하였다. 균사의 두께는 약 1.5-2 μm정도이며 conidiophores 관찰되지 않았다 (Fig. 3A). 이러한 형태적 특징은 원기재문에 서술된 microconidia (4.7-9.7×1.5-3.2 µm)와 유사하였다 [10]. B. allantoides 은 2021년 Savary 등에 의해 프랑스에서 보고된 종이며, French soft 치즈에서 처음 분리되었다. DUCC15030의 ITS 및 LSU rDNA를 분석한 결과, 각각 B. allantoides UBOCC_A_120035 (MW654536) 및 B. allantoides UBOCC_A_120035 (MW654524)와 99.78% 및 100% 상동성을 보였다. 두 서열을 유합하여 계통수를 작성한 결과 DUCC15030은 B. allantoides UBOCC_A_120035와 같은 계통을 형성하였다 (Fig. 4). 형태학적 비교 및 분자유전학적 분석을 통한 계통수 결과를 바탕으로 DUCC15030 (NIBRFGC000508320)을 국내 미기록종인 Bisifusarium allantoides로 동정하였다.
Fig. 1
Image of colony morphology after culturing unrecorded fungi isolated from fire blight burial-controlled soil in Anseong in 2020 on potato dextrose agar (PDA) for 7 days at 25℃. Capital letter indicates front side and small letter indicates back side. A, a: Bisifusarium allantoides. B, b: Botryotrichum domesticum. C, c: Microascus paisii. D, d: Paraphoma pye. E, e: Phaeosphaeria culmorum. F, f: Ramopialophora globispora. G, g: Sordaria tomentoalba. H, h: Striaticonidium brachysporum.
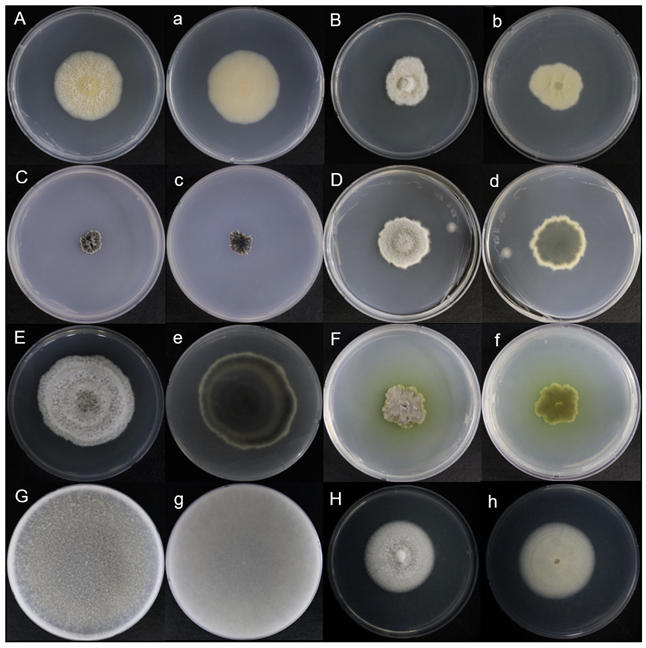
Fig. 2
Image of colony morphology after culturing unrecorded fungi isolated from fire blight burial- controlled soil in Anseong in 2020 on malt extract agar (MEA) for 7 days at 25℃. Capital letter indicates front side and small letter indicates back side. A, a: Bisifusarium allantoides. B, b: Botryotrichum domesticum. C, c: Microascus paisii. D, d: Paraphoma pye. E, e: Phaeosphaeria culmorum. F, f: Ramopialophora globispora. G, g: Sordaria tomentoalba.
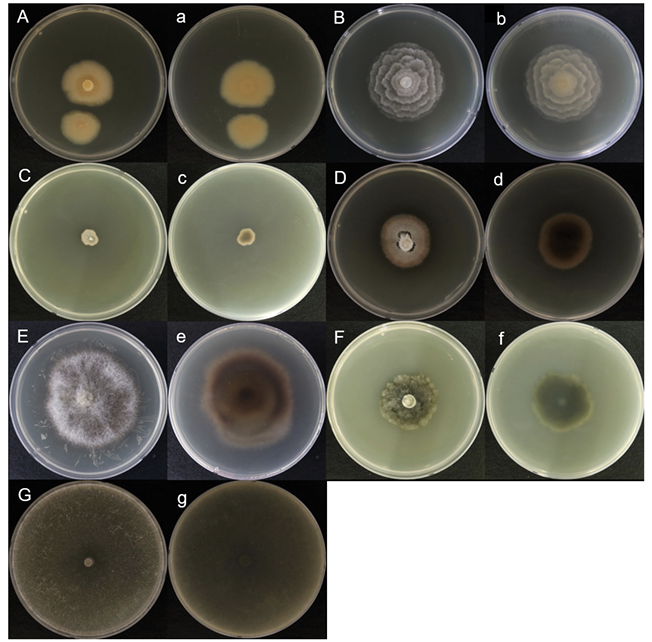
Fig. 3
Light microscopic images of unrecorded fungi isolated from Anseong soil in 2020. A: Bisifusarium allantoides. B: Botryotrichum domesticum. C: Microascus paisii. D: Paraphoma pye. E: Phaeosphaeria culmorum. F: Ramopialophora globispora. G: Sordaria tomentoalba. H: Striaticonidium brachysporum. Scale bar=10 μm.
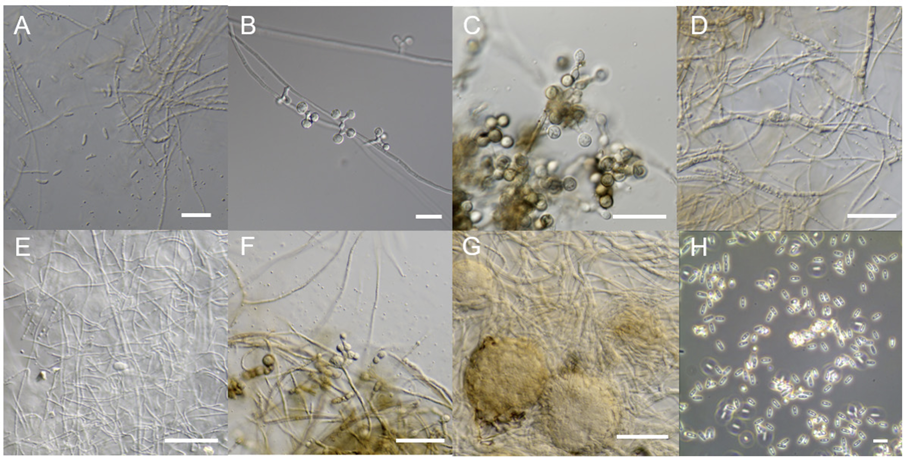
Fig. 4
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) nucleotide sequences of Striaticonidium brachysporum DK14-5 (NIBRFGC000505480). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium chrysogenum CBS 412.69 was used as an out-group.
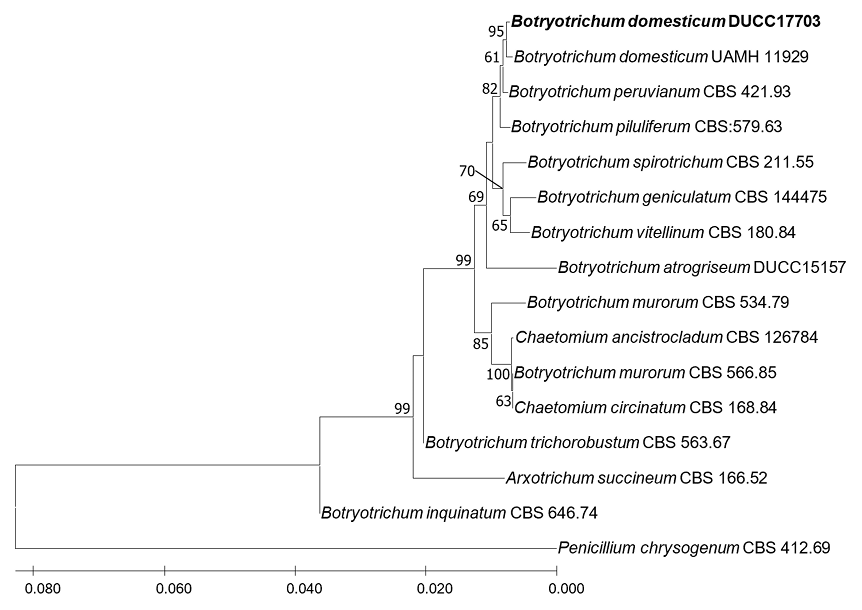
Botryotrichum domesticum D.W. Li & N.P. Schultes, Botany 97(6): 314 (2019) [MB#828185]
DUCC17703 (NIBRFGC000508334) 균주는 PDA 배지에서 7일간 배양하였을 때 균총의 직경이 17-18 mm 정도로, 아이보리 색의 두꺼운 균사를 생성하며 생장하였다 (Fig. 1B). MEA에서는 PDA와는 크게 다른 중앙에서부터 물결이 이는 모양의 얇은 균사를 가지는 아이보리 색의 균총을 형성하였다 (Fig. 2B). 광학현미경으로 관찰한 결과 격벽이 존재하는 4 μm 정도 두께의 균사에서 2-4갈래의 conidiophore를 형성하고 끝에 표면이 울퉁불퉁한 12-13 μm 크기의 conidia를 생성하였다 (Fig. 3B). 이는 원기재문에 서술된 9.7-17×9.7-11 μm 크기의 conidia와 비슷한 크기이다 [11]. B. domesticum을 포함하는 Botryotrichum 속은 1885년 토양과 실내 공기에서 처음 분리 동정되었다 [12]. B. domesticum 또한 2019년에 미국 매사추세츠 가정집에서 처음 분리되었다 [11]. DUCC17703의 ITS 및 LSU rDNA를 분석한 결과, B. domesticum UAMH 11929의 ITS (MH899168) 및 LSU rDNA (NG_068583) 서열과 각각 100% 및 99.54%의 상동성을 보였다. ITS 및 LSU 서열을 유합하여 계통수를 그렸을 때 DUCC17703은 B. domesticum UAMH 11929과 같은 계통을 형성하였다 (Fig. 5). 형태학적 비교 및 분자유전학적 분석을 통한 계통수 결과에 따라 DUCC17703 (NIBRFGC000508334)를 국내 미기록종인 Botryotrichum domesticum으로 동정하였다.
Fig. 5
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) sequences of Botryotrichum domesticum DUCC17703 (NIBRFGC000508334). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium chrysogenum CBS 412.69 was used as out group.
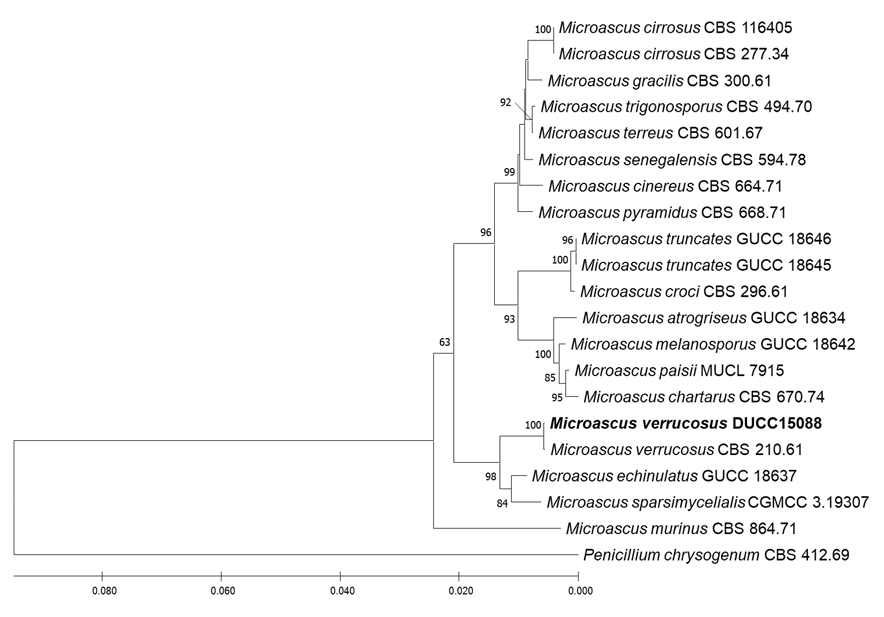
Microascus verrucosus Sand.-Den., Gené & Cano, Persoonia 36: 23 (2015) [MB#809421]
DUCC15088 (NIBRFGC000508314) 균주는 PDA 배지에서 7일간 배양하였을 때 균총의 직경은 7-8 mm 정도였으며, 회색의 외곽선을 가지는 검은색의 딱딱하고 두꺼우며 위로 약 1cm 정도 융기된 형태를 보였다 (Fig. 1C). MEA에서는 아이보리 색의 부드러우며 융기되지 않은 균총을 형성하였다 (Fig. 2C). 광학현미경 관찰 결과 5-6×4-5 μm 크기의 2-3개의 원형 conidia가 7-8 ×1.5-2.5 μm의 annellide에 부착되는 형태를 보였다. Conidia는 내부가 보이는 투명하고 겉이 매끄러운 것과 갈색으로 불투명하며 겉이 울퉁불퉁한 것이 존재하였다 (Fig. 3C). 이런 형태는 원기재문에 서술된 M. verrucosus의 Annellides (8-10×1-3 μm) 및 conidia(5-7×4.5-6 μm)와 비슷하였다 [13]. Microascus 속은 면역 저하 환자에게 손발톱 감염, 만성 육아종 질환, 화농성 피부염 등 각종 염증 및 질환을 유발할 수 있다고 알려져 있다 [14]. 또한 최근에는 폐 이식을 받은 사람에게 감염하여 사망에 이르게 한 경우도 있다 [15]. DUCC15089 균주의 ITS 및 LSU rDNA를 분석한 결과, M. verrucosus CBS 210.61의 ITS (KX923951) 및 LSU rDNA (MH869590)이 각각 98.94%와, 99.66% 상동성을 보였다. ITS와 LSU rDNA서열을 유합하여 계통수를 그린 결과, DUCC15088은 M. verrucosus CBS 210.61과 같은 계통을 형성하였다 (Fig. 6). 형태학적 비교 및 분자유전학적 분석을 통한 계통수 결과에 따라 DUCC15088 (NIBRFGC000508314)를 국내 미기록종인 Microascus verrucosus로 동정하였다.
Fig. 6
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) nucleotide sequences of Microascus verrucosus DUCC15088 (NIBRFGC000508314). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium chrysogenum CBS 412.69 was used as an out-group.
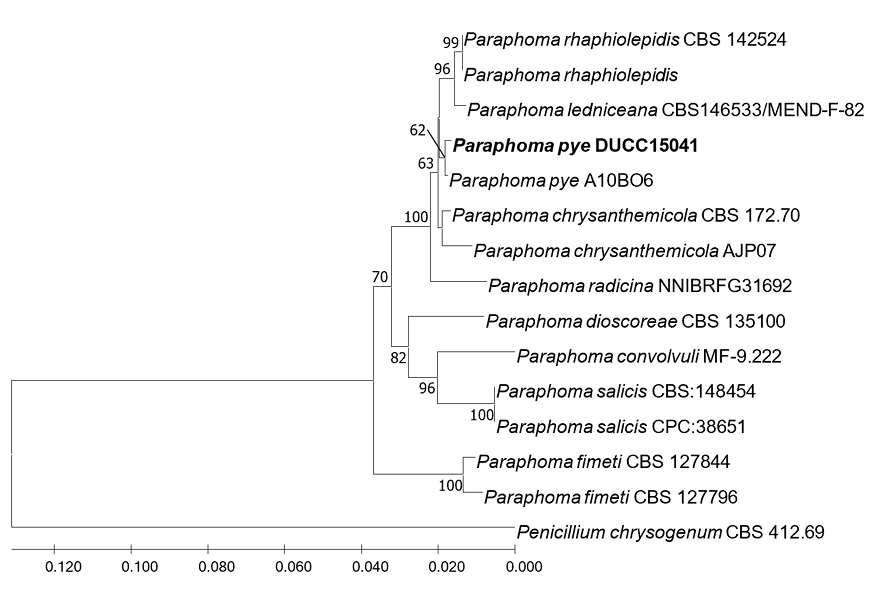
Paraphoma pye A. Moslemi & P.W.J. Taylor, Plant Pathology 67(1): 131 (2017) [MB#819470]
DUCC15041 (NIBRFGC000508312) 균주는 PDA 배지에서 7일간 배양하였을 때 균총의 직경은 약 31-32 mm 정도였으며 배지에서 약 0.3 cm정도 융기된 중앙이 짙은 카키색이며 외곽으로 갈수록 연한 카키색을 나타내다가 하얀 외곽선을 가지는 형태를 보였다 (Fig. 1D). MEA 배지에서는 융기되지 않은 중앙이 밝은 갈색이고 짙은 갈색의 외곽선을 가지는 균총을 형성하였다 (Fig. 2D). 약 2 μm 정도의 얇은 균사부터 4-5 μm 정도의 두꺼운 균사가 존재하였으며, conidia와 conidiophore형성하지 않았으나 8.3-12.5×3.1-4.6 μm 크기의 chlamydospore를 형성하였다 (Fig. 3D). P. pye는 들국화의 잎과 꽃에 감염을 일으켜 피해를 줄 수 있다고 보고되어 있다 [16]. P. pye를 포함하는 Paraphoma spp.는 Phoma-like fungi로써 현재 형태학적 식별에 상당한 어려움이 있어 종 구분에는 주로 분자유전학적 동정이 사용된다 [17]. DUCC15041 균주의 ITS 및 LSU rDNA 분석한 결과, P. pye A10BO6의 ITS (OQ274950) 및 LSU rDNA (OQ746386)는 각각 99.47, 100%의 상동성을 보였다. ITS 및 LSU rDNA서열을 유합하여 계통수를 그린 결과, DUCC15041은 P. pye A10BO6와 같은 계통을 형성하였다 (Fig. 7). 형태학적 비교 및 분자유전학적 분석을 통한 계통수 결과에 따라 DUCC15041 (NIBRFGC000508312)를 국내 미기록종인 Paraphoma pye로 동정하였다.
Fig. 7
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) nucleotide sequences of Paraphoma pye DUCC15041 (NIBRFGC000508312). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium chrysogenum CBS 412.69 was used as out group.
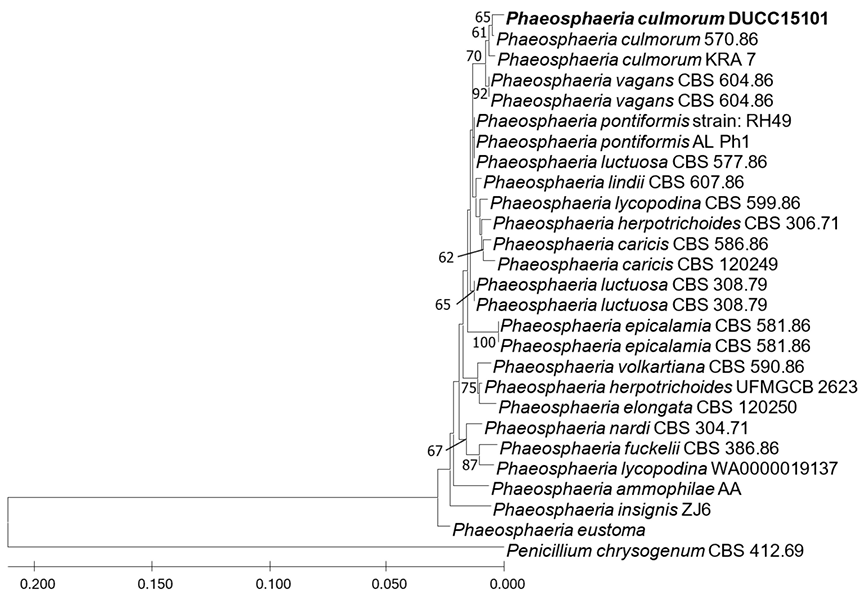
Phaeosphaeria culmorum (Auersw.) Leuchtm., Sydowia 37: 113 (1985) [MB#114984]
DUCC15101 (NIBRFGC000508302) 균주는 PDA 배지에서 7일간 배양하였을 때 균총의 직경은 56-57 mm 정도였으며, 약 2 cm마다 하얀색 테두리를 가지는 융기된 회색 균총을 형성하였다 (Fig. 1E). MEA 배지에서는 융기된 하얀색의 균사가 아래의 갈색 균사를 덮고 있는 형태의 균총을 형성하였다 (Fig. 2E). 현미경 관찰 결과 균사의 두께는 1-2 μm 정도였다 (Fig. 3E). 원기재문에선 P. culmorum이 100-180 μm의 자낭각, 55-90×13-18 μm 크기의 자낭, 20-29×6.5-8 μm 크기의 자낭포자를 가진다고 서술되어 있었지만 [18] DUCC15101에선 유성세대 구조를 찾을 수 없었다. P. culmorum을 포함하는 Phaeosphaeria 속은 pyrazine alkaloids, isocoumarins, perylenequinones, anthraquinones, diterpenes, and cyclic peptides을 포함하는 다양한 대사 물질을 생성하는 것으로 알려져 있어 그 가치가 높이 평가되고 있다 [19]. 포자를 형성하지 않아 형태학적으로는 비교하기 어려웠지만 ITS 및 LSU rDNA로 분자유전학적 동정을 진행한 결과 DUCC15101 균주는 P. culmorum CBS 570.86의 ITS (MH861992) 및 LSU rDNA (MH873682)와 각각 99.06, 99.88% 상동성을 보였다. 두 서열을 유합하여 계통수를 그렸을 때 DUCC15101은 P. culmorum CBS 570.86과 같은 계통을 형성하였다 (Fig. 8). 형태학적 비교 및 분자유전학적 분석을 통한 계통수 결과에 따라 DUCC15101 (NIBRFGC000508302)를 국내 미기록종인 Phaeosphaeria culmorum 로 동정하였다.
Fig. 8
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) nucleotide sequences of Phaeosphaeria culmorum DUCC15101 (NIBRFGC000508302). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium chrysogenum CBS 412.69 was used as out group.
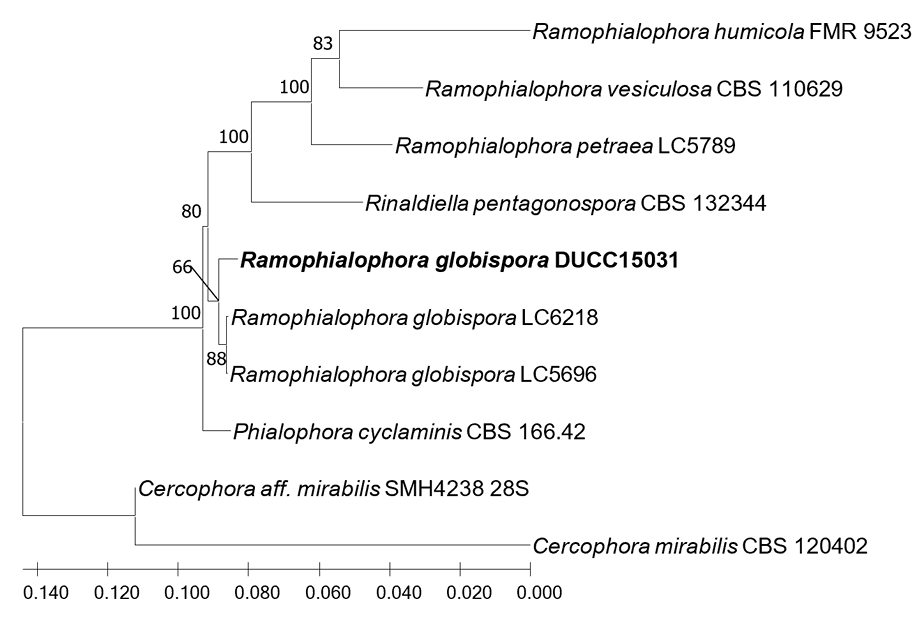
Ramophialophora globispora Z.F. Zhang, F. Liu & L. Cai, Persoonia 39: 24 (2017) [MB#818258]
DUCC15031 (NIBRFGC000508310)로 명명된 이 균주는 PDA 배지에서 7일간 배양하였을 때 균총의 직경은 약 18-19 mm 정도이었고 배지에서 약 0.2 cm 정도 융기된 전체적으로 회색의 균총을 형성하였고 배지는 초록색을 나타내었다 (Fig. 1F). MEA에서는 푹신푹신한 느낌의 하얀색 균사가 짙은 초록색 균사를 덮고 있는 형태의 균총을 형성하였다 (Fig. 2F). 광학현미경으로 관찰한 결과 chlamydospore로 보이는 4-5×3-4 μm 크기의 구조를 관찰하였다. 균사는 전체적으로 초록색을 띠며 두께는 약 2-3 μm였고 격벽이 존재하지 않았다 (Fig. 3F). 원기재문의 형태와 비교하였을 때 2-3 μm의 포자를 만든다고 서술되어 있었지만 [20] DUCC15031에선 포자를 관찰하지 못하였다. R. globispora가 속하는 Ramophialophora 속은 세계적으로 5종만이 존재하며 Sordariales의 새로운 무성세대 속으로 2004년에 보고되었다 [21]. 이 속에 대해서는 아직 연구보고가 부족한 실정이다. DUCC15031의 ITS 및 LSU rDNA를 분석한 결과, R. globispora LC5696의 ITS (KU535834) 및 LSU rDNA (MH867608)와 각각 99.56, 99.66% 상동성을 보였다. 이들 서열을 유합하여 계통수를 그린 결과, DUCC15031은 R. globispora LC5696과 같은 계통을 형성하였다 (Fig. 9). R. globispora와 형태적으로는 차이점이 존재하지만 분자유전학적으로 같은 계통을 형성하였기에 DUCC15031 (NIBRFGC000508310)을 국내 미기록종인 Ramophialophora globispora로 동정하였다.
Fig. 9
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and 28S ribosomal DNA gene (LSU rDNA) nucleotide sequences of Ramophialophora globispora DUCC15031 (NIBRFGC000508310). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Cercophora mirabilis was used as outgroup.
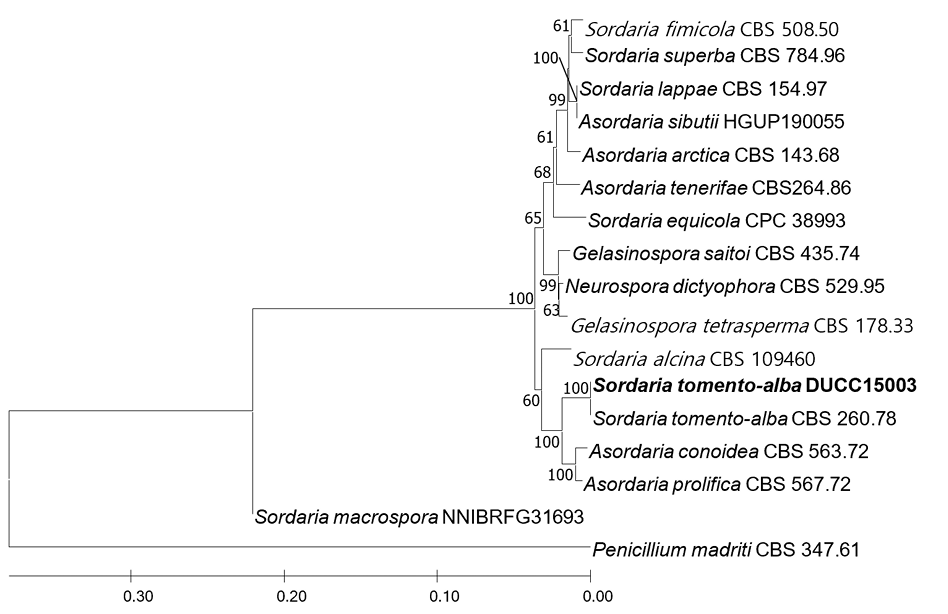
Sordaria tomentoalba Cailleux, Bulletin de la Société Mycologique de France 87: 483, 488, 499 (1971) [MB#323634]
DUCC15003 (NIBRFGC000508307) 균주는 PDA 배지에서 5일간 배양하였을 때 균총의 직경은 90 mm 이상이었고 배지를 꽉 채우는 하얀색 균총을 형성하였다 (Fig. 1H). MEA에서는 PDA에서와 같이 빠르게 생장하였으나 중앙 부분이 상대적으로 비어있게 균총을 형성하였다 (Fig. 2H). 현미경으로 관찰했을 때 균사의 두께는 3-4 μm 정도로 두꺼웠으며 약 20-25 μm 정도 크기의 균사가 뭉쳐 있는 구조를 형성하였다 (Fig. 3H). Conidia 또는 conidiophore는 관찰되지 않았다. S. tomentoalba가 속하는 Sordaira 속은 일반적으로 초식 동물의 배설물에서 발견되며 [22], 세계적으로 약 260여 종이 MycoBank에 등록되어 있으나 대부분 1900년도 초 또는 1800년도에 분리되어 자세한 정보가 거의 부재하였다. S. tomentoalba 또한 1971년에 서술되었다는 것 [23]을 제외하면 정보가 부재한 실정이기 때문에 형태학적인 비교를 수행하지 못하였다. DUCC15003의 ITS 및 BenA를 분석한 결과, S. tomentoalba CBS 260.78의 ITS (AY681195)및 BenA (AY681229)와 99.81, 100% 상동성을 보였다. 이 두 서열을 유합하여 계통수를 그린 결과, DUCC15003은 S. tomentoalba CBS 260.78과 같은 계통을 형성하였다 (Fig. 10). 따라서 DUCC15003 (NIBRFGC000508307)을 국내 미기록종인 Sordaria tomentoalba로 동정하였다.
Fig. 10
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and BenA nucleotide sequences of Sordaria tomento-alba DUCC15003 (NIBRFGC000508307). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium madriti CBS 347.61 was used as outgroup.
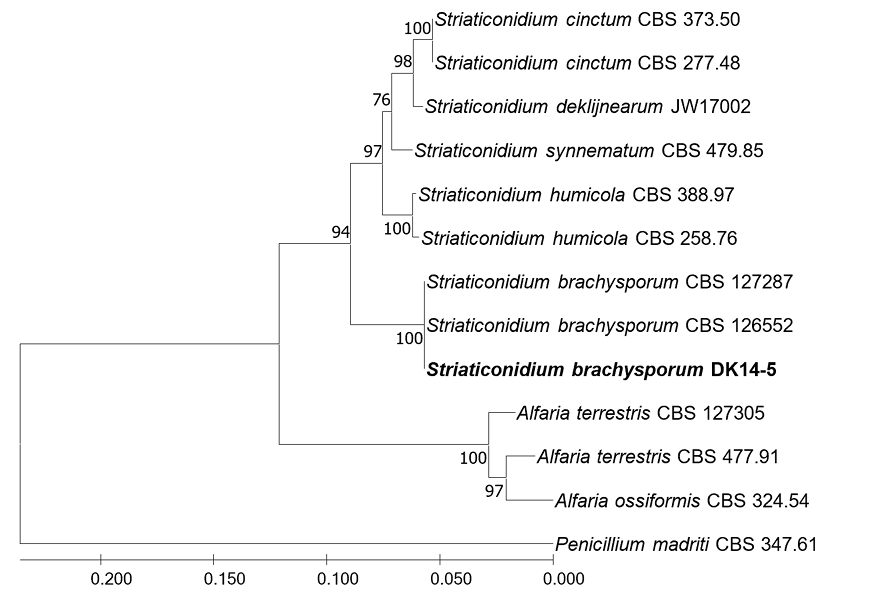
Striaticonidium brachysporum (Nicot) L. Lombard & Crous, Persoonia 36: 228 (2016) [MB#816043]
DK14-5 (NIBRFGC000505480) 균주는 PDA에서 7일간 배양하였을 때 균총의 직경은 39-40 mm 정도였으며 위에서 보았을 때 푹신해 보이는 하얀색의 균총을 형성하였다 (Fig. 1H). 균총의 뒷면은 아이보리 색을 나타냈으며 균사가 중앙에서부터 방사형으로 뻗어 나가는 형태를 보였다 (Fig. 1h). 광학현미경 관찰 결과 약 6-7 μm의 좁쌀 모양의 conidia를 관찰하였다 (Fig. 3H). 이러한 형태적 특징은 원기재문에 서술되어 있는 conidia ((6-) 6.5-7.5 (-9.5)×(2-) 2.5-3.5 (-5))와 유사하였다 [24]. Striaticonidium 속은 세계적으로 MycoBank에 5종만 등록되어 있다. S. brachysporum은 1961년에 처음 Myrothecium brachysporum으로 분리되었으나 2016년 현재의 종명으로 바뀌었다 [24,25]. 최근에는 사과나무에 궤양을 일으키는 것으로도 알려졌다 [26]. DK14-5 균주의 ITS 및 BenA를 분석한 결과, ITS는 S. cinctum과 S. brachysporum 둘 다 100% 상동성을 보여 뚜렷하게 구분되지 않았다. BenA는 S. brachysporum CBS 127287 (KU847324)와 100% 상동성을 보였다. ITS과 BenA 서열을 유합하여 계통수를 그렸을 때 DK14-5는 S. brachysporum CBS 127287 과 같은 계통을 형성하였다 (Fig. 11). 형태학적 및 분자유전학적 계통수 결과에 따라 DK14-5 (NIBRFGC000505480)를 국내 미기록종인 Striaticonidium brachysporum으로 동정하였다.
Fig. 11
Neighbor joining phylogenetic tree based on combined internal transcribed spacer (ITS) and BenA nucleotide sequences of Striaticonidium brachysporum DK14-5 (NIBRFGC000505480). The number of nodes represents the reliability value through 1,000 bootstrap repetitions, and those below 60% were removed. Fungal strain isolated in this study is indicated in bold. Penicillium madriti CBS 347.61 was used as outgroup.
과수화상병은 사과와 배나무에서 세균성 궤양을 일으키는 병해로서 피해가 큰 바 식물검역상 국내에서는 금지급 병원체로 관리되고 있다. 급격한 확산을 방지하기 위해 토양에 매몰 방제 처리하였기에 방제의 안전성을 확보하기 위해서는 이병식물체의 신속한 부숙 진행이 요구된다. 신속한 부숙은 과수화상병 유발 원인균인 Erwinia amylovora의 생존 가능성을 낮게하기 때문이다.
이에 따라 토양에 목질성 배나무의 부숙에 관여하는 미생물로서 리그닌, 펙틴, 셀룰로즈, 등의 난분해성 고분자 화합물을 분해할 수 있는 진균 존재의 중요한 정보이다. 본 연구에서 분리된 8종의 진균 중 리그닌 분해 능력이 뛰어난 담자균류에 속하는 종은 분리되지 않았다. 8종 모두 분류학상 자낭균류에 속하는 균이었다. 다만 이들 중 식물에 병을 일으키는 것으로 알려진 2종의 진균으로는 Paraphoma pye 와 Striaticonidium brachysporum 가 존재하였다. S. brachysporum 는 사과나무에 궤양을 일으키는 것으로 알려진 바 목질 성분 분해 능력이 있을 것으로 추정된다. 동시에 이 균은 배나무에도 감염하여 문제가 될 소지가 있으므로 향 후 지켜보아야 할 균으로 사료된다. 또한 이 종이 속하는 Striaticonidium 속에는 5종만 알려져 있는 바 S. brachysporum는 희귀 종으로서도 가치가 있다고 사료된다. 희귀 종 관점에서 보면 Ramophialophora globispora 도 세계적으로 5종만이 존재하기 때문에 본 연구에서 미기록종으로 발굴은 의미가 크다고 볼 수 있다. Microascus verrucosus는 인체병원성 균인데 3미터 깊이의 토양에서 나온 것 볼 때 토양에서 장기 생존이 가능할 것으로 보이며 아직 국내에서 임상적으로 분리되지 않은 점을 고려하면 토양 유래 감염이 있을 수 있음을 보여준다. 나머지 미기록 종에 대해서는 아직 알려진 생물학적 정보가 많지 않기 때문에 좀 더 연구가 필요하다.
적요
식물병원 세균 Erwinia amylovora 의해 발생하는 과수화상병은 사과, 배 등 장미과에 속하는 식물에 심한 피해를 주고 있다. 병의 빠른 확산을 막기 위해 국내에서는 병이 발생한 사과나무와 배나무에 대해 매몰하는 병관리를 실시해 왔다. 방제방법의 안전성을 입증하기 위해 매몰한 이병식물과 이병식물 주변의 토양을 조사하였다. 매몰토양 미생물군 조사과정에서 Bisifusarium allantoides, Botryotrichum Domesticum, Microascus verrucosus, Paraphoma pye, Phaeosphaeria culmorum, Ramophialophora globispora, Sordaria tomentoalba, Striaticonidium brachysporum 등이 국내 미기록종으로 확인되었다. 이에 이들 8종 진균에 대한 형태학적, 분자유전학적 특성을 보고한다.


