표고버섯은 약용물질로서 항암작용이 있는 lentinan과 혈중 콜레스트롤의 함량을 낮추는 eritadenine 등을 포함하고 있으며[1-3] 다이어트에도 도움이 되는 버섯이다[4]. 또한 탄수화물, 단백질, 지방, 비타민, 미네랄 등 다양한 성분을 함유하고 있는[5] 맛과 풍미가 뛰어난 식용버섯이다. 이렇듯 맛과 생물학적 기능을 담고 있는 표고버섯은 재배하는 임가의 증가에 더하여 재배면적, 생산량이 매년 증가하고 있다[6]. 표고버섯 생산은 현재 원목재배와 톱밥배지를 이용하고 있다. 최근에는 재배자의 노령화에 따라 원목재배보다 톱밥배지를 이용한 버섯 생산을 선호하고 있다. 기존에 발표된 논문에 의하면 원목재배와 톱밥배지를 이용하여 표고버섯을 재배하는 재배사 내의 환경에서 다양한 미생물 상이 존재하는 것으로 보고되었다[7-10]. 표고버섯 재배사 내 환경에서 분리된 진균들은 대체로 포자가 바람에 의해 쉽게 부유하여 전파되기 쉬운 종들이었고 이중 몇몇 종은 인체에 유해한 종으로 보고되었다. 더불어 국내에 보고되지 않았던 미기록 진균들도 발굴되었다. 이에 따라 본 연구에서는 지속적으로 미기록 진균을 발굴하기 위해서 표고버섯 재배사의 실내공기로부터 진균 모니터링을 수행하여 진균을 포집하고 분리하였다. 분리된 진균을 동정한 결과 국내에 보고되지 않은 미기록 진균 6종이 검출되어 이들에 대해 보고하고자 한다.
공기시료 채취는 2016년 4월 전라남도 장흥군, 5월 경상북도 구미시, 경상남도 거제시, 6월 경기도 여주시 소재의 원목으로 버섯을 재배하는 임가 4곳 (Fig. 1A)과 톱밥으로 버섯을 재배하는 임가 1곳(2016년 6월 충청남도 청양군) (Fig. 1B)의 버섯재배사 내서 실내공기를 포집하였다. 실내공기 채집 방법은 ISO 16000-18에 기반한 충돌법에 따라 Andersen sampler (KAS-110, Kemik Corporation, Seongnam, Korea)를 사용하여 28.3 L/min 유량으로 1분간 포집하였다[8-10]. 공기 채집은 표고버섯 재배사의 가운데 지점에서 진행하였고 1.3 m 높이에서 수평을 맞추어 수행하였다. 배양 배지로서는 암피실린(ampicillin)을 100 μg/mL 농도로 첨가한 malt extract agar (Difco, Detroit, MI, USA)를 사용하였다. 실내공기를 채집한 배지를 5일 동안 25℃ 배양기에서 배양하였고, 자라난 진균은 단포자 분리를 통해 순수 분리하였다. 형태적 동정을 위해서는 PDA에 순수 배양된 진균의 포자와 균사를 대상으로 광학현미경 (Axioskop40; Carl Zeiss, Oberkochen, Germany)을 이용하여 미세구조를 관찰 기록하였다. 배지에 나타난 균총의 형태는 육안으로 관찰하고 사진으로 촬영 기록하였다. 분자생물학적 동정을 위해서는 Kim의 drilling 방법[11]을 참조하여 진행 후 Qiagen사의 Plant genomic DNA extraction kit(69106, Germany)을 이용하여 genomic DNA를 추출하였다. 1차 동정으로 internal transcribed spacer (ITS) rDNA region을 증폭하기 위해서는 ITS1 (5'-TCCGTAGGTGAACCTGCG-3')/ ITS4 (5'-TCCTCCGCTTATTGATATGC-3')[12] 프라이머를 이용하여 중합효소연쇄반응 (polymerase chain reaction, PCR)을 수행하였다. PCR 증폭된 DNA 산물은 1% 아가로스겔에서 전기영동을 수행하여 기대되는 밴드의 크기를 확인한 후 NAVIGenTM PCR Purification Kit (NAVIBIOTECH, Korea)를 사용하여 정제하고 마크로젠사 (Seoul, Korea)에 의뢰하여 염기서열을 분석하였다. 분석된 염기서열의 분자동정을 위해서는 미국 National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov)의 웹에 있는 BLAST 프로그램을 사용하여 DNA 데이터베이스에 등록되어 있는 진균들의 DNA와 상동성을 비교하였다. ITS rDNA region 염기서열 결과를 1차적으로 검토 후 알려진 진균 종과 염기서열의 상동성이 낮은 균주는 논문검색을 통하여 그 종이 속한 속을 동정할 때 사용된 여러 다른 유전자 특이적 프라이머를 이용하여 증폭하였다. 사용된 증폭용 특이적 프라이머로는 28S를 증폭하는 프라이머 LR0R (5'-ACCCGCTGAACTTAAGC)/ LR7 (5'-GCAGATCTTGGTGGTAG-3')[13], β-tubulin gene을 증폭하는 프라이머 BT12 (5'-GTTGTCAATGCAGAAGGTCTC-3')/ T10 (5'-ACGATAGGTTCACCTCCAGAC-3')[12], translation elongation factor 1 a (tef-1a) gene을 증폭하는 프라이머 TEF728 (5’-CATCGAGAAGTTCGAGAAGG-3’)/ TEF1 (5’-GCCATCCTTGGAGATACCAGC-3’)[14]를 이용하였다. 계통분석을 위해서는 분리 균주와 관련된 taxon의 염기서열을 NCBI의 GenBank에서 다운받아 다중정렬 한 후 MEGA 6 프로그램[15]을 이용하여 염기서열의 유사도 및 phylogenetic analysis를 수행하였다. 계통도는 neighbor-joining 방법[16]으로 분석하였고 계통도 가지의 clade 신뢰도는 1,000번의 bootstrap resampling을 수행하여 분석하였다. 동정된 진균 중 미기록 균주인 DK12-2, DK12-3, DK12-4, DK12-5, DK12-6, DK12-7번은 국립생물자원관(National Institute of Biological Resource)에 기탁하여 등록번호를 받았다. DK12-2는 NIBRFGC000499879, DK12-3은 NIBRFGC000499880, DK12-4는 NIBRFGC000499881, DK12-5는 NIBRFGC000499882, DK12-6은 NIBRFGC000499883, DK12-7은 NIBRFGC000499884번으로 등록번호를 받았다. 또한 이들 균주의 분석된 염기서열은 NCBI의 GenBank DNA database에 등록하였고 등록번호는 Table 1에 제시하였다.
균주 DK12-2 (Cenangium acuum)
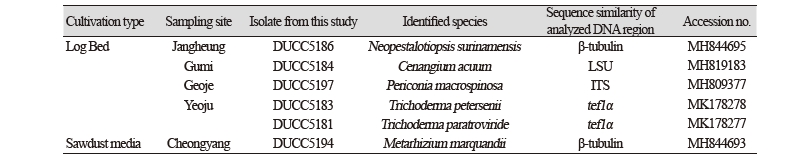
2016년 5월 경상북도 구미시에 위치한 표고 원목 재배사 내 실내공기에서 분리한 균을 PDA에서 10일간 배양하였을 때 흰색의 균사체로 생장하며 중앙에 균사가 올리브색으로 부분적으로 변하였다(Fig. 2-A). 광학현미경으로 관찰하였을 때, 균사 끝에 5 - 7 × 2.5 - 3 μm 크기의 곤봉 모양의 포자를 가지고 있었다(Fig. 3-A). 1차적으로 ITS region을 분석하였을 때 Cenangium 속으로 동정이 되었으며, 추가로 LSU rDNA region을 분석한 결과 Cenangium accuum Cooke & Peck, Grevillea (KX090828)과 98% 상동하였다(Fig. 4-A). Cennangium종 중에 C. ferruginosum은 소나무류의 피목가지마름병을 유발시키는 것으로 알려져 있다[17].
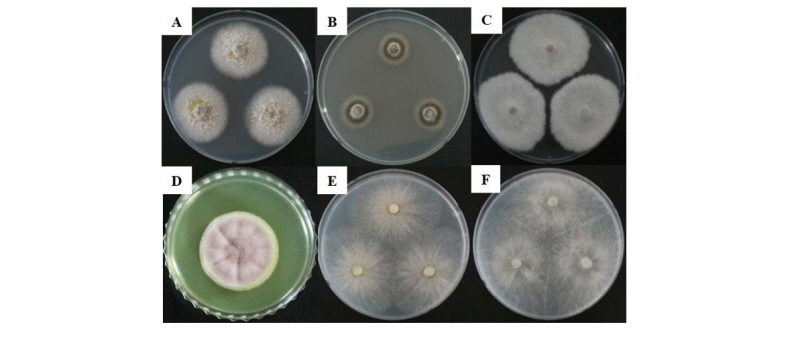
Fig. 2. Unrecorded fungal species isolated from mushroom cultivation houses in this study. The fungi were grown on potato dextrose agar at 25℃ for 7 days. A, Cenangium acuum; B, Periconia macrospinosa; C, Neopestalotiopsis surinamensis; D, Metarhizium marquandii; E, Trichoderma petersenii; F, Trichoderma paratroviride.
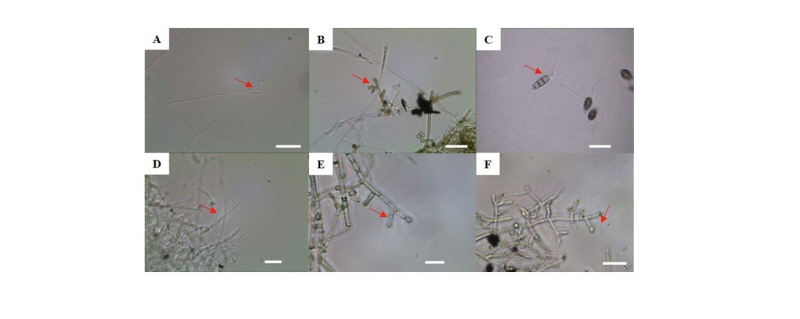
Fig. 3. Microscopic image of unrecorded fungal species isolated from mushroom cultivation houses in this study. A, Cenangium acuum; B, Periconia macrospinosa; C, Neopestalotiopsis surinamensis; D, Metarhizium marquandii; E, Trichoderma petersenii; F, Trichoderma paratroviride. Red arrow is important feature (scale bar = 2 μm).
균주 DK12-3 (Periconia macrospinosa)
2016년 5월 경상남도 거제시의 표고 원목 재배사 내 실내공기에서 분리한 균을 PDA에서 6일간 배양하였을 때 균사가 동심 윤문을 그리며 생장하였으며 짙은 카키색 또는 녹회색의 균사로 생장하였다(Fig. 2-B). 광학현미경으로 관찰한 결과 균사 끝에 1 – 1.5 × 1 – 1.5 μm 크기의 구형 모양의 포자가 체인형태로 존재하였다(Fig. 3-B). ITS region을 분석한 결과 Periconia macrospinosa Lefebvre & Aar.G. Johnson (JQ781723)과 100% 상동하였다(Fig. 4-B). Periconia macrospinosa균은 염소를 포함한 대사과정에서 생합성을 하는 균으로 보고되었으며[18], 미소균핵과 후막포자를 형성하는 균으로 알려져 있다[19].
Fig. 4-1. Phylogenic relationships of the six fungal isolates inferred by the neighbor joining analysis based on 28S rDNA (A), internal transcribed spacer rDNA (B, D), β-tubulin gene (C) or the translation elongation factor 1a gene (E, F) sequences. A, Cenangium acuum (NIBRFGC000499879); B, Periconia macrospinosa (NIBRFGC000499880); C, Neopestalotiopsis surinamensis (NIBRFGC000499881);
균주 DK12-4 (Neopestalotiopsis surinamensis)
2016년 4월 전라남도 장흥군의 표고 원목 재배사 내 실내공기에서 분리한 균은 PDA에서 6일간 배양하였을 때 흰색의 균사체가 조밀하며 솜처럼 약간 부유하여 생장하였다(Fig. 2-C). 광학현미경 관찰하였을 때 2개의 편모와 3개의 격벽을 가지고 있는 7 - 8 × 3 – 3.5 μm 크기의 포자를 확인하였다(Fig. 3-C). 1차적으로 ITS region을 분석하였을 때 Neopestalotiopsis속으로 동정되었으며, β-tubulin region을 분석하여 Neopestalotiopsis surinamensis Maharachch., K.D. Hyde & Crous (KM199465)와 100% 상동하였다(Fig. 4-C). Neopestalotiopsis속의 다른 종들은 토마토에 무름병[20] 또는 포도덩굴의 잎에 반점을 야기시키는 것으로 알려져 있다[21].

Fig. 4-2. Phylogenic relationships of the six fungal isolates inferred by the neighbor joining analysis based on 28S rDNA (A), internal transcribed spacer rDNA (B, D), β-tubulin gene (C) or the translation elongation factor 1a gene (E, F) sequences. D, Metarhizium marquandii (NIBRFGC000499882); E, Trichoderma petersenii (NIBRFGC000499883); F, Trichoderma paratroviride (NIBRFGC000499884).
균주 DK12-5 (Metarhizium marquandii)
2016년 6월 충청남도 청양군에 있는 표고 톱밥 재배사 내 실내공기에서 분리한 균을 PDA에서 6일간 25℃에서 배양하였을 때 아래 Fig. 2-D와 같이 생장이 매우 느렸다. 균사는 연보라색 또는 핑크색의 균사로 생장하였다. 광학현미경으로 균사를 관찰한 결과 분생포자경 정단에 0.5 - 1 × 0.5 - 1 μm 크기의 구형 모양의 작은 분생포자를 가지고 있었다(Fig. 3-D). 청양 톱밥 재배사 내 실내공기에서 분리한 균은 β-tubulin region을 분석하였을 때 Metarhizium marquandii (Massee) Kepler, S.A. Rehner & Humber (KJ398548)와 99% 상동하였다(Fig. 4-D). Metarhizium속의 M. viride는 카멜레온, 턱수염도마뱀 등에 진균증을 일으키는 종으로 보고되어있다[22].
균주 DK12-6 (Trichoderma petersenii)
2016년 6월 경기도 여주시의 표고 원목 재배사 내 실내공기에서 분리한 균을 potato dextrose agar (PDA)에서 3일 간 배양하였을 때 아이보리 또는 연한 회색의 균사가 빠르게 생장하였다(Fig. 2E). 광학현미경으로 균사를 관찰한 결과 삼지창 모양의 분생포자경 정단에 2~2.5 × 2~2.5 μm 크기의 구형 모양의 분생포자를 가지고 있었다(Fig. 3E). tef-1a region을 분석하였을 때 Trichoderma petersenii Samuels, Dodd & Schroers (FJ860670)과 100% 상동하였다(Fig. 4E). Trichoderma속은 기존 다른 논문에 의하면 표고버섯 재배사 내에서 자주 발견되는 진균이다[7, 23]. 본 T. petersenii 와 더불어 다음의 T. paratroviride 균에 대한 정보는 아직 분류학적 정보를 제외하고는 연구된 바가 없다. 본 연구에서도 표고버섯 재배사에서 분리된바 향후 버섯재배에 대해 어떠한 영향을 줄지에 대한 연구가 시급히 필요하다고 생각된다.
균주 DK12-7 (Trichoderma paratroviride)
2016년 6월 경기도 여주시의 표고 원목 재배사 내 실내공기에서 분리한 균을 PDA에서 3일 간 배양하였을 때 아이보리 또는 연한 회갈색의 균사가 빠르게 생장하였다(Fig. 2F). 광학현미경으로 균사를 관찰한 결과 삼지창 모양의 분생포자경 정단에 3~3.5 × 3~3.5 μm 크기의 구형 모양의 분생포자를 가지고 있었다(Fig. 3F). tef-1a region을 분석하였을 때 Trichoderma paratroviride Jaklitsch & Voglmayr (KT149301)과 99% 상동하였다(Fig. 4F).
적요
2016년 4월부터 6월까지 표고 원목재배 임가 4곳과 표고 톱밥재배 임가 1곳의 재배사 내 실내 공기에서 진균을 포집 및 분리 동정하였다. 총 6개의 국내 미기록 진균(Cenangium acuum, Periconia macrospinosa, Neopestalotiopsis surinamensis, Metarhizium marquandii, Trichoderma petersenii, Trichoderma paratroviride)을 분리하였으며 이들 균류에 대하여 형태학적 특성 및 internal transcribed spacer rDNA region, large subunit region, β-tubulin 또는 translation elongation factor 1α유전자 염기서열에 기반하여 계통학적 분석 결과를 기술하였다.