서론
Sordariomycetes은 자낭균문(Ascomycota)에서 두 번째로 큰 분류군으로 32목 107과가 포함된다 [1]. Sordariomycetes은 지의류를 형성하지 않고, 플라스크 형태의 자낭과를 가지며, 원시적인 자낭벽을 갖거나 하나의 자낭벽을 갖는 자낭을 형성한다[2]. Sordariomycetes에 속하는 대부분의 분류군은 육상 환경에서 분포하지만, 몇몇 분류군은 수중에서 서식할 수 있으며, 병원균이나 내생균으로 식물 내부에서 분리되기도 한다[3]. Sordariomycetes에 속하는 분류군 중 Colletotrichum 속, Diaporthe 속, Chaetomium 속은 내생균으로 빈번하게 발견되며, Neurospora 속도 내생균으로 보고된 바 있다[4].
내생균(Endophytic fungi)은 식물의 조직 내에서 서식하며 식물에게 부정적인 영향을 나타내지 않는 곰팡이이다[5]. 내생균은 식물에게 생물학적, 비생물학적 스트레스에 대한 내성을 제공하며, 내생균이 생성하는 항균 물질은 병원균으로부터 식물을 보호한다[6]. 내생균이 생성하는 효소와 이차 대사산물은 농업과 산업에서 활용 가능성이 있으며, 바이오 연료, 생물 정화 등의 분야에도 활용하기 위한 연구가 수행되고 있다[7]. 따라서 내생균에 대한 연구는 곰팡이와 식물의 관계를 파악하고 새로운 미생물 자원을 발굴할 가능성을 높인다. 본 연구는 국내 내생균을 분리하던 중 확인된 Sordariomycetes에 속하는 4속 5종의 국내 미기록 내생균에 대한 형태적 특징을 기술하고 분자계통학적 결과를 보고하고자 한다.
재료 및 방법
시료 채집과 균주 분리
시료의 채집은 2021년 9월부터 2023년 4월까지 진행하였다. 충청남도 보령시, 충청남도 태안군, 경상북도 경주시, 경상북도 울진군, 대전광역시에서 목본 식물의 잎과 줄기를 채집하여 24시간 이내에 실험실로 운반하였다.
식물 잎과 줄기 시료는 증류수로 세척한 후 1% NaClO 용액에 1분간 살균하고, 다시 70% 에탄올로 2분간 표면을 살균하였다. 살균이 완료된 시료들을 1.5 cm 크기로 잘라 potato dextrose agar (PDA; Difco Lab., Detroit, USA)에 치상하고, 시료의 내부에서부터 자란 균사를 계대배양하여 순수한 균주를 분리하였다.
형태적, 분자생물학적 분석
균주는 PDA배지를 이용하여 25℃, 암소에서 배양하였다. 균총의 형태는 PDA배지에서 7일간 배양한 후 관찰하였다. 미세구조의 관찰을 위해 slide culture 방법[8]을 이용하여 균주를 배양하였으며, Diaporthe 속 균주의 분생포자과(conidiomata) 형성을 유도하기 위해 소나무(Pinus densiflora Siebold & Zucc.) 잎을 멸균하여 2% water agar 배지에 균주와 함께 치상하였다[9]. 배양한 균주의 미세구조는 광학현미경(Axio Imager A1, Carl ZWISS, Overkochen, Germany)을 이용하여 관찰하였다.
균사에서 HiGene Genomic DNA Prep Kit (BioFACT, Daejeon, Korea)를 이용하여 DNA를 추출한 후, 각 속의 분자생물학적 동정을 위한 6개 DNA 지역을 Polymerase Chain Reaction (PCR)으로 증폭하였다. 증폭한 DNA 지역과 primer, PCR 조건은 Table 1에 기술하였다. 증폭된 DNA는 1.5% 아가로스 겔에서 20분간 전기영동하여 DNA 절편의 크기를 확인하고, Sanger 염기서열 분석을 의뢰하였다(SolGent, Daejeon, Korea). DNA 염기서열은 National Center for Biotechnology Information에서 제공하는 Basic Local Alignment Search Tool (BLAST)를 이용해 유사 서열을 탐색하였다. 동정을 위해 MEGA7 프로그램으로[18] DNA 서열을 결합하여 neighbor-joining 방법으로 계통수를 작성하였으며, bootstrap 방법을 이용해 1,000회 반복하여 계통수를 검증하였다. 미기록종 균주는 국립생물자원관(National Institute of Biological Resources, NIBR)에 기탁하였으며, 계통학적 분석에 이용한 DNA 염기서열은 NCBI에 등록하였다.
결과 및 고찰
Chaetomium subaffine Sergeeva ex X. Wei Wang & Houbraken, Studies in Mycology 101: 167 (2022) [MB#842311]
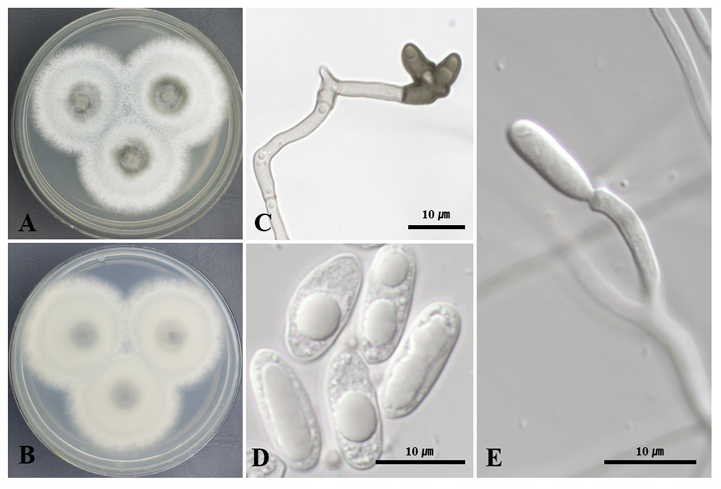
형태적 특징: PDA배지에서 25℃에서 7일간 배양했을 때 균총의 크기는 53.87-66.58 mm이고, 흰색 공중 균사가 발달하며, 배지 표면의 균총은 녹회색이고, 솜털 같은 질감을 나타낸다. 배지의 뒷면은 올리브색이며, 중앙으로 갈수록 점점 색이 진해진다(Fig. 1A and B). 분생포자경(conidiophore)은 분지된 균사의 끝에서 발달한다. 분생자(conidia)는 달걀 형태이고, 유리질이며, 세포벽의 질감은 부드럽다. 격벽(septate)이 없고, 사슬형태로 연결되며 사슬의 끝에서 말린 형태를 나타낸다. 분생자의 크기는 약 (2.72-) 3.19 (-3.59)×(1.77-) 2.17 (-2.53) μm (n=20)이다(Fig. 1D and E). 자낭과(ascomata)의 균사는 밝은 갈색이며, 돌기가 있고, 곧거나 휘어 있다(Fig. 1C). 완전히 발달한 자낭과와 자낭포자(ascospore)는 관찰되지 않았다.
Fig. 1
Morphological characteristics of Chaetomium subaffine KNUE 22N617. Colonies grown for 7 days on potato dextrose agar (PDA) (A, B), ascomatal hairs (C), conidium (D), conidiophore and conidia (E).

Specimen examined: Wonsando island, Boryeong-si, Chungcheongnam-do, Korea, 36° 23'05.2"N 126°25'30.2"E, April 8, 2022, isolated from leaf of Juniperus rigida, strain KNUE 22N617, NIBRFGC000510081, GenBank No. OQ271280 (ITS), OR794364 (LSU), OR805129 (TUB2) and OR805130 (RPB2).
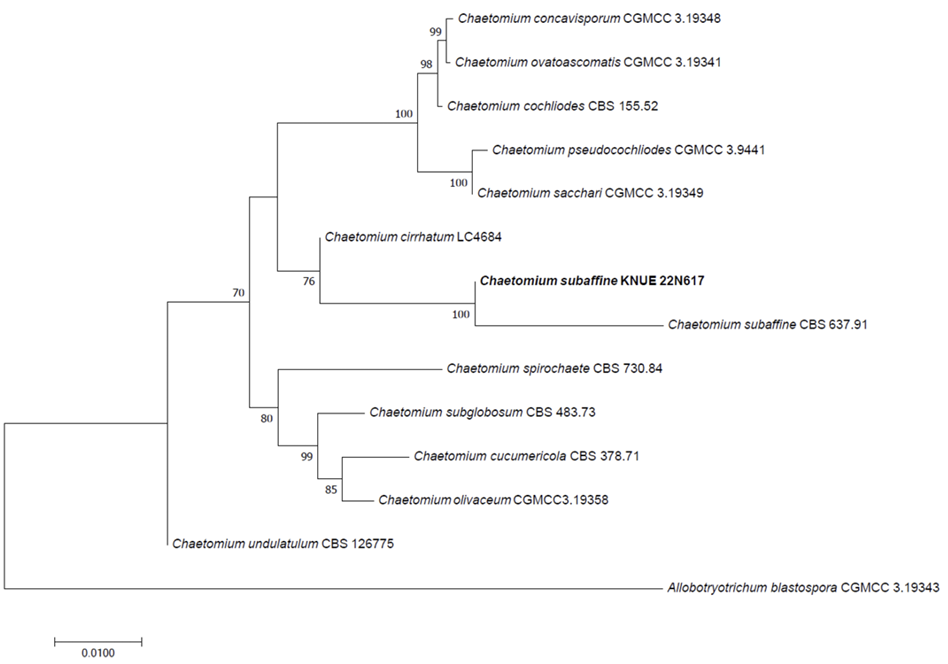
Notes: C. subaffine는 1961년에 Sergejeva에 의해 최초 보고되었으나 특징 기술의 부족으로 인해 2022년 Wang 등에 의해 다시 보고되었으며, 자낭과를 덮고 있는 흰색의 풍부한 균사체와 Acremonium 속의 무성포자와 유사한 형태의 분생자를 갖는다고 기술되었다[19,20]. 이는 C. subaffine KNUE 22N617의 형태적 특징과 일치하였다(Table 2). C. subaffine는 곡물에서 분리된 바 있으며[20], 식물 병원성 곰팡이에 대한 항생 효과가 보고되었다[21]. ITS, LSU, TUB2, RPB2 영역의 DNA 염기서열 분석 결과 ITS는 C. subaffine MN264617과 99.62%, LSU는 C. subaffine MH877811과 100%, TUB2는 C. subaffine MK026424와 99.50%, RPB2는 C. subaffine KF001817과 98.57% 일치하였으며, neighbor joining 계통수에서 C. subaffine CBS 637.91과 같은 그룹으로 형성되었다(Fig. 2).
Fig. 2
Neighbor-joining phylogenetic tree of Chaeotomium subaffine KNUE 22N617 based on a concatenated alignment of internal transcribed spacer (ITS), β-tubulin (TUB2), RNA polymerase Ⅱ second largest subunit (RPB2), and large subunit ribosomal RNA gene (LSU) sequences. Allobotryotrichum blastospora was used as an outgroup. Numbers on branches indicate bootstrap values (1,000 replicates) upper than 50%. Fungal strain isolated in this study is in a bold.
Colletotrichum jiangxiense F. Liu & L. Cai, Persoonia 35: 82 (2015) [MB#809161]
형태적 특징: PDA배지에서 25℃에서 7일간 배양했을 때 균총의 크기는 46.07-47.73 mm이고, 원형이며, 가장자리는 매끄럽고, 고도는 납작하다. 균총의 색은 중앙이 회녹색, 가장자리는 흰색을 띠며, 솜털 같은 질감을 나타낸다. 배지의 뒷면은 연한 회색 또는 아이보리색을 띠며, 가장자리는 흰색이다(Fig. 3A and B). 분생포자경은 가지를 치며 분지하고, 유리질이며, 분생자생성세포(conidiogenous cell)는 원통형이고, 유리질이다(Fig. 3E). 분생자는 원통형이고 양쪽 말단은 둥글며, 세포벽의 질감은 부드럽고, 격벽이 없다(Fig. 3D). 내부에 과립이 관찰되며 크기는 약 (11.06-) 12.95 (-15.29)×(4.53-) 5.50 (-6.91) μm (n=20)이다. 부착기는 불규칙하고, 나눠지지 않거나 2가닥으로 나눠지며, 검정색을 띠고, 세포벽은 두껍다(Fig 3C).
Fig. 3
Morphological characteristics of Colletotrichum jiangxiense KNUE 22N642. Colonies grown for 7 days on potato dextrose agar (PDA) (A, B), appressorium (C), conidia (D), conidiophore and conidiogenous cell (E).
Specimen examined: Anmyeondo island, Taean-gun, Chungcheongnam-do, Korea, 36°29'30.9"N 126°21'37.4"E, April 8, 2022, isolated from leaf of Daphniphyllum macropodum, strain KNUE 22N642, NIBRFGC000510044, GenBank No. OR085434 (ITS), OR826299 (TUB2), OR826297 (GAPDH) and OR826295 (CHS1).
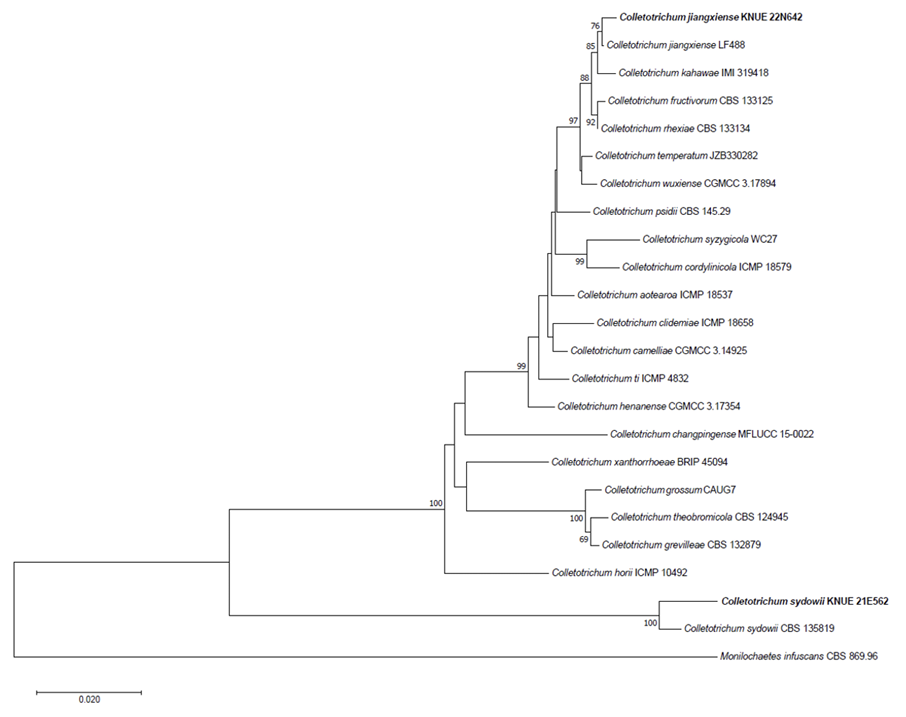
Notes: C. jiangxiense는 2015년에 Liu 등에 의해 최초 보고되었으며, 콜로니 가장자리가 평평하고 밀집된 공중 균사는 솜털 같은 질감에 백색에서 회색을 띠며 분생자는 투명하거나 무색으로 원통형, 매끄러운 세포벽과 양쪽 끝이 둥근 형태적 특징을 나타낸다[22]. C. jiangxiense KNUE 22N642도 같은 특징을 나타내었다(Table 3). 중국의 차나무에서 분리된 균주가 최초 보고되었으며[22], 고추와 아보카도에서 탄저병(anthracnose)를 일으키는 병원균으로 보고된 바 있다[23,24]. ITS, TUB2, GAPDH, CHS1 영역의 DNA 염기서열 분석 결과 ITS는 C. jiangxiense MN744288과 99.42%, TUB2는 C. jiangxiense KX009039와 100%, GAPDH는 C. jiangxiense OM684529와 100%, CHS1은 C. jiangxiense MZ457431과 99.60% 일치하였으며, neighbor joining 계통수에서 C. jiangxiense LF 488과 같은 그룹으로 형성되었다(Fig. 4).
Fig. 4
Neighbor-joining phylogenetic tree of Colletotrichum jiangxiense KNUE 22N642 and Colletotrichum sydowii KNUE 21E562 based on a concatenated alignment of internal transcribed spacer (ITS), glyceraldehyde 3-phosphate dehydrogenase (GAPDH), chitin synthase (CHS1), and β-tubulin (TUB2) sequences. Monilochaetes infuscans was used as an outgroup. Numbers on branches indicate bootstrap values (1,000 replicates) upper than 50%. Fungal strain isolated in this study is in a bold.
Colletotrichum sydowii Damm, Studies in Mycology 86: 152 (2017) [MB#820688]
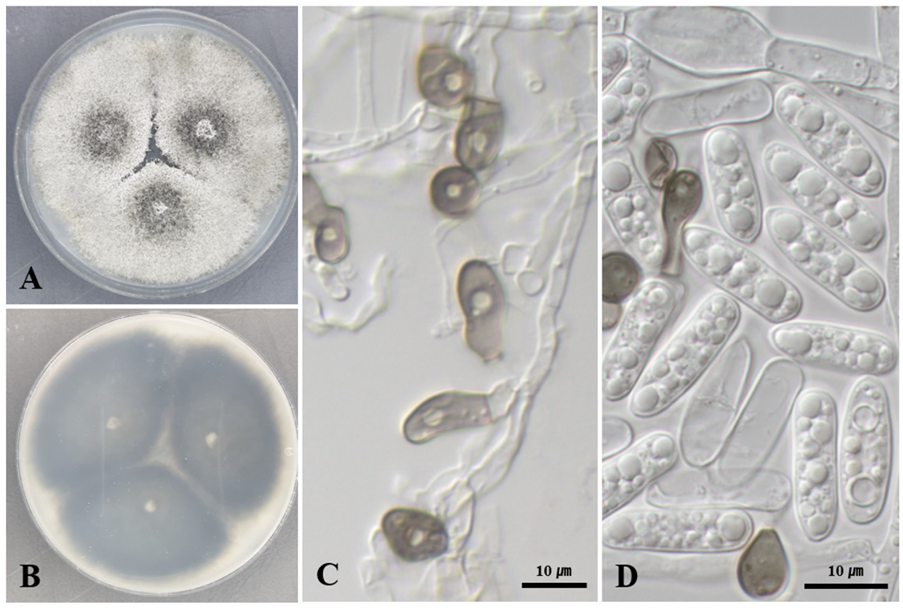
형태적 특징: PDA배지에서 25℃에서 7일간 배양했을 때 균총의 크기는 62.53-65.60 mm이고, 백색에서 회색을 가진 솜털 질감의 균사가 짙은 회색의 균사로 구성된 중앙부를 둘러싼 모습이 관찰되었다. 고도는 평평하고 가장자리는 불규칙하며 섬유상의 형태로 발달하였다. 배지의 뒷면은 짙은 회색이다(Fig. 5A and B). 검정색 또는 회색의 부착기가 관찰되었고(Fig. 5C), 분생자는 투명하고 원통형이며 양 끝이 둥근 모양으로 직경은 (13.50-) 17.09 (-18.52)×(4.87-) 5.52 (-6.40) μm (n=20)이다(Fig. 5D).
Fig. 5
Morphological characteristics of Colletotrichum sydowii KNUE 21N562. Colonies grown for 7 days on potato dextrose agar (PDA) (A, B), appressoria (C), conidia (D).
Specimen examined: Gyeongju-si, Gyeongsangbuk-do, Korea, 35°41'52.7"N 129°21'04.0"E, September 2, 2021, isolated from leaf of Albizia julibrissin, strain KNUE 21E562, NIBRFGC000509078, GenBank No. OR 805364 (ITS) OR826300 (TUB2), OR826298 (GAPDH) and OR826296 (CHS1).
Notes: C. sydowii는 2017년에 Marin-Felix 등에 의해 최초로 보고되었으며, 분생자가 투명하고 부드러운 세포벽을 가지며 원통형의 특징을 나타낸다[25]. C. sydowii KNUE 21E562도 동일한 특징을 나타내는 분생자가 관찰되었다(Table 4). 딱총나무 속의 잎에서 분리된 것이 최초로 보고되었으며[25], 이후 Bergenia ciliata의 잎에서 내생균으로 분리되었다[26]. ITS, TUB2, GAPDH, CHS1 영역의 DNA 염기서열 분석 결과 ITS는 C. sydowii ON631989와 99.82%, TUB2는 C. sydowii KY263793과 99.72%, GAPDH는 C. sydowii MZ330608과 100%, CHS1은 C. sydowii MZ330610과 99.19% 일치하였으며, neighbor joining 계통수에서 C. sydowii CBS 135819과 같은 그룹으로 형성되었다(Fig. 4).
Diaporthe vacuae S. Hilário, L. Santos & A. Alves, Mycologia 112 (2): 303 (2020) [MB#831440]
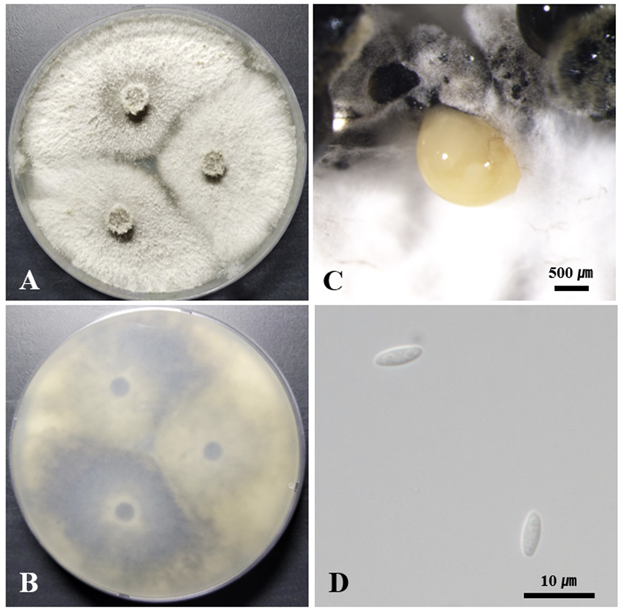
형태적 특징: PDA배지에서 25℃에서 7일간 배양했을 때 균총의 크기는 90 mm 페트리 접시를 모두 채웠으며, 중앙은 회색, 가장자리로 갈수록 흰색을 띤다. 균총의 고도는 배지에 납작하게 붙어 있으며 양털 같은 질감을 나타낸다. 배지 뒷면의 중앙은 짙은 회색 및 노란색이 관찰되며, 가장자리는 흰색을 띤다(Fig. 6A and B). 분생포자과는 연한 베이지에서 노란색을 띠며 구형이다(Fig. 6C). Alpha-분생자는 투명하고 매끄러운 타원형으로 크기는 약 (4.33-) 5.16 (-8.27)×(2.30-) 3.09 (-3.68) μm (n=20) 정도이다(Fig. 6D). Beta-분생자와 gamma-분생자는 관찰되지 않았다. 분생포자과(Conidiomata) 주변으로 검은색에서 짙은 살구색의 삼출물이 분비된다.
Fig. 6
Morphological characteristics of Diaporthe vacuae KNUE 23N743. Colonies grown for 7 days on potato dextrose agar (PDA) (A, B), conidiomata (C), alpha conidia (D).
Specimen examined: Mt. Gaphasan, Daejeon, Korea, 36°21'43.5"N 127°17'7.6"E, April 28, 2023, isolated from stem of Stephanandra incisa, strain KNUE 23N743, NIBRFGC000510477, GenBank No. OR805471 (ITS), OR826301 (TUB2), OR826293 (CAL) and OR826294 (HIS3).
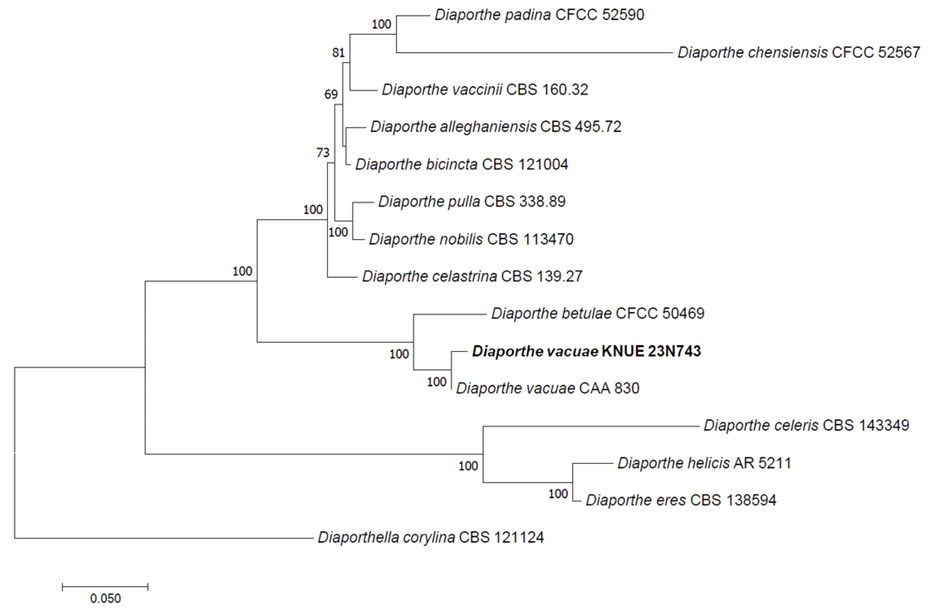
Notes: D. vacuae는 2020년에 Hilário 등에 의해 신종으로 보고되었으며, 진달래과 식물인 Vaccinium corymbosum의 줄기 병변에서 최초로 분리되었다[27]. D. vacuae는 D. eres 및 D. celeris와 계통적으로 가까우나 더 큰 alpha-분생자와 beta-분생자를 가지며, PDA배지에서의 배양 특징이 다르다(Table 5). D. vacuae는 앞면은 흰색에서 회색 균사체가 관찰되고 뒷면은 노란색인 반면 D. eres는 흰색의 솜털같은 균사체가 나타나고 뒷면은 어둡고, D. celeris는 흰색 균사체가 점점 크림에서 갈색으로 변화하며 뒷면은 하얀색 또는 크림색이다[28,29]. D. vacuae KNUE 23N743은 분생자의 크기는 Hilário 등이 기술한 것보다 작게 측정되었으나 PDA배지에서 배양체의 특성이 일치하였다. ITS, TUB2, CAL, HIS3 영역의 DNA 염기서열 분석 결과 ITS는 D. vacuae OM950742와 100%, TUB2는 D. vacuae MT309458과 99.59%, CAL은 D. vacuae MT309449와 100%, HIS3은 D.vacuae MK871446과 99.33% 일치하였으며, neighbor joining 계통수에서 D. vacuae CAA 830과 같은 그룹으로 형성되었다(Fig. 7).
Fig. 7
Neighbor-joining phylogenetic tree of Diaporthe vacuae KNUE 23N743 based on a concatenated alignment of internal transcribed spacer (ITS), β-tubulin (TUB2), calmodulin (CAL) and histone H3 gene (HIS3) sequences. Diaporthella corylina was used as an outgroup. Numbers on branches indicate bootstrap values (1,000 replicates) upper than 50%. Fungal strain isolated in this study is in a bold.
Neurospora tetraspora Dania García, Stchigel & Guarro, Mycological Research 108 (10): 1134 (2004) [MB#488632]
형태적 특징: PDA배지에서 25℃에서 7일간 배양했을 때 균총의 크기는 90 mm 페트리 접시를 모두 채웠으며, 중앙은 연한 회색이며, 가장자리와 중앙 사이에 회갈색의 균사가 발달하고, 고도는 배지에 납작하게 붙어 있다. 배지 뒷면의 중앙은 노란빛을 띄는 회색이며, 가장자리는 짙은 회색이다(Fig. 8A and B). 자낭과는 검은색의 균사로 덮힌 구형이며, 자낭에는 4개의 자낭포자가 발달한다(Fig. 8D and E). 자낭포자는 타원형이며 매끄럽고 검은색이며 크기는 약 (20.84-) 24.08 (-28.78)×(11.70-) 13.64 (-15.40) μm (n=20) 정도이다(Fig. 8C).
Fig. 8
Morphological characteristics of Nuerospora tetraspora KNUE 22N2526. Colonies grown for 7 days on potato dextrose agar (PDA) (A, B), ascospores (C), ascomata (D), asci and ascospores (E).
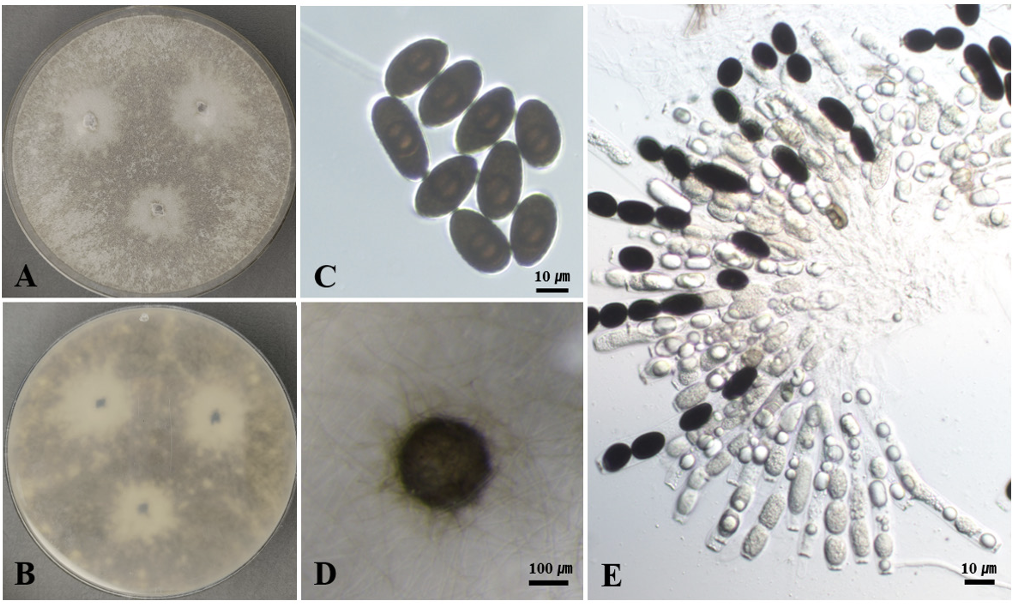
Specimen examined: Uljin-gun, Gyeongsangbuk-do, Korea, 37°02'03.6"N 129°23'27.5"E, July 7, 2022, isolated from leaf of Quercus mongolica, strain KNUE 22N2526, NIBRFGC000510053, GenBank No. OQ271307 (ITS), and OR059183 (LSU).
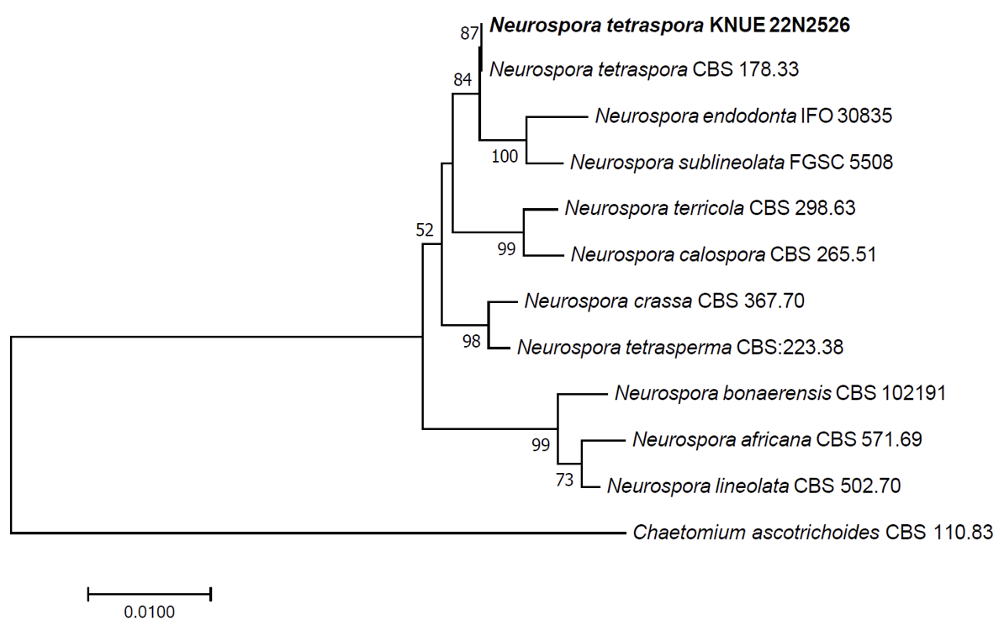
Notes: N. tetraspora는 최초에 Gelasinospora 속으로 분류되었으나 유성생식 구조의 유사성과 28S rDNA 서열 정보를 바탕으로 2004년에 Neurospora 속으로 재분류되었다[30]. 4개의 포자가 든 자낭과 타원형으로 길쭉한 자낭포자를 형성하며, N. tetraspora KNUE 22N2526도 동일한 특징을 나타냈다(Table 6). 주요 분포는 배설물과 토양이며, 재분류된 이후 보고는 2018년 이집트에서 비근권성 토양에서 분리한 것이 있다[30,31]. 따라서 N. tetraspora KNUE 22N2526은 식물 내생균으로 보고된 최초의 균주이다. ITS, LSU 영역의 DNA 염기서열 분석 결과 ITS는 N. tetraspora MH859381과 100%, HIS3은 N. tetraspora NG_068996과 100% 일치하였으며, neighbor joining 계통수에서 N. tetraspora CBS 178.33과 같은 그룹으로 형성되었다(Fig. 9).
Fig. 9
Neighbor-joining phylogenetic tree of Neurospora tetraspora KNUE 22N2526 based on a concatenated alignment of internal transcribed spacer (ITS) and large subunit ribosomal RNA gene (LSU) sequences. Chaetomium ascotrichoides was used as an outgroup. Numbers on branches indicate bootstrap values (1,000 replicates) upper than 50%. Fungal strain isolated in this study is in a bold.