서론
효모는 예로부터 식품을 발효하는데 이용되어 왔으며, 현대에는 식품뿐 아니라 향장소재, 바이오 에너지, 환경개선제 등 다양한 분야에서 활용되며 미생물 산업화에 기여하고 있다. 예를 들어, 대표적인 자낭균문 효모인 Starmerella cerevisiae는 버려지는 감껍질을 발효시켜 바이오에탄올 생산이 가능하게 하고, Candida tropicalis는 바이오필터에 적용 시 인체에 유해한 톨루엔 제거 성능을 증가시키며, S. exiguous는 돈분뇨에 첨가 시 악취를 제거할 뿐 아니라 대장균 증식 억제를 통해 환경개선제로서 이용할 수 있다고 밝혀진 바 있다. 따라서, 야생 효모 발굴은 단순 미생물의 발굴을 넘어 효모의 융복합적 활용을 통해 산업화에 기여할 수 있다[1-3].
포도밭과 포도나무 주변 환경은 효모가 많이 분포하는 장소 중 하나로 알려져 있으며, 전 세계적으로 포도밭 등에서 발견되는 효모는 Rhodotorula graminis, Sporidiobolus pararoseus, Saturnispora divera, Starmerella bacillaris 및 Hanseniaspora uvarum 등과 같이 발효성을 가지거나 산화성을 가지는 효모로 알려져 있다[4].
Cyberlindnera 속은 Phaffomycetaceae 과의 효모이다. 그 이름은 ’음식’과 ’생계’를 의미하는 “Cibus”에서 유래한 라틴어 “Ciber”를 사용하였고, Saccharomyces 및 Pichia 속 발굴에 기여한 독일의 미생물 분류학자 Paul Linder의 이름에서 유래한 “-lindnera”를 사용하였다. 이 속은 다양한 방식으로 다른 유기체와 상호작용하며, 전세계 각지의 여러 환경에서 발견되고 있다[5].
Starmerella 속은 1998년에 Rosa > Lachance에 의해 분류된 효모 속으로, Candida stellata가 재분류 되었다. 이 속은 C. bombicola와 유성생식이 가능한 것으로 알려져있다[6]. Starmerella 속에 속하는 S. basillaris 종은 Saccharomyces cerevisae 종과 함께 발효에 사용될 경우 와인의 향과 구성에 긍정적인 영향을 미친다고 알려져 있다[7].
본 연구에서는 국내 포도밭의 포도나무 밑 흙에서 분리된 야생 효모 중 국내 미기록종을 선별하여 위상차 현미경을 통해 형태학적 특징을 관찰하고, 20C AUX를 실시하여 탄소원 활성을 측정하였다. 미기록종 효모의 정확한 분자생물학적 유연관계를 확인하기 위하여 26S rDNA의 D1/ D2 region을 분석하였다.
재료 및 방법
포도밭 내 야생 효모의 분리 및 동정
2023년 4월 경기도 김포시 소재 Campbell Early 품종의 포도를 재배하는 포도밭(37°41'39.5"N 126°38'55.3"E)에서 토양 샘플을 채집하였다. 멸균된 튜브에 9 mL의 멸균수를 넣고, 포도나무 주변 토양 샘플을 1 g 넣어 희석하였다. 이 현탁액을 연쇄 희석하여 YPD (Yeast extract peptone dextrose, DB Difco, USA) 한천 배지(pH 3.7-5.0)에 100 μL씩 분주하여 도말하였다. 이후 10℃에서 3~5일 배양한 후 효모 집락을 분리하였다[8]. 실험에 사용한 YPD 한천 배지는 chloramphenicol (0.4 g/mL) 0.04%, sodium propionate (0.2 g/mL) 0.02%를 첨가하여 제조하였다.
분리한 효모의 동정을 위해 26S rDNA의 D1/D2 region을 NL1, NL4 primer를 이용하여 polymerase chain reaction (PCR)을 수행하고, 증폭된 산물에 대하여 염기서열 분석을 수행하였다[9]. 확보한 염기서열은 미국 국립생물정보센터(National Center for biotechnology Information, NCBI)의 Basic Local Alignment Search Tool (BLAST) 데이터베이스를 통해 다른 효모와의 상동성을 비교하였고, Clustal X 2.0 프로그램을 이용하여 주변 서열들과 정렬하였다[10]. 계통수는 MEGA 11.0 프로그램에서 neighbor-joining method를 사용하여 구축하였다[11].
포도밭에서 발견된 국내 미기록 효모들의 선별 및 균학적 특성 확인
효모의 위상차 현미경 관찰을 위해, YPD 한천 배지에서 3일간 배양한 후 단일 집락을 분리하여 슬라이드 글라스 위의 멸균 증류수에 희석하고 커버 글라스로 덮어주었다. 위상차 현미경을 통하여 세포의 크기와 출아 여부를 확인하였다.
포도밭에서 발견된 국내 미기록 야생 효모들의 탄소원 활성 측정
효모의 탄소원 활성을 측정하기 위해API 20C AUX kit (bioMerieux, Hazelwood, USA)를 이용하였다. Suspension medium (2 mL)에 YPD 한천 배지에 균을 모아 2McFarland의 탁도에 맞추어 희석하였다. 탁도를 맞춘 균주 혼합물은 API C Medium의 앰플에 100 μL씩 넣어 기포가 생기지 않도록 혼합하였다. 피펫으로 API C medium균액을 스트립의 큐플에 기포가 생기지 않도록 분주하였다. 이를 30℃에서 48-72시간 배양하였다. 음성 대조군과 비교하여 혼탁도가 높은 큐플을 양성 반응으로 결과를 기록하였다[12].
결과
포도밭에서의 야생 효모 분리 및 동정과 미기록종 선별
경기도 김포시의 토양에서 야생 효모 20균주가 분리되었다. 26S rDNA의 D1/D2 region을 분석하여 미국 국립생물정보센터(NCBI, http://ncbi.nlm.nih.gov)의 데이터베이스를 통해 기존에 보고된 종과의 유사도를 확인하였다. 그 결과, 샘플로부터 Moesziomyces 속 4 균주, Solicoccozyma 속 8 균주, Pseudozyma 속 1 균주, Filobasidium 속 2 균주, Rhodosporidiobolus 속 2 균주, Rhodotorula 속 2 균주, Cyberlindnera 속 1 균주 그리고 Starmerella 속 1 균주로 총 10 종, 20 균주가 분리되었다(Table 1). 분리된 20 개의 균주 중 Moesziomyces 속이 4 균주, Solicoccozyma 속이 8 균주로 다른 속 균주들에 비해 상대적으로 많이 분리되었다.
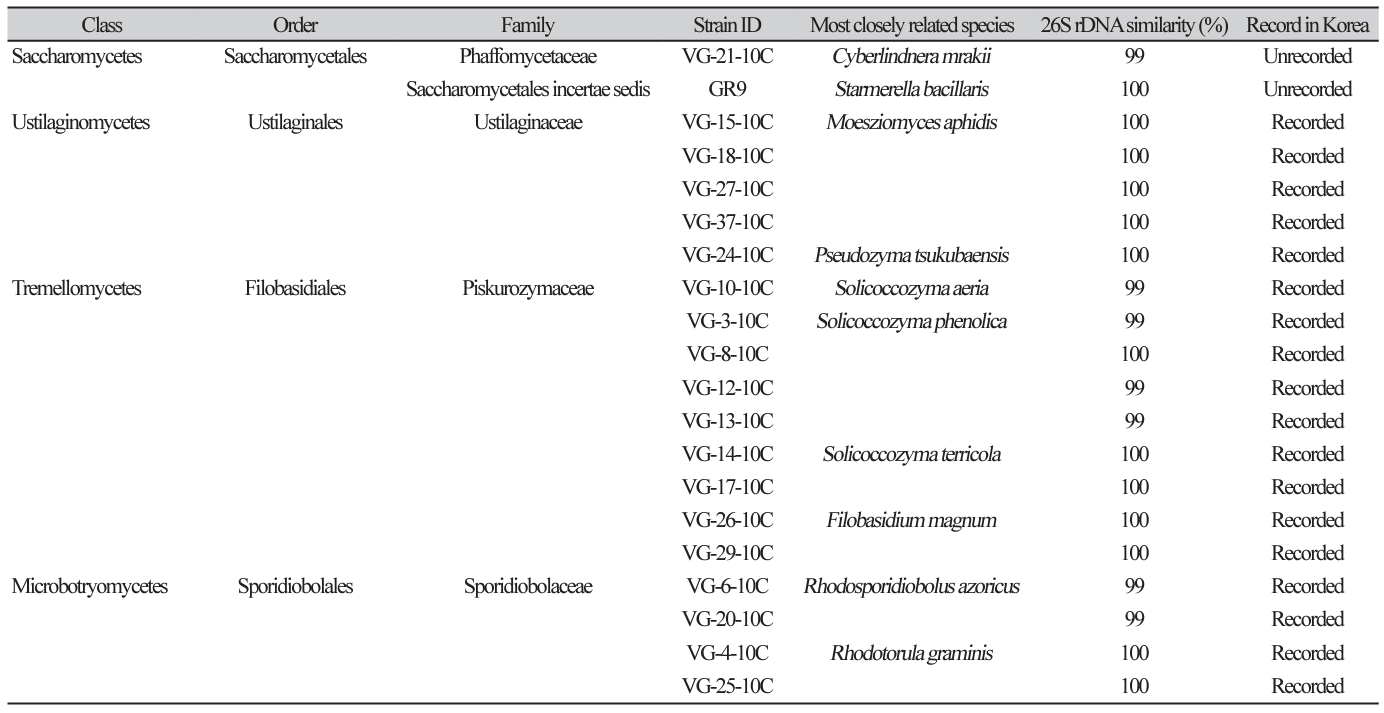
분리한 20 개 균주 중 VG-21-10C 균주는 Cyberlindnera mrakii NRRL Y-1364T와 D1/D2 영역 염기서열이 99.60% 일치하였으며, GR9 균주는 Starmerella bacillaris CBS 843T와 D1/D2 영역 염기서열이 100% 일치하였다.
포도밭에서 분리, 선별한 국내 미기록 야생 효모의 분자계통학적 위치
국내 미기록종으로 선별된 Cyberlindnera mrakii VG-21-10C 균주와 Starmerella bacillaris GR9 균주의 D1/D2 영역을 기준으로 시퀀스 유사도의 결과는 다음과 같았다. VG-21-10C 균주는 C. mrakii NRRL Y-1364T 균주와 염기서열 유사도가 99.6%이었고, 균주 GR9은 S. bacillaris CBS 843T 균주와 염기서열이 100% 일치하였다. 이를 통해 균주 VG-21-10C는 C. mrakii와 균주 GR9는 S. bacillaris와 매우 유사한 것으로 확인되었다. Neighbor-joining 기법을 이용하여 구성한 계통수는 각각 Fig. 1 그리고 Fig. 2와 같다. C. mrakii VG-21-10C 균주와 S. bacillaris GR9에서 획득한 D1/D2 영역 염기서열을 Genbank에 등록하였으며, 각각의 accession 번호는 OR826467과 OR827441이다.
Fig. 1
A neighbor-joining phylogenetic tree based on the nucleotide sequences of D1/D2 region of 26S rDNA showing the position of Cyberlindnera mrakii VG-21-10C and other species of the genera Cyberlindnera and Candida. The tree was generated by the neighbor-joining method, using MEGA11. Bootstrap values (>70%) based on the neighbor-joining method were shown at the branch nodes. Bar, 0.02 substitutions per nucleotide position. Wickerhamomyces pijperi NRRL_YB-4309T was used as the outgroup. T , type strain.
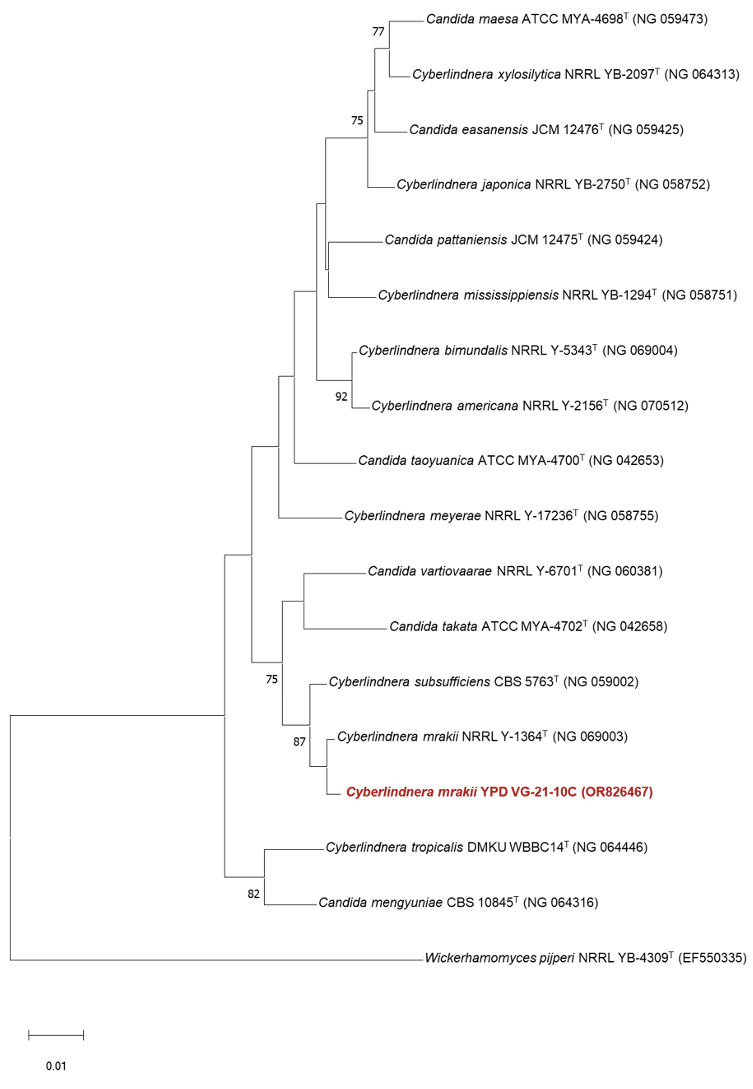
Fig. 2
A neighbor-joining phylogenetic tree based on the nucleotide sequences of D1/D2 region of 26S rDNA showing the position of Starmerella bacillaris GR9 and other species of the genus Starmerella. The tree was generated by the neighbor-joining method, using MEGA11. Bootstrap values (>70%) based on the neighbor-joining method were shown at the branch nodes. Bar, 0.02 substitutions per nucleotide position. Alloascoidea africana CBS 377.68T was used as the outgroup. Ttype strain.
포도밭에서 분리, 선별한 국내 미기록 야생 효모들의 균학적 특성
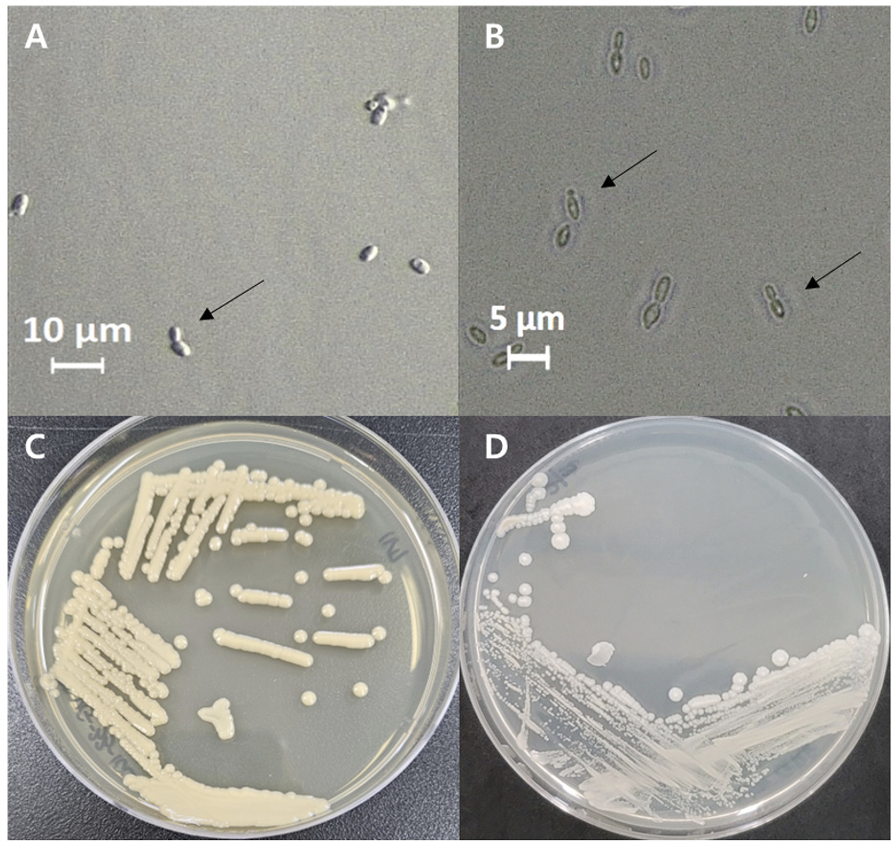
국내 미기록종으로 최종 선별된 Cyberlindnera mrakii VG-21-10C, Starmerella bacillaris GR9 균주의 균학적 특성은 Table 2와 Table 3에 나타냈다. 이들 미기록 효모들의 형태적, 배양적 특징으로는 두 균주 모두 세포의 형태가 난형이고, 출아로 영양증식하였다. 또한, 세포의 크기는 각각 균주 VG-21-10C는 5.3-5.6 μm와 3.1-3.4 μm, 균주 GR9은 2.2-2.5 μm와 0.8-1.0 μm이었다(Fig. 3). VG-2110C 균주는 최근연종인 C. mrakii NRRL Y-1364T 균주의 형태 및 배양적 특징을 비교한 결과, 형태학적으로 동일한 특징을 보여주었다[13]. GR9 균주는 S. bacillaris CBS 843T 균주의 형태 및 배양적 특징을 비교한 결과, 형태학적으로 동일한 특징을 보여주었다[14]. 동정된 VG-21-10C와 GR9 균주는 국립농업과학원 농업미생물은행에 각각 KACC 410281과 KACC 410282로 기탁하였다.
Table 2
Characteristics of unrecorded wild yeast Cyberlindnera mrakii VG-21-10C and closely related species.
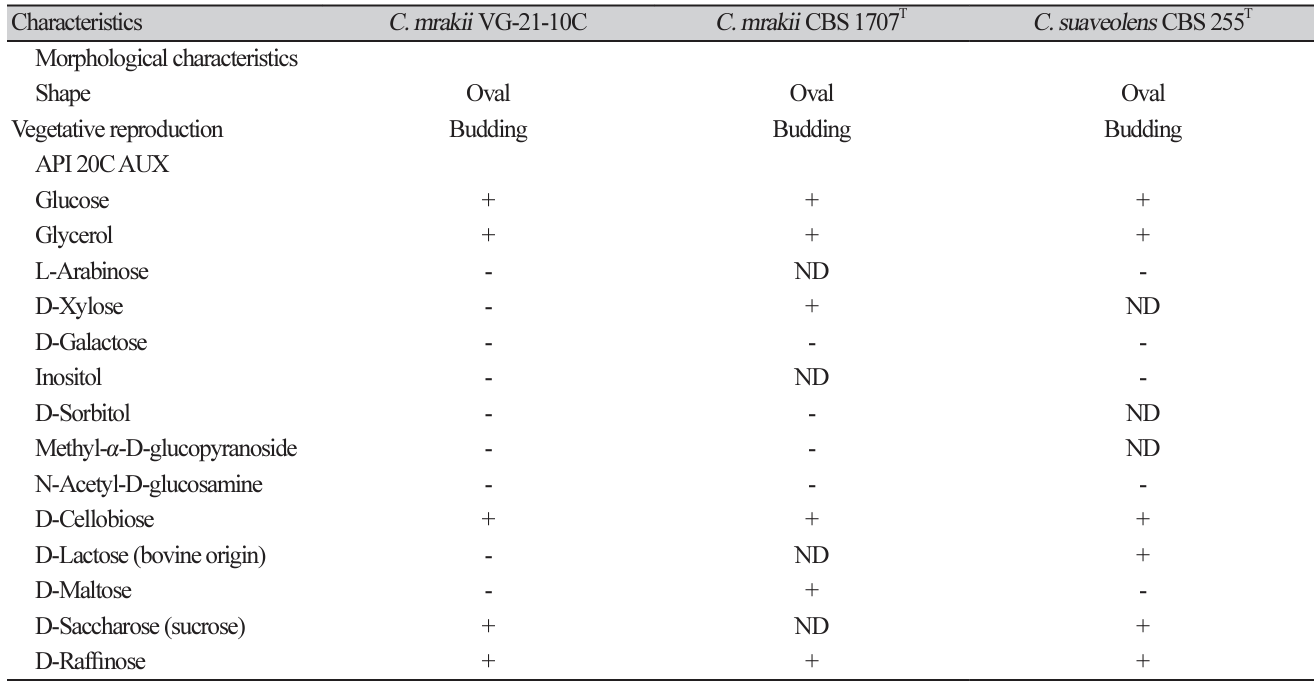
Table 3
Characteristics of unrecorded wild yeast Starmerella bacillaris GR9 and closely related species.

Fig. 2
Microscopic features of the unrecorded yeasts isolated from vineyard soil in Gimpo-si, Gyeonggido, Korea. The budding cells of Starmerella bacillaris GR9 (A) and Cyberlindnera mrakii VG-21-10C (B). bars, 10 µm and 5 µm, respectively. The colonies of Starmerella bacillaris GR9 (C) Cyberlindnera mrakii VG-21-10C (D).
국내 미기록 야생 효모들의 탄소원 활성 측정
Cyberlindnera mrakii VG-21-10C 균주의 탄소원 활성 측정 결과는 다음과 같았다. Glucose, glycerol, D-xylose, D-cellobiose, D-saccharose 그리고 D-raffinose는 양성, calcium 2-keto-gluconate, L-arabinose, anoitol, xylitol, D-galactose, inositol, D-sorbitol, methyl-α-D-glucopyranoside, N-acetylglucosamine, D-lactose, D-maltose, D-trehalose 그리고 D-melezitose는 음성이었다. VG-21-10C 균주는 최근연종인 C. mrakii CBS 1707T와 D-lactose (bovine origin) assimilation 실험 결과에서 차이를 보였다[13]. Starmerella bacillaris GR9 균주의 탄소원 활성 측정 결과는 다음과 같았다. Glucose, D-saccharose 그리고 D-raffinose는 양성, glycerol, calcium 2-keto-gluconate, L-arabinose, D-xylose, adonitol, xylitol, D-galactose, inositol, D-sorbitol, methyl-α-D-glucopyranoside, N-acetyl-glucosamine, D-cellobiose, D-maltose, D-trehalose 그리고 D-melezitose는 음성이었다. GR9 균주는 최근연종인 S. bacillaris CBS 844T 균주와 D-Lactose (bovine origin) assimilation 실험 결과에서 차이를 보였다[14].
고찰
국내 포도밭의 포도나무 주변 흙에서 야생 효모를 분리, 동정하였다. 토양 샘플에서 총 20개의 야생 효모가 분리되었다. 26S rRNA 유전자의 D1/D2 영역 서열을 분석한 결과 국내에서 보고되지 않은 효모 2 균주를 선별하였다. 국내 미기록종 효모인 Cyberlindnera mrakii VG-21-10C와 Starmerella bacillaris GR9 균주는 위상차 현미경과 API 20C AUX kit를 통해 표현형적 특성을 조사하였다. VG-21-10C 균주와 GR9 균주의 세포 모양은 타원형이며, 집락은 볼록하고, 부드러운 질감을 가지는 것으로 관찰되었다. 탄소원 활성 측정 결과, VG-21-10C 균주는 glucose, glycerol, D-xylose, D-cellobiose, D-saccharose, D-raffinose을 탄소원으로 사용할 수 있었고, GR9 균주는 glucose, D-saccharose, D-raffinose을 탄소원으로 사용할 수 있음을 확인하였다.
균주 VG-21-10C는 C. mrakii CBS 1707T 균주와 유전적으로 매우 유사한 특성이 확인되었다. 두 균주는 D-lactose (bovine origin) assimilation 실험 결과에서 차이를 보였지만 집락이 볼록하고, 부드러운 형태를 가졌으며 위상차 현미경 하에서 관찰한 형태적 특징도 일치하였다[13]. 계통수 상에서도 균주 VG-21-10C는 C. mrakii CBS 1707T 균주와 동일한 종임을 알 수 있었다. C. mrakii 종은 지금까지 국내에서 보고된 바 없으므로, 균주 VG-21-10C는 미기록종으로 판단된다(Table 1).
균주 GR9는 유전적으로 매우 유사한 S. bacillaris CBS 844T 균주와 유사한 특성이 확인되었다. 하지만 S. bacillaris CBS 844T 균주와 D-lactose (bovine origin) assimilation 실험 결과에서 차이를 보였지만 집락이 볼록하고 부드러운 형태를 가지며 위상차 현미경 하에서 관찰한 형태적 특징이 일치하였다[14]. 계통수 상에서도 S. bacillaris CBS 844T 균주와 동일한 종임을 알 수 있다. S. bacillaris 종은 아직까지 국내에서 보고된 바 없는 미기록종이므로 균주 GR9는 국내 미기록종으로 판단된다.