우리나라 산림에서 가장 넓은 면적에 분포하고 있는 소나무(Pinus densiflora Siebold & Zucc.)는 침엽수를 대표하는 수종으로서 총 산림 면적의 23.5%인 150만 ha를 차지하며 전국에 걸쳐 분포하고 있어 산림생태계를 유지하는 주요 수종이다[1,2]. 소나무는 척박한 토양에서도 잘 생장하고 생태 적응범위도 매우 넓어, 오랜 세월 동안 목재, 연료, 식용 등 다양한 방면으로 활용되어 왔다[2,3]. 내생균류는 오랜 기간동안 숙주식물과 특이적으로 친화적인 관계를 형성하여 상호보완적인 작용을 해 왔다[4]. 일반적으로 내생균류는 식물의 분포상에 따라 다르게 나타나며, 한 종의 기주 식물에서 다양한 종류의 내생균이 분리될 수 있다[5,6]. 내생균은 기주에 따라 지역, 환경, 계절, 부위별로 다양성이 다르게 나타나며[7], 백 만종 이상이 존재하는 것으로 추정하고 있다[8]. 산림에 존재하는 내생균은 숙주식물 내에서 2차대사산물을 분비하여 식물 생장을 촉진시키고, 비생물적 환경스트레스에 대한 저항성을 증가시킨다[9]. 내생균은 외부에서 침입하는 선충, 식물 병원균 등 생장을 저해하는 요소들로부터 숙주 식물을 보호하는 역할을 한다[10]. 내생균과 숙주 식물 사이의 특이적인 관계에 대해서는 아직까지 명확하게 밝혀지지는 않았으나[5], 건강한 식물의 경우 병징 발현을 저해하여 식물을 보호하고 식물의 종자 발아에 도움을 주며[11], 병원체에 대하여 기주 식물이 저항성을 갖게 하기도 한다[12]. 최근 목본식물의 잎 조직에서 공생하는 내생균이 다른 병원미생물에 대하여 항생 효과를 나타낸다는 연구들이 보고되면서[13,14], 식물 종 자체에 대한 연구뿐만 아니라 식물 내에 존재하는 내생균에 대한 연구의 필요성이 제기된 바 있다[4]. 국외 연구의 경우 Hate 등은 침엽수에 존재하는 내생균의 분포와 계절별 다양성을 연구하였고, Hoff 등은 침엽수의 뿌리에 공생하는 내생균을 분리하여 다양성을 보고하였으며, Wang 등은 만주 곰솔의 잎에서 다양한 내생균을 분리하는 연구를 보고하였다[15-17]. Wang 등은 Pinus tabulaeformis의 수피에서 분리한 내생균의 다양성을 분석하는 연구를 보고하였으며, Sousa 등은 내생균 Diaporthe sp.의 항균 활성에 대한 연구를 보고하는 등 전 세계적으로 침엽수에서 분리한 내생균의 다양성 연구와, 분리 내생균의 활용 가능성에 대한 연구가 활발하게 진행되고 있다[18,19]. 국내의 경우 Park 등은 전나무(Abies holophylla)에서 분리한 내생균류에서 추출한 물질이 식물병원균에 미치는 영향을 연구하였으며, Seo 등은 한국의 잣나무(Pinus koraiensis)에서 113 균주의 내생균을 분리하였고, Kim 등은 민주지산에 서식하는 침엽수의 잎에서 분리한 내생균 39균주를 22종으로 보고하였다[20-23].
국내에서도 내생균의 다양성 연구는 활발하게 이루어지고 있으나 주요 수종인 소나무 내생균의 다양성 관련 연구는 보은 지역의 소나무에서 분리한 내생균의 동정 및 다양성 연구와[24], 충남 오서산에 분포하는 소나무와 일본 잎갈나무에서 분리한 내생균의 다양성에 대한 연구[25] 등 매우 제한적이다. 이처럼 국내에서는 잣나무, 소나무의 일부 부위에서 분리한 내생균의 다양성 연구에 국한되어 있으며 소나무 내생균의 부위별, 시기별 다양성 분석에 대한 연구는 현재까지 보고되지 않은 실정이다. 이에 본 연구에서는 다양한 균주의 확보와 균주의 다양성을 확인하여 내생균을 활용할 수 있는 방안의 기초자료를 제공하고자 국내 우점 수종인 소나무에서 내생균을 분리하여 부위별, 시기별 다양성을 파악하고자 하였다.
2017년 가을과 2018년 봄에 강원도 춘천시에 위치하고 있는 강원대학교 구내림(수정산, 37.869444 N, 127.744472 E)과 애막골 야산(37.867364 N, 127.761542 E)에 분포하는 소나무(Pinus densiflora) 8본으로부터 침엽, 가지, 줄기를 채집하였다. 채집한 소나무의 침엽, 줄기, 가지시료는 병해나 해충 피해가 없는 건전 시료를 사용하였으며 침엽은 4×4 mm 크기로, 가지와 줄기는 2 ×8 mm의 크기로 절단하였다. 채집한 시료는 24시간 이내에 표면살균을 실시하였고 시료 각 부위별로 9개 조각을 0.5% 차아염소산나트륨(sodium hypochlorite)과 0.05% Triton X-100 용액에 3분씩, 70% 에탄올에서 1분간 표면살균한 후 멸균수(sterilized water)에 15초간 3회 반복하여 세척하였다. 표면살균한 시료는 건조시킨 뒤 streptomycin과 chloramphenicol을 각각 0.1 g씩 첨가한 potato dextrose agar (PDA, Difco, Franklin Lakes, USA) 평판 배지에 각각 3개의 절편을 치상하여 25℃ 항온기에서 2주 간 암배양하고 생장해 나온 균사를 새로운 PDA배지에 이식하여 순수분리하였다[26].
순수분리한 내생균을 potato dextrose broth (PDB, Difco, Franklin Lakes, USA) 배지에 접종하고 25℃에서 150 rpm으로 1-3주간 진탕 배양하여 균사를 수확하고 동결건조 후 마쇄하여 DNeasy Plant Mini Kit (QIAGEN DNA extraction kit, QIAGEN, Hilden, Germany)의 공시 방법에 따라 DNA를 추출하였다. 내생균을 동정하기 위하여 모든 내생균을 대상으로 rDNA의 internal transcribed spacer (ITS) 영역을 분석하였고, 필요한 경우 rDNA의 large subunit (LSU), RNA polymerase II gene (RPB2), elongation factor-1α (TEF) 또는 beta-tubulin (TUB) 영역도 추가로 분석하였다. Ribosomal DNA의 ITS영역 증폭을 위해 ITS1과 ITS4 프라이머를 사용하였고 LSU는 LROR과 LR7 프라이머를 RPB2는 fRPB2-5F와 fRPB2-7cR 프라이머를 TEF는 EF1-728F과 EF1-986R 프라이머를 TUB는 bt2a와 bt2b 프라이머를 사용하였다[27-29]. 모든 영역의 annealing 온도는 58℃로 설정하여 PCR을 수행하였고 증폭산물은 Macrogen Inc. (https://dna.macrogen.com)에 의뢰하여 염기서열을 분석하였다. 염기서열 편집에는 SeqMan Program version 7.2.1 (DNA Star Lasergene 7)을 이용하였고 편집한 염기서열은 National Center for Biotechnology Information (NCBI)의 Basic Local Alignment Search Tool (BLAST)를 사용하여 가장 유사한 염기서열 종을 확인하였다. 동정결과는 Shannon-Wiener index를 이용하여 다양도 지수를 산출하였다[30]. 계통수 분석에는 MEGA 11 프로그램[31]을 이용하여 Neighbor-Joining 방식으로 계통도를 작성하였고, bootstrap replications은 1,000회로 반복하여 신뢰도를 분석하였으며 Model은 Tamura-Nei model로 설정하였다.
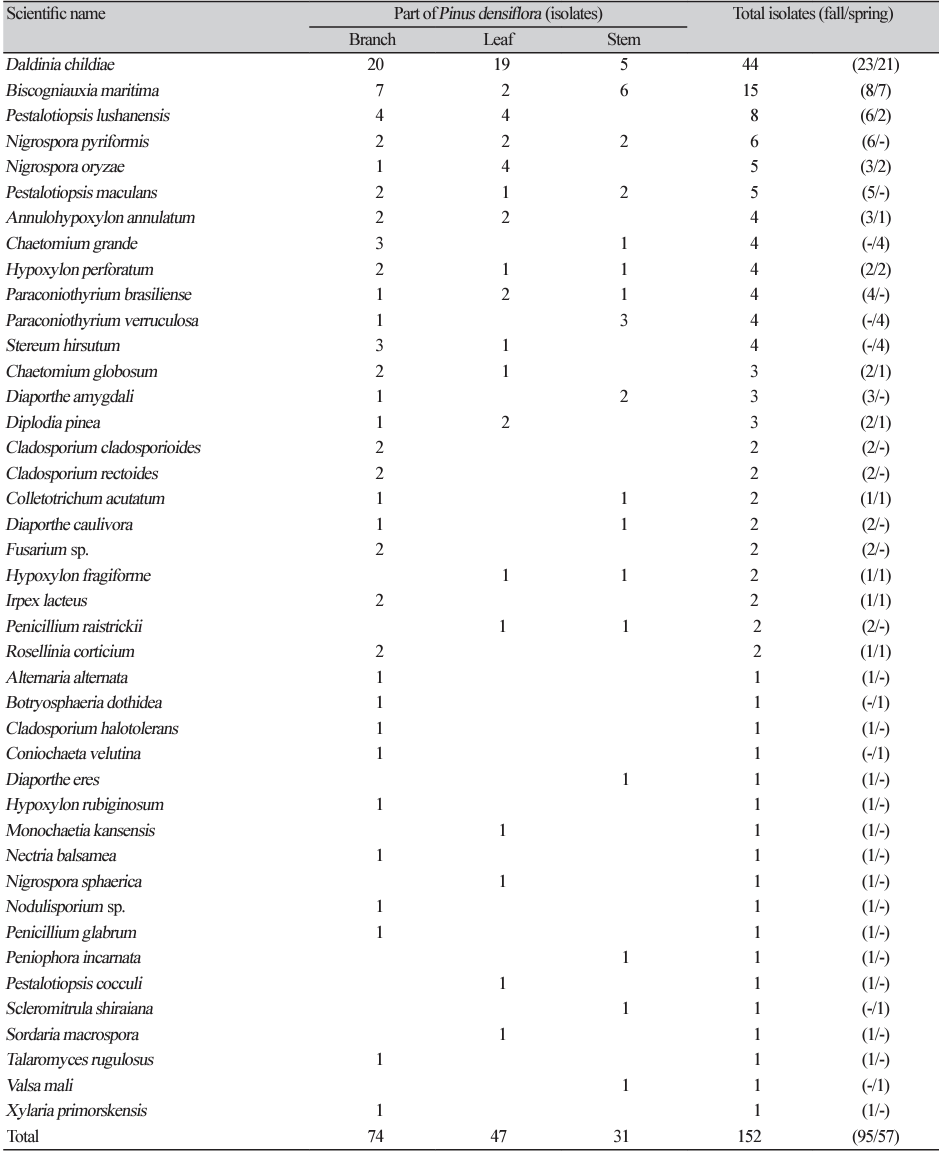
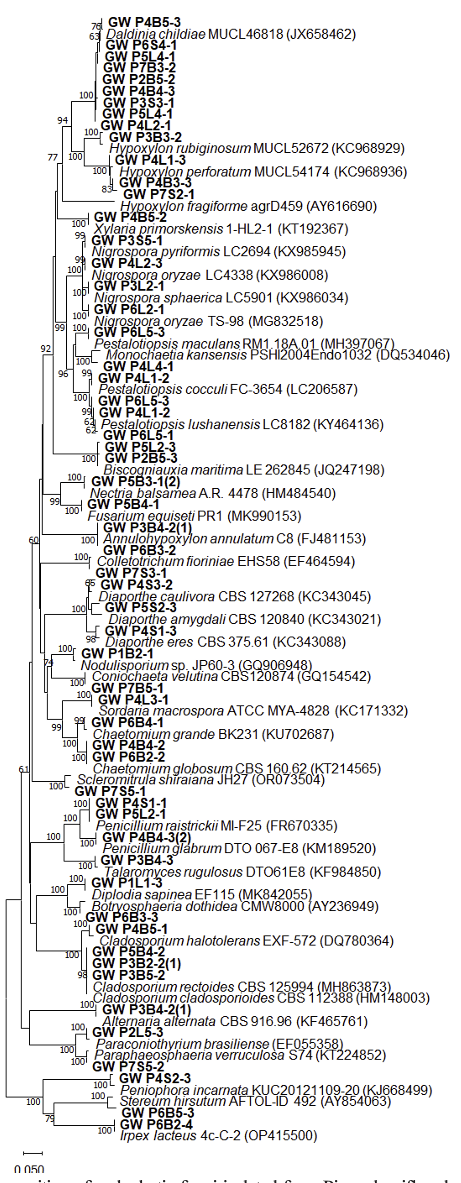
소나무에서 분리한 152 균주의 내생균은 PDA배지에서의 배양적 특징을 확인하여 동일 종으로 생각되는 균주들은 대표균주를 선발하여 분자적 특징을 조사하였다. 선발된 대표균주들은 대부분 rDNA-ITS영역의 분석을 통해 동정하였고(Fig. 1), rDNA-ITS영역만으로 동정이 어려운 종들은 rDNA-LSU (Scleromitrula shiraiana), RPB2 (Alternaria alternata), TEF (Cladosporium rectoides, Diaporthe spp., Diplodia sapinea, Fusarium sp.) 또는 TUB (Rosellinia corticium, Diaporthe spp.) 영역을 분석하여 동정하였다. 동정결과 분리균들은 2문 5강 13목 22과 29속 42종으로 동정하였다(Table 1). 대부분의(145/152균주, 95.4%) 균주는 자낭균문으로 동정되었고, 7균주는 담자균문(1강 2목 3과 3속 3종)으로 동정되었다. 자낭균문에 속한 균주들은 대부분 Sordariomycetes (122/145, 85.1%)강 Xylariales (콩꼬투리버섯목, 101/145 69.7%) Hypoxylaceae (55/145, 37.9%)에 속하는 것으로 나타났다. Daldinia속이 44균주(28.9%)로 가장 높은 비율로 분리되었으며 Biscogniauxia속이 15균주(9.8%), Pestalotiopsis속이 14균주(9.2%), Nigrospora속이 12균주(7.9%) 분리되었다. 가장 많이 분리된 우점종은 44균주(28.9%)가 분리된 Daldinia childiae, 다음은 15균주(9.8%)가 분리된 Biscogniauxia maritime이었다. Gil 등과 Eo 등의 소나무(Pinus densiflora)에서 분리한 내생균의 다양성 연구에서 Lophodermium속이 우점한다는 결과와 본 연구의 결과와는 다르게 나타났다[24,25]. 이는 충청도와 강원도라는 지리적 차이에 의한 것으로 추측된다.
Fig. 1
Phylogenetic position of endophytic fungi isolated from Pinus densiflora based on rDNA-ITS. Sequences of the isolates were compared with those of closely related species from NCBI Genbank site. The combined data were analyzed using Tamura-Nei parameter distance calculation model, and then used to construct the Neighbor-Joining (NJ) tree with MEGA ver. 11 [31]. Bootstrap analysis was performed with 1,000 replications. Strains isolated in this study are indicated in bold text.
본 연구에서 우점종인 Daldinia childiae는 주로 식물의 잎, 줄기, 뿌리에서 분리되는 내생균으로 항균, 항산화 작용을 하는 균으로 보고된 바 있다[32,33]. 국내에서도 인삼 잎에서 분리된 내생균의 다양성과 탄저병 내성 연구[34], 침엽수의 잎에 공생하는 내생균의 다양성과 계절적 변이 연구[22], 한국 자생 난초과 식물의 뿌리에서 분리한 내생균의 다양성 연구[35] 등에서도 D. childiae가 내생균으로 보고되었다. 또한 Oh 등의 지의류 내생균 다양성 조사에서도 D. childiae가 가장 많이 분리된 종으로 보고되었으며 지의류가 척박한 환경에도 잘 생장할 수 있는 이유 중 하나로 추측하였다[36]. 소나무도 지의류와 유사하게 척박한 환경에서도 잘 자라는 특징을 가지고 있는데 이는 D. childiae와 같은 내생균들에 의한 결과로 예상된다[37]. 이처럼 목본류를 포함한 다양한 식물뿐만 아니라 지의류에서도 내생균으로 자주 분리되는 D. childiae는 활용 가능성이 높은 것으로 보이므로 향후 추가적인 연구의 필요성이 있다.
소나무 부위별 내생균 분리 결과, 가지에서 74균주(49%), 잎에서 47균주(31%), 줄기에서 31균주(20%)가 분리되었다. 가지와 잎에서는 Daldinia속이 각각 20균주와 19균주로 분리빈도가 높았고, 가지에서는 Biscogniauxia속(7균주), Pestalotiopsis속(6균주)이, 잎에서는 Nigrospora속(7균주), Pestalotiopsis속(6균주)이 높은 빈도로 분리되었다. 줄기에서는 Biscogniauxia속이 6균주로 분리빈도가 가장 높았으며, Daldinia속이 5균주, Paraconiothyrium속이 4균주 분리되었다. 소나무 부위별 우점종은 가지와 잎에서는 D. childiae (각각 20균주, 19균주)이었고 가지에서는 Biscogniauxia maritima (7균주)도 높은 빈도로 분리되었다. 잎에서는 B. maritima가 6균주, D. childiae가 5균주 분리되었다.
소나무의 잎, 가지, 줄기 등 모든 부위에서 공통적으로 분리된 내생균은 D. childiae, B. maritima 뿐만 아니라 Hypoxylon perforatum, Nigrospora pyriformis, Paraconiothyrium brasiliense, Pestalotiopsis maculans이었다. 반면, Botryosphaeria dothidea, Diaporthe eres, Peniophora incarnata, Scleromitrula shiraiana, Valsa mali는 줄기에서만 분리되었고, Monochaetia kansensis, Pestalotiopsis cocculi, Sordaria macrospora는 잎에서만 분리되었다. Fisher 등은 식물의 부위에 따라 내생균의 군집과 다양성이 다르게 나타난다고 보고하였는데[38], 본 연구의 소나무 부위별 분리된 내생균의 동정결과 잎과 줄기는 비교적 다른 내생균 종들이 분리되었다. 가지의 경우 일부 잎과 줄기 두 부위에서 분리된 내생균 종들이 분리되었는데 이는 가지는 잎과 줄기 사이에 위치하고 있기 때문이라고 추측할 수 있지만 본 실험에 사용된 소나무 시료가 다양하지 않아 보다 자세한 결과를 얻기 위하여 보다 많은 시료를 이용한 다양성 분석이 필요한 것으로 생각된다.
채집시기에 따른 소나무 내생균의 다양성 분석 결과, 봄철에는 57균주가 분리되어 17속 19종으로 동정되었고 가을철에는 95균주가 분리되어 24속 35종으로 동정되었다. 분리빈도가 높은 종은 D. childiae로 봄철과 가을철 모두 공통적(각각 21균주, 23균주)으로 가장 많이 분리되었고 Biscogniauxia maritima (7균주, 8균주)도 높은 빈도로 분리되었다. 봄철과 가을철에 모두 가장 많이 분리된 2종은 본 연구에서 분리된 모든 분리균 중에서도 우점종이며 계절에 상관없이 우점하는 것으로 나타났다. 가을철에는 Nigrospora pyriformis (6균주), Pestalotiopsis lushanensis (6균주), Pestalotiopsis maculans (5균주)도 높은 빈도로 분리되었는데 P. lushanensis만 봄철과 가을철 모두 분리되었고 다른 2종은 가을철에만 분리되었다. 선행연구에 의하면 내생균의 분리빈도는 계절적인 영향을 받아 계절이 경과될수록 종 다양성과 종 풍부도가 증가하여 내생균의 다양성에 영향을 미친다고 보고하였다[15]. 봄에서 여름, 여름에서 가을, 가을에서 겨울로 시간이 경과될수록 주변 환경과 외부요인에 의해 성숙되는 내생균으로 인하여 다양성 또한 높게 나타난다고 할 수 있다[39]. 본 연구에서도 봄과 가을에 채취한 시료에서 내생균을 분리한 결과, 봄철에 비하여 가을철에 보다 다양한 내생균이 분리되었다. 또한 Shannon-Wiener 다양성 지수 분석결과에서도 봄철은 2.318 가을철은 3.029로 가을철이 보다 높은 다양성 지수를 보였다.
적요
우리나라 산림에 가장 많이 서식하고 있는 수종인 소나무(Pinus densiflora)의 내생균 다양성을 분석할 목적으로 강원도 춘천에 서식하고 있는 소나무 시료를 봄철과 가을철에 침엽, 가지, 줄기 등 부위별로 구분하여 채집하였다. 채집 시료에서 내생균 152 균주를 분리하여 29속 42종으로 동정하였다. Daldinia childiae가 44균주(28.9%)로 가장 많이 분리된 우점종이었고, Biscogniauxia maritima가 15균주(9.8%)로 분리빈도가 높았다. 부위별로는 가지에서 74균주(49%), 잎에서 47균주(31%), 줄기에서 31균주(20%)로 가지에서 가장 많은 내생균이 분리되었다. 부위별 우점종은 가지와 잎에서는 D. childiae (각각 20균주, 19균주)이었고 가지에서는 B. maritima (7균주)도 자주 분리가 되었다. 잎에서는 B. maritima가 6균주, D. childiae가 5균주로 자주 분리되었다. 채집시기에 따른 소나무 내생균의 다양성은 봄철에는 57균주가 분리되어 17속 19종으로 동정되었고 가을철에는 95균주가 분리되어 24속 35종으로 동정되었다. 자주 분리된 종으로는 봄철과 가을철 모두 공통적으로 D. childiae (각각 21균주, 23균주)의 분리빈도가 가장 높았고 Biscogniauxia maritima (7균주, 8균주)도 자주 분리되었다.